elga-cdalab-2.06.2:Spezifikation der Befunddarstellung Level 2: Unterschied zwischen den Versionen
| [unmarkierte Version] | [unmarkierte Version] |
(→Empfehlungen für die Darstellung der Allergiediagnostik) |
(→Empfehlungen für die Darstellung der Allergiediagnostik) |
||
| Zeile 304: | Zeile 304: | ||
<tr ID="OBS-1-1"> | <tr ID="OBS-1-1"> | ||
<td>sx1 Inhalatives Screening<br/> | <td>sx1 Inhalatives Screening<br/> | ||
| − | <content styleCode="italics">Lieschgras, Roggen, Birke, Beifuß, Dermatophagoides pteronyssinus, Katzenschuppen, Hundeschuppen, Cladosporium herbarum</content> | + | <content styleCode="italics">Lieschgras, Roggen, Birke, Beifuß, Dermatophagoides pteronyssinus, |
| + | Katzenschuppen, Hundeschuppen, Cladosporium herbarum</content> | ||
</td> | </td> | ||
<td>negativ</td> | <td>negativ</td> | ||
| Zeile 311: | Zeile 312: | ||
<tr ID="OBS-1-2" styleCode="xELGA_red"> | <tr ID="OBS-1-2" styleCode="xELGA_red"> | ||
<td>mx1 Schimmelpilzemix 1<br/> | <td>mx1 Schimmelpilzemix 1<br/> | ||
| − | <content styleCode="italics">Alternaria alternata, Aspergillus fumigatus, Cladosporium herbarum, Penicillium notatum</content></td> | + | <content styleCode="italics">Alternaria alternata, Aspergillus fumigatus, Cladosporium herbarum, |
| + | Penicillium notatum</content></td> | ||
<td>positiv</td> | <td>positiv</td> | ||
<td>A</td> | <td>A</td> | ||
Version vom 7. August 2017, 13:02 Uhr
Inhaltsverzeichnis
1 Spezifikation der Befunddarstellung Level 2
1.1 Überblick
Die nachfolgende Tabelle gibt einen Überblick über die im lesbaren Text anzugebenden und in Level 2 (unter component/section) zu anzugebenden medizinischen Inhalte. Die Optionalität bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für Level 3 (z.B. ist die „Bemerkung Labor“ in dieser Übersicht mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es nur werden, wenn tatsächlich eine Bemerkung vorhanden ist).
Die Reihenfolge der Elemente innerhalb einer Gruppe ist zu beachten.
Die Angabe der Sektion Brieftext ist im Laborbefund ERLAUBT (Siehe Allgem. Leitfaden [4] TemplateID: 1.2.40.0.34.11.1.2.1).
| Feld | Opt | Darstellung | Details | |
|---|---|---|---|---|
| Befundbereiche | <section/title>Name des Befundbereichs</section/title>
Der Name des Befundbereichs wird in <section/title> codiert und nicht innerhalb des <section/text> Elements | |||
| <Component/section/text> Inhalt | ||||
| Allgemeine Befundinformationen | O | |||
| 1 | Auftragsdiagnose (Zuweiser-diagnose) | O [0..*] |
<paragraph> </paragraph> |
|
| 2 | Fragestellung | O [0..1] |
<paragraph> </paragraph> |
|
| 3 | Befundtext | O | |
|
| Spezimeninformation | < table> pro Spezimen eine Zeile < tr> </ tr>
Sollte ein Befund aus mehreren Sections bestehen, wird die Spezimeninformation ausschließlich in einer eigenen Section angegeben und als erste Section geführt.Generell gilt, dass die Angabe von Informationen zu Proben/Spezimen/Material vorgeschrieben ist.
| |||
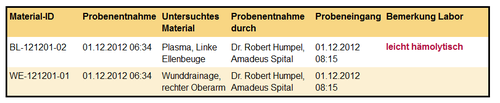
| 1 | Material-ID | O | <td></td> | Identifikator der Probe |
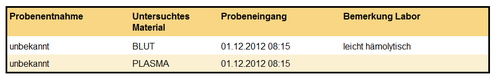
| 2 | Probenentnahme | R | <td></td> | Zeitpunkt der Probebentnahme, muss nicht angegeben werden bzw darf „unbekannt“ sein. Format: dd.MM.yyyy hh24:mi (4.4.5.3.3.4) |
| 3 | Untersuchtes Material | R | <td></td> | Materialart [R] (4.4.5.3.3.7) und Entnahmeort [O] (4.4.5.3.3.5) (Freitext ist zulässig) |
| 4 | Probenentnahme durch | O | <td></td> | Für Probenentnahme zuständige Person und ggf Organisation [O] (4.4.5.3.3.6) |
| 5 | Probeneingang | R | <td></td> | Probeneingang im Labor, Format: dd.MM.yyyy hh24:mi |
| 6 | Bemerkung Labor | R | <td></td> | Allfällige Bemerkungen zur Probenqualität sollen angegeben werden |
| Befundgruppen | <paragraph styleCode="xELGA_h3"> Name der Gruppe</paragraph> | |||
| Gruppierung / Befundgruppen (Organizer) | ||||
| Ergebnistabelle (Observations)4 | < table> je Test eine Zeile <tr></tr> | |||
| 1 | Analyse | M | <td></td> | Bezeichnung der Analyse (entsprechen dem displayName in Value Set ELGA-Laborparameter) |
| 2 | Ergebnis | M | <td></td> | Ergebnis der Analyse5, siehe 4.3.5.2 |
| 3 | Einheit | M | <td></td> | Einheit (UCUM printName), siehe 4.3.5.3 |
| 4 | R2 | <td></td> | Mehrere Referenzbereiche können angegeben werden, getrennt durch Zeilenumbruch im Text6 | |
| 5 | Interpretation | R2 | <td></td> | Codiert! Siehe 4.3.5.4 sowie Kapitel Tabelle 7 und Tabelle 8 |
| 6 | Externes Labor | R2 | <td></td> | Angabe von „E“, wenn die Analyse von einem externen Dienstleister gemessen wurde |
| Eigenschaften des Materials / Mikroskopie | < table> pro Eigenschaft eine Zeile <tr></tr> | |||
| 1 | Eigenschaft | M [1..1] |
<td></td> | |
| 2 | Ergebnis | M [1..1] |
<td></td> | |
| 3 | Einheit | O [0..1] |
<td></td> | |
| Kultureller Erregernachweis | < table> pro Erreger eine Zeile<tr></tr> | |||
| 1 | Erreger | M [1..1] |
<td></td> | |
| 2 | Methode | R2 [1..1] |
<td></td> | Mögliche Werte vgl. Tabelle 12: Beispiele für Codes für Erregernachweis-Methodik |
| 3 | Keimzahl | M [1..1] |
<td></td> | |
| Antibiogramm | < table> je Antibiotikum Zeile <tr></tr> | |||
| 1 | Name des Erregers | M [1..1] |
<th></th> | Darstellung als Spaltenüberschriften |
| 2 | Wirkstoff | M | <td></td> | Antibiotischer Wirkstoff |
| 3 | Resistenzkennung | M | <td></td> | Codiert! Siehe Tabelle 13. (Am Schnittpunkt von Erreger (Spalte) und Wirkstoff (Zeile)) |
| minimale Hemmkonzentration | < table> je Antibiotikum Zeile <tr></tr> | |||
| 1 | Name des Erregers, sowie Einheit der Konzentration | M [1..1] |
<th></th> | Darstellung als Spaltenüberschriften |
| 2 | Wirkstoff | M | <td></td> | Antibiotischer Wirkstoff |
| 3 | Konzentration | M | <td></td> | Schnittpunkt von Erreger (Spalte) und Wirkstoff (Zeile) |
| Testergebnisse / Molekularer Erregernachweis | < table> je Analyse/Erreger eine Zeile <tr></tr> | |||
| 1 | Analyse / Erreger / Methode | M [1..1] |
<td></td> | |
| 2 | Ergebnis | M | <td></td> | |
| 3 | Einheit | O [0..1] |
<td></td> | |
| 4 | Referenzbereich / Nachweisgrenze / Linearitätsbereich | O [0..1] |
<td></td> | |
| 5 | Interpretation | R2 | <td></td> | |
1.2 Formatierung von Datums- und Zeitangaben
Datums- und Zeitangaben sind im lesbaren Teil im Format „dd.MM.yyyy“ bzw. „dd.MM.yyyy hh24:mi“ anzugeben. Dabei gilt:
| dd | Tag |
| MM | Monat als zweistellige Zahl |
| yyyy | Jahr |
| hh24 | Stunden im 24 Stunden Format |
| mi | Minuten |
1.3 Level 2 Befundstruktur
Bei der Darstellung der Befunde ist die Struktur gemäß Abbildung 8 verpflichtend abzubilden.
| Spezimen Bereich | Spezimen Section: enthält Tabelle mit Spezimen in <text> | ||
| Befundbereich (Section) | Befundgruppe | Befundgruppenbezeichnung | |
| Ergebnistabelle Befundgruppe | |||
| Bemerkung zur Befundgruppe | |||
| Befundgruppe | Befundgruppenbezeichnung | ||
| Ergebnistabelle Befundgruppe | |||
| Bemerkung zur Befundgruppe | |||
| .. | .. | ||
| Befundbereich (Section) | Befundgruppe | Befundgruppenbezeichnung | |
| Ergebnistabelle Befundgruppe | |||
| Bemerkung zur Befundgruppe | |||
| Befundgruppe | Befundgruppenbezeichnung | ||
| Ergebnistabelle Befundgruppe | |||
| Bemerkung zur Befundgruppe | |||
| .. | .. | ||
| .. | ... | ||
| Befund Bemerkung Bereich | Befundbemerkung Section enthält: Bemerkung zu Befund in <text> | ||
Enthält ein Befund nur genau einen Bereich (Section) kann eine vereinfachte Darstellung mit folgender Befundstruktur verwendet werden:
| Tabelle mit Spezimen | ||
| Befundgruppe | Befundgruppenbezeichnung | |
| Ergebnistabelle Befundgruppe | ||
| Bemerkung zur Befundgruppe | ||
| Befundgruppe | Befundgruppenbezeichnung | |
| Ergebnistabelle Befundgruppe | ||
| Bemerkung zur Befundgruppe | ||
| ... | ... | |
| Bemerkung zur Befundart | ||
1.4 Probeninformation
Der Inhalt dieser Sektion enthält sämtliche Information über das zu befundende Material, inklusive, soweit sinnvoll, der Lokalisation, der Entnahmeart, des Entnahmegeräts, der Person, welche die Entnahme durchgeführt hat, sowie Zeitpunkt der Materialentnahme und der Materialannahme.
Menschenlesbare Informationen zum Spezimen MÜSSEN angegeben werden, wenn bekannt.
In dem folgenden Strukturbeispiel ist die Codierung der Informationen in einer Tabelle ersichtlich. Die einzelnen Zeilen, welche jeweils ein Spezimen codieren, können mit Identifikatoren gekennzeichnet werden um auf diese im weiteren CDA Befund referenzieren zu können. Die Optimierung der Spaltenbreiten kann im ELGA Referenz-Stylesheet durch die ELGA-Stylecodes „xELGA_colw:nn“ erfolgen.
Die Abbildung der Spezimeninformation kann auf zwei Arten erfolgen:
- Enthält ein Befund nur einen Bereich, so kann die Codierung gemäß IHE LAB TF-3 innerhalb der einen Befundsektion erfolgen
ODER
- Bei Verwendung von mehreren Bereichen (vgl. 4.2.1) in einem Laborbefund kann es zu Überschneidungen der Spezimeninformationen kommen (ein spezielles Spezimen kann in zwei Bereichen analysiert werden). Die Level 3 Codierung eines Spezimens darf jedoch nur einmal im gesamten Laborbefund erfolgen. Daher sind die Informationen zu den Spezimen in einer eigenen führenden Probeninformation Section mit dem Code „10“ und der TemplateID 1.2.40.0.34.11.4.2.1 zu codieren.
1.5 Vorgaben zur Darstellung einzelner Elemente
1.5.1 Analysen
Analysen (bzw Laborwerte, Laborleistungen oder Labormessgrößen) MÜSSEN in der einheitlichen Schreibweise angegeben sein, die im Value Set „ELGA_Laborparameter“ vorgegeben wird („Begriff“ bzw „display name“ im Value Set). Das erleichtert das Lesen und speziell für Patienten die Recherche von Laborwerten im Gesundheitsportal (www.gesundheit.gv.at). Siehe dazu auch den „Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund“ [9].
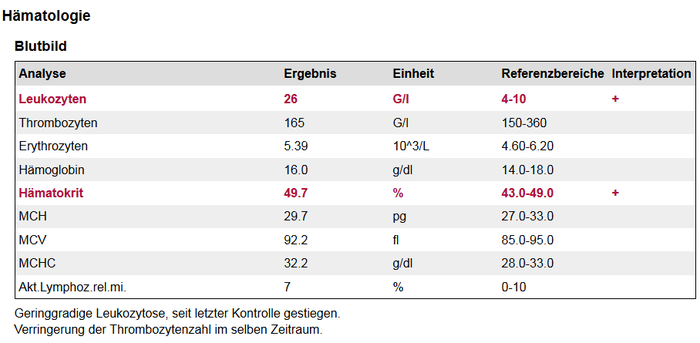
Ein Beispiel zur Darstellung findet sich in Abbildung 12. Die Tabelle besteht aus mindestens fünf und maximal sechs Spalten. Für jede Gruppe wird ein Block angelegt, Bereichsüberschriften entsprechen Kapitelüberschriften. Zusätzlich kann eine Spalte mit „Extenes Labor“ notwendig sein. Sollen genauere Angaben zur Methode gemacht werden, als durch den Namen der Analysen bereits hervorgeht, soll dies über einen Analysenkommentar umgesetzt werden (siehe 4.3.8.1).
1.5.2 Ergebnis
Dieses Element enthält ein numerisches, nominales, ordinales oder narratives Ergebnis der Analyse zu diesem Testcode. Da in der Definition des CDA-Schemas keinerlei Längenbeschränkung vorgegeben ist, kann dieses Feld auch größere Textmengen fassen um große verbale Beurteilungen zu ermöglichen.
1.5.3 Einheit
Zu jedem Ergebnis MUSS eine passende Einheit angegeben werden. Bevorzugt zu verwenden sind die Einheiten, die im Value Set „ELGA_Laborparameter“ vorgeschlagen werden.
Es wird EMPFOHLEN, anstelle von Einheitenpräfixen („Giga“, „Mega“, „Milli“, „Mikro“ etc.) eine Potenzschreibweise zu wählen, vor allem, wenn die Groß/Klein-Schreibung eine Rolle spielt und Verwechslungen möglich sind (z.B. „G/L“=Giga pro Liter vs. „g/L“=Gramm/Liter). Also '10^6 ' statt 'M' (Mega), '10^9 ' statt 'G ' (Giga) usw.
1.5.4 Befundinterpretation
Es ist in Laborbefunden üblich, eine codierte Bewertung zu jedem Ergebnis anzugeben. Häufig wird eine Notierung mit +/- verwendet.
Folgende Tabelle 7 ist ein Auszug aus dem Value Set „ELGA_ObservationInterpretation“ und zeigt die normative Befundinterpretation für numerische Ergebnisse, Tabelle 8 (ebenfalls aus dem gleichen Value Set) die Kennzeichnung für nicht numerische Ergebnisse.
| Darstellung Level 2 | Codierung Level 3 | Beschreibung |
|---|---|---|
| ++ | HH | Oberhalb des Referenzbereiches und über einer oberen Warngrenze |
| + | H | Oberhalb des Referenzbereiches |
| N | Normal (innerhalb des Referenzbereiches) | |
| - | L | Unterhalb des Referenzbereiches |
| -- | LL | Unterhalb des Referenzbereiches und unter einer unteren Warngrenze |
Tabelle 7: Befundinterpretation numerischer Ergebnisse
| Darstellung Level 2 | Codierung Level 3 | Beschreibung |
|---|---|---|
| N | Normal (innerhalb des Referenzbereiches) | |
| * | A | Abnormal |
| ** | AA | Abnormal Warngrenze |
Tabelle 8: Befundinterpretation nicht numerischer Ergebnisse (nominal, ordinal, narrativ)
Zur Interpretation von Ergebnissen der Allergiediagnostik wurden zusätzlich RAST-Klassen als Klassifikation erlaubt, siehe Kapitel 4.3.6.
1.6 Empfehlungen für die Darstellung der Allergiediagnostik
In der Allergiediagnostik gibt es gegebenenfalls Abweichungen zur normalen Struktur des Laborbefundes. Die Angabe der getesteten Allergene bei Globalmarkern oder die zusätzliche Angabe von RAST-Klassen machen eine alternative Darstellung notwendig.
Folgende Darstellung wird dazu EMPFOHLEN:
Folgendes Codebeispiel zeigt die Befunddarstellung der Globalmarker im narrativen Text:
<table>
<thead>
<tr>
<th styleCode="xELGA_colw:80">Analyse</th>
<th styleCode="xELGA_colw:10">Ergebnis</th>
<th styleCode="xELGA_colw:10">Interpretation</th>
</tr>
</thead>
<tbody>
<tr ID="OBS-1-1">
<td>sx1 Inhalatives Screening<br/>
<content styleCode="italics">Lieschgras, Roggen, Birke, Beifuß, Dermatophagoides pteronyssinus,
Katzenschuppen, Hundeschuppen, Cladosporium herbarum</content>
</td>
<td>negativ</td>
<td></td>
</tr>
<tr ID="OBS-1-2" styleCode="xELGA_red">
<td>mx1 Schimmelpilzemix 1<br/>
<content styleCode="italics">Alternaria alternata, Aspergillus fumigatus, Cladosporium herbarum,
Penicillium notatum</content></td>
<td>positiv</td>
<td>A</td>
</tr>
</tbody>
</table>
1.7 Stylecodes
Für die spezifische grafische Darstellung und bessere optische Aufbereitung stehen verschiedene definierte Stylecodes zur Verfügung. Tabelle 9 zeigt einen Überblick.