|
|
| (197 dazwischenliegende Versionen von 4 Benutzern werden nicht angezeigt) |
| Zeile 3: |
Zeile 3: |
| | |titlemode=append | | |titlemode=append |
| | |keywords= Laborbefund, Mikrobiologiebefund, Labor, Mikrobiologie | | |keywords= Laborbefund, Mikrobiologiebefund, Labor, Mikrobiologie |
| − | |description=Der Labor- und Mikrobiologiebefund enthält Ergebnisse zu Labor- und Mikrobiologieuntersuchungen durchgeführt an einer Probe eines Patienten. | + | |description=Der Labor- und Mikrobiologiebefund enthält Ergebnisse zu Labor- und Mikrobiologieuntersuchungen durchgeführt an dem Untersuchungsmaterial eines Patienten. |
| | }} | | }} |
| | | | |
| − | {{Underconstruction}}
| + | {{#customtitle:Labor- und Mikrobiologiebefund (Version 3.0.0+20211214)}} |
| − | | |
| − | {{#customtitle:Labor- und Mikrobiologiebefund (Version 3.0.0)}} | |
| | | | |
| | <!-- Implementierungsleitfaden Metadaten--> | | <!-- Implementierungsleitfaden Metadaten--> |
| Zeile 18: |
Zeile 16: |
| | |Namespace = ILF | | |Namespace = ILF |
| | |Type = Implementierungsleitfaden | | |Type = Implementierungsleitfaden |
| − | |Version = 3.0.0 | + | |Version = 3.0.0+20211214 |
| | |Submitted = ELGA GmbH | | |Submitted = ELGA GmbH |
| | |Copyright = © HL7 Austria 2021 | | |Copyright = © HL7 Austria 2021 |
| − | |Date = 2021.xx.xx | + | |Date = 14.12.2021 |
| | |Status = Normativ | | |Status = Normativ |
| | |Verfahren = Normatives Abstimmungsverfahren | | |Verfahren = Normatives Abstimmungsverfahren |
| Zeile 29: |
Zeile 27: |
| | }} | | }} |
| | | | |
| − | {{ILF-TOC limit}} | + | {{ILF-TOC limit|5}} |
| | | | |
| | <!-- Zusammenfassung an erster Stelle --> | | <!-- Zusammenfassung an erster Stelle --> |
| − | {{BeginYellowBox}} | + | {{BeginYellowBox}}Dieses Dokument bildet den '''vollständigen Implementierungsleitfaden des Labor- und Mikrobiologiebefundes''' ab und richtet sich an Softwareentwickler und Berater. Zum besseren Verständnis empfehlen wir Ihnen, den [[ILF:Laborbefund_Guide|'''zusammenfassenden Guide''']] im Vorfeld zu lesen. |
| − | Dieses Dokument bildet den '''vollständigen Implementierungsleitfaden des Labor- und Mikrobiologiebefundes''' ab und richtet sich an Softwareentwickler und Berater. Zum besseren Verständnis empfehlen wir Ihnen, den [[ILF:Laborbefund_Guide|'''zusammenfassenden Guide''']] im Vorfeld zu lesen. | |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| | =Zusammenfassung= | | =Zusammenfassung= |
| − | Der Implementierungsleitfaden "Labor- und Mikrobiologiebefund" beschreibt die Inhalte, die für den Austausch von fertiggestellten, und fachärztlich vidierten Befunden innerhalb und zwischen Einrichtungen des österreichischen Gesundheitswesens, notwendig sind. | + | Der Implementierungsleitfaden "Labor- und Mikrobiologiebefund" beschreibt die Inhalte, die für den Austausch von fertiggestellten und fachärztlich vidierten Befunden, innerhalb und zwischen Einrichtungen des österreichischen Gesundheitswesens, notwendig sind. |
| | | | |
| | Der Leitfaden enthält Festlegungen, Einschränkungen und Bedingungen auf Grundlage des internationalen Standards ISO/HL7 27932:2009 HL7 Clinical Document Architecture, Release 2.0 (CDA) und ist ein nationaler Standard der HL7 Austria. | | Der Leitfaden enthält Festlegungen, Einschränkungen und Bedingungen auf Grundlage des internationalen Standards ISO/HL7 27932:2009 HL7 Clinical Document Architecture, Release 2.0 (CDA) und ist ein nationaler Standard der HL7 Austria. |
| Zeile 44: |
Zeile 41: |
| | Der "Labor- und Mikrobiologiebefund" basiert auf den Vorgaben des [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeinen Implementierungsleitfadens]]. Darin werden die notwendigen Datentypen, Dokument-Metadaten (Header), die Möglichkeiten der Textstrukturierung, grundlegende Vorgaben für die Anwendung von Terminologien, einige allgemein genutzten Inhaltsstrukturen (Sections) sowie Codebeispiele und praktische Implementierungshilfen gezeigt. Alle weiteren, für diesen Leitfaden benötigten Elemente werden hier erklärt. Die Notation der Spezifikation der Datenaustauschformate folgt der "Art-Decor"-Schreibweise, die auf einer eigenen Seite ([[Hilfe:Art-Decor-Tabellen_verstehen|Art-Decor-Tabellen verstehen]]) erläutert wird. | | Der "Labor- und Mikrobiologiebefund" basiert auf den Vorgaben des [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeinen Implementierungsleitfadens]]. Darin werden die notwendigen Datentypen, Dokument-Metadaten (Header), die Möglichkeiten der Textstrukturierung, grundlegende Vorgaben für die Anwendung von Terminologien, einige allgemein genutzten Inhaltsstrukturen (Sections) sowie Codebeispiele und praktische Implementierungshilfen gezeigt. Alle weiteren, für diesen Leitfaden benötigten Elemente werden hier erklärt. Die Notation der Spezifikation der Datenaustauschformate folgt der "Art-Decor"-Schreibweise, die auf einer eigenen Seite ([[Hilfe:Art-Decor-Tabellen_verstehen|Art-Decor-Tabellen verstehen]]) erläutert wird. |
| | | | |
| − | Der vorgesehene Ablauf des Datenaustausches wird im Kapitel [[#Anwendungsfälle|Anwendungsfälle]] beschrieben. | + | Der vorgesehene Ablauf des Datenaustausches wird im Kapitel [[#Anwendungsfälle / User Stories|Anwendungsfälle / User Stories]] beschrieben. |
| | | | |
| | '''Übersichtstabellen für Header und Body-Strukturen''' | | '''Übersichtstabellen für Header und Body-Strukturen''' |
| Zeile 67: |
Zeile 64: |
| | ''Abbildungen:'' © ELGA GmbH | | ''Abbildungen:'' © ELGA GmbH |
| | | | |
| − | ''Nutzung'': Das Dokument enthält geistiges Eigentum der Health Level Seven® Int. und HL7® Austria, Franckstrasse 41/5/14, 8010 Graz; [http://www.hl7.at www.hl7.at].<br /> | + | ''Nutzung'': Das Dokument enthält geistiges Eigentum der Health Level Seven® Int. und HL7® Austria, Erdbergweg 7/8, 8052 Graz; [http://www.hl7.at www.hl7.at].<br /> |
| | Die Nutzung ist ohne Lizenz- und Nutzungsgebühren zum Zweck der Erstellung medizinischer Dokumente ausdrücklich erlaubt. Andere Arten der Nutzung und auch auszugsweise Wiedergabe bedürfen der Genehmigung des Medieneigentümers. | | Die Nutzung ist ohne Lizenz- und Nutzungsgebühren zum Zweck der Erstellung medizinischer Dokumente ausdrücklich erlaubt. Andere Arten der Nutzung und auch auszugsweise Wiedergabe bedürfen der Genehmigung des Medieneigentümers. |
| | | | |
| Zeile 73: |
Zeile 70: |
| | </div></div> | | </div></div> |
| | <div class="toccolours mw-collapsible mw-collapsed" overflow:auto;"> | | <div class="toccolours mw-collapsible mw-collapsed" overflow:auto;"> |
| | + | |
| | ==Haftungsausschluss== | | ==Haftungsausschluss== |
| | <div class="mw-collapsible-content"> | | <div class="mw-collapsible-content"> |
| Zeile 83: |
Zeile 81: |
| | </div></div> | | </div></div> |
| | {{ILF:Lizenzinformationen}} | | {{ILF:Lizenzinformationen}} |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Der Labor- und Mikrobiologiebefund basiert auf den Vorgaben des '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeinen Implementierungsleitfadens (Version 3)]]'''. |
| − | Der Labor- und Mikrobiologiebefund basiert auf den Vorgaben des '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeinen Implementierungsleitfadens (Version 3)]]'''. | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| | Für die Modellierung der technischen Spezifikation der Inhalte des Labor- und Mikrobiologiebefundes wurde das "Pathology and Laboratory Medicine (PaLM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019"<ref name="IHEPaLMTF3" /> der Integrating the Healthcare Enterprise (IHE) als wesentliche Grundlage gewählt. | | Für die Modellierung der technischen Spezifikation der Inhalte des Labor- und Mikrobiologiebefundes wurde das "Pathology and Laboratory Medicine (PaLM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019"<ref name="IHEPaLMTF3" /> der Integrating the Healthcare Enterprise (IHE) als wesentliche Grundlage gewählt. |
| − | ==Verbindlichkeit== | + | ==Verbindlichkeit und rechtliche Grundlagen== |
| | Die Verbindlichkeit und die Umsetzungsfrist dieses Leitfadens sind im Gesundheitstelematikgesetz 2012, BGBl.I Nr.111/2012 sowie in den darauf fußenden ELGA-Verordnungen geregelt. | | Die Verbindlichkeit und die Umsetzungsfrist dieses Leitfadens sind im Gesundheitstelematikgesetz 2012, BGBl.I Nr.111/2012 sowie in den darauf fußenden ELGA-Verordnungen geregelt. |
| | | | |
| | Der Leitfaden in seiner jeweils aktuell gültigen Fassung sowie die aktualisierten Terminologien sind vom zuständigen Minister auf [https://www.gesundheit.gv.at/ www.gesundheit.gv.at] zu veröffentlichen. Der Zeitplan zur Bereitstellung der Datenaustauschformate wird durch das Gesundheitstelematikgesetz 2012 und darauf basierenden Durchführungsverordnungen durch den zuständigen Bundesminister vorgegeben. Hauptversionen, also Aktualisierungen des Implementierungsleitfadens, die verpflichtende Änderungen in den erstellenden Systemen nach sich ziehen, sind mit ihren Fristen zur Bereitstellung per Verordnung kundzumachen. Andere Aktualisierungen (Nebenversionen) dürfen auch ohne Änderung dieser Verordnung unter [https://www.gesundheit.gv.at/ www.gesundheit.gv.at] veröffentlicht werden. | | Der Leitfaden in seiner jeweils aktuell gültigen Fassung sowie die aktualisierten Terminologien sind vom zuständigen Minister auf [https://www.gesundheit.gv.at/ www.gesundheit.gv.at] zu veröffentlichen. Der Zeitplan zur Bereitstellung der Datenaustauschformate wird durch das Gesundheitstelematikgesetz 2012 und darauf basierenden Durchführungsverordnungen durch den zuständigen Bundesminister vorgegeben. Hauptversionen, also Aktualisierungen des Implementierungsleitfadens, die verpflichtende Änderungen in den erstellenden Systemen nach sich ziehen, sind mit ihren Fristen zur Bereitstellung per Verordnung kundzumachen. Andere Aktualisierungen (Nebenversionen) dürfen auch ohne Änderung dieser Verordnung unter [https://www.gesundheit.gv.at/ www.gesundheit.gv.at] veröffentlicht werden. |
| | | | |
| − | Die Anwendung dieses Implementierungsleitfadens hat im Einklang mit der Rechtsordnung der Republik Österreich und insbesondere mit den relevanten Materiengesetzen (z.B. Ärztegesetz 1998, Apothekenbetriebsordnung 2005, Krankenanstalten- und Kuranstaltengesetz, Gesundheits- und Krankenpflegegesetz, Rezeptpflichtgesetz, Datenschutzgesetz 2000, Gesundheitstelematikgesetz 2012) zu erfolgen. Technische Möglichkeiten können gesetzliche Bestimmungen selbstverständlich nicht verändern, vielmehr sind die technischen Möglichkeiten im Einklang mit den Gesetzen zu nutzen. | + | Die Anwendung dieses Implementierungsleitfadens hat im Einklang mit österreichischem und europäischem Recht, insbesondere mit den relevanten Materiengesetzen (z.B. Ärztegesetz 1998, Apothekenbetriebsordnung 2005, Krankenanstalten- und Kuranstaltengesetz, Gesundheits- und Krankenpflegegesetz, Rezeptpflichtgesetz, Datenschutzgesetz, Gesundheitstelematikgesetz 2012, DSGVO) zu erfolgen. Technische Möglichkeiten können gesetzliche Bestimmungen selbstverständlich nicht verändern, vielmehr sind die technischen Möglichkeiten im Einklang mit den Gesetzen zu nutzen. |
| | | | |
| | Die Einhaltung der gesetzlichen Bestimmungen liegt im Verantwortungsbereich der Ersteller der CDA-Dokumente. | | Die Einhaltung der gesetzlichen Bestimmungen liegt im Verantwortungsbereich der Ersteller der CDA-Dokumente. |
| | + | {{BeginYellowBox}}Bitte stellen Sie sicher, dass Sie die '''datenschutzrechtliche Einwilligung von dritten Personen''' (Probenabnehmende Person o.Ä.), die im Befund dokumentiert werden, haben. |
| | + | {{EndYellowBox}} |
| | + | |
| | ==Wichtige unterstützende Materialien== | | ==Wichtige unterstützende Materialien== |
| − | {{BeginYellowBox}} | + | {{BeginYellowBox}}Auf der Website [[ILF:Labor-_und_Mikrobiologiebefund_Guide|Labor- und Mikrobiologiebefund Guide]] werden unter anderem folgende Materialien zur Verfügung gestellt: |
| − | Auf der Website [[ILF:Laborbefund_Guide|Labor- und Mikrobiologiebefund Guide]] werden unter anderem folgende Materialien zur Verfügung gestellt: | |
| | * die PDF-Version dieses Leitfadens | | * die PDF-Version dieses Leitfadens |
| | * Beispieldokumente | | * Beispieldokumente |
| Zeile 105: |
Zeile 104: |
| | Die im weiteren angeführten Templatespezifikationen wurden im Art-Decor Projektrepository [https://art-decor.org/art-decor/decor-templates--at-lab-?section=templates Labor- und Mikrobiologiebefund] erstellt und können dort eingesehen werden. | | Die im weiteren angeführten Templatespezifikationen wurden im Art-Decor Projektrepository [https://art-decor.org/art-decor/decor-templates--at-lab-?section=templates Labor- und Mikrobiologiebefund] erstellt und können dort eingesehen werden. |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| − | Gemeinsam mit diesem Leitfaden werden auf der Website der ELGA GmbH ([http://www.elga.gv.at/CDA www.elga.gv.at/CDA]) weitere Dateien und Dokumente zur Unterstützung bereitgestellt: | + | Gemeinsam mit diesem Leitfaden werden auf der [http://www.elga.gv.at/CDA Website der ELGA GmbH] weitere Dateien und Dokumente zur Unterstützung bereitgestellt: |
| | *Beispieldokumente | | *Beispieldokumente |
| | *Referenz-Stylesheet (Tool zur Darstellung im Browser - Konvertierung in HTML) | | *Referenz-Stylesheet (Tool zur Darstellung im Browser - Konvertierung in HTML) |
| Zeile 113: |
Zeile 112: |
| | *Hinweise für die zu verwendenden Terminologien | | *Hinweise für die zu verwendenden Terminologien |
| | *Leitfaden zur richtigen Verwendung von Terminologien | | *Leitfaden zur richtigen Verwendung von Terminologien |
| − | {{BeginYellowBox}} | + | {{BeginYellowBox}}Fragen, Kommentare oder Anregungen für die Weiterentwicklung können an [mailto:cda@elga.gv.at cda@elga.gv.at] gesendet werden. |
| − | Fragen, Kommentare oder Anregungen für die Weiterentwicklung können an [mailto:cda@elga.gv.at cda@elga.gv.at] gesendet werden. Weitere Informationen finden Sie unter [http://www.elga.gv.at/CDA www.elga.gv.at/CDA]. | |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| | + | |
| | ==Bedienungshinweise== | | ==Bedienungshinweise== |
| | ===Farbliche Hervorhebungen und Hinweise=== | | ===Farbliche Hervorhebungen und Hinweise=== |
| | ''<u>Themenbezogene Hinweise zur besonderen Beachtung:</u>'' | | ''<u>Themenbezogene Hinweise zur besonderen Beachtung:</u>'' |
| − | {{BeginYellowBox}} | + | {{BeginYellowBox}}'''Hinweis:'''<br />Es dürfen keine Elemente oder Attribute verwendet werden, die nicht vom allgemeinen oder einem speziellen ELGA-Implementierungsleitfaden definiert wurden |
| − | '''Hinweis:'''<br />Es dürfen keine Elemente oder Attribute verwendet werden, die nicht vom allgemeinen oder einem speziellen ELGA-Implementierungsleitfaden definiert wurden | |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| | ''<u>Hinweis auf anderen Implementierungsleitfaden:</u>'' | | ''<u>Hinweis auf anderen Implementierungsleitfaden:</u>'' |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}'''Verweis'''<br />Verweis auf den Allgemeinen Leitfaden:… |
| − | '''Verweis'''<br />Verweis auf den Allgemeinen Leitfaden:… | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| | ''<u>Themenbezogenes CDA Beispiel-Fragment im XML Format:</u>'' | | ''<u>Themenbezogenes CDA Beispiel-Fragment im XML Format:</u>'' |
| Zeile 144: |
Zeile 141: |
| | | | |
| | <!-- Tatsächlicher Inhalt --> | | <!-- Tatsächlicher Inhalt --> |
| | + | |
| | + | =Begriffsdefinitionen= |
| | + | {| class="wikitable" style="margin-left: 16px; margin-right: 16px;" |
| | + | !Begriff |
| | + | !Definition |
| | + | |- |
| | + | |'''Laborbefund''' |
| | + | |Ein Laborbefund (aus dem Bereich der med. u. chem. Labordiagnostik) ist der fachärztlich vidierte, kommentierte/interpretierte Befund morphologischer, biologischer, chemischer, molekularer, physikalischer und spezieller immunologischer Analyseverfahren aus Körpersäften, der Beurteilung ihrer morphologischen Bestandteile sowie von ab- und ausgeschiedenem Untersuchungsmaterial zur Erkennung physiologischer Eigenschaften, krankhafter Zustände, zu Verlaufskontrollen und zur Gesundheitsvorsorge/Prophylaxe. |
| | + | |
| | + | Das Verständnis eines Laborbefundes im Rahmen dieses Leitfadens erstreckt sich dabei über das gesamte Spektrum der laboranalytisch ermittelten Befunde. Laborbefunde umfassen u. a. klinische Chemie und Immunchemie, Hämatologie, Hämostaseologie, Proteinchemie, Serologie, molekulare Diagnostik, Toxikologie, Drugmonitoring, Mikrobiologie, Infektionsserologie, Zytologie, Untersuchungen und die Hilfestellung für andere Fächer im Rahmen von Therapievorschlägen bei Gerinnungsstörungen, Antikoagulanzientherapien, der Impfkontrolle, Vorsorgediagnostik und Risikostratifizierung. Die gewählten Strukturen ermöglichen prinzipiell eine Übermittlung des gesamten Befundspektrums des Laborbereiches. |
| | + | {{BeginYellowBox}}Analysen des Sonderfaches "Blutgruppenserologie und Transfusionsmedizin" werden in einer gesonderten ELGA Dokumentenklasse (geplant) abgehandelt. |
| | + | {{EndYellowBox}} |
| | + | {{BeginYellowBox}}Im ELGA Laborbefund dürfen nur dann Ergebnisse aus genetischen Analysen enthalten sein, wenn ihre Dokumentation in Übereinstimmung mit dem Gentechnikgesetz (GTG § 71a, BGBl. I Nr. 127/2005) erfolgt. |
| | + | {{EndYellowBox}} |
| | + | |- |
| | + | |'''Mikrobiologiebefund''' |
| | + | |Der Mikrobiologiebefund als spezieller Laborbefund umfasst im Allgemeinen die Bereiche der Beschreibung des entnommenen Materials inklusive einer makroskopischer Beurteilung, die mikroskopische Analyse des Materials, kulturelle Erregernachweise (inkl. Antibiogramm), molekularer Erregernachweise sowie der Infektionsserologie. |
| | + | |- |
| | + | |'''Analyse''' |
| | + | |Sammelbegriff für Laboruntersuchung, Laborleistung und Labormessgröße. |
| | + | |- |
| | + | |'''Erreger''' |
| | + | |Erreger oder Krankheitserreger sind Stoffe oder Organismen, die in anderen Organismen potenziell gesundheitsschädigende Abläufe verursachen können. |
| | + | |- |
| | + | |'''Untersuchungsmaterial''' |
| | + | |Sammelbegriff für Spezimen, Probe, etc. |
| | + | |
| | + | Die englische Bezeichnung "Specimen" wird vorwiegend dann verwendet, wenn sich der Kontext auf das IHE PaLM TF3<ref name="IHEPaLMTF3" /> oder auf das RIM<ref name="RIM"/> bezieht. |
| | + | |} |
| | | | |
| | =Einleitung= | | =Einleitung= |
| | ==Ausgangslage und Motivation== | | ==Ausgangslage und Motivation== |
| − | Mit dem "Implementierungsleitfaden für Laborbefunde" konnte über viele Jahre bereits Erfahrung im Bereich der CDA-basierten Labordokumentation gesammelt werden. Im Laufe der Zeit haben sich neue Anforderungen - vor allem im Bereich der Mikrobiologie - ergeben bzw. wurde es z.B. durch die Lizensierung von SNOMED CT möglich gemacht, einzelne Bereiche vollständig zu codieren. Insofern überarbeitet und erweitert der gegenständliche Implementierungsleitfaden das Spektrum der codierbaren Daten grundsätzlich. Vor allem der "Mikrobiologiebefund" ermöglicht es, Befunde im Sinne des üblichen Untersuchungsverlaufs der Mikrobiologie abzubilden. In Summe soll dadurch die Datenqualität erhöht, der Informationsabgleich verbessert und die Interpretation der Information erleichtert werden. | + | Mit dem "Implementierungsleitfaden für Laborbefunde" konnte über viele Jahre bereits Erfahrung im Bereich der CDA-basierten Labordokumentation gesammelt werden. Im Laufe der Zeit haben sich neue Anforderungen - vor allem im Bereich der Mikrobiologie - ergeben bzw. wurde es z.B. durch die österreichweite Verfügbarkeit von SNOMED CT möglich gemacht, einzelne Bereiche vollständig zu codieren. Insofern überarbeitet und erweitert der gegenständliche Implementierungsleitfaden das Spektrum der codierbaren Daten. Vor allem der "Mikrobiologiebefund" ermöglicht es, Befunde im Sinne des üblichen Untersuchungsverlaufs der Mikrobiologie abzubilden. In Summe soll dadurch die Datenqualität erhöht, der Informationsabgleich verbessert und die Interpretation der Information erleichtert werden. |
| | + | |
| | + | Der "Labor- und Mikrobiologiebefund" basiert auf den Vorgaben des [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeinen Implementierungsleitfadens]] und aktualisiert und erweitert den bestehenden ELGA CDA Implementierungsleitfaden Labor 2.06.2. |
| | + | |
| | ==Zweck des Dokuments== | | ==Zweck des Dokuments== |
| | | | |
| | Der vorliegende "Implementierungsleitfaden für den Labor- und Mikrobiologiebefund" beschreibt die einheitliche Implementierungsvorschrift für den Informationsaustausch von Labor- und Mikrobiologiebefunden im österreichischen Gesundheitswesen. Der Leitfaden basiert auf den vorangegangenen Erfahrungen in der Erstellung von Implementierungsleitfäden für ELGA CDA Dokumente. | | Der vorliegende "Implementierungsleitfaden für den Labor- und Mikrobiologiebefund" beschreibt die einheitliche Implementierungsvorschrift für den Informationsaustausch von Labor- und Mikrobiologiebefunden im österreichischen Gesundheitswesen. Der Leitfaden basiert auf den vorangegangenen Erfahrungen in der Erstellung von Implementierungsleitfäden für ELGA CDA Dokumente. |
| | | | |
| − | Der sogenannte "Header" beinhaltet zum einen administrative Daten (allgemeine Angaben zum Dokument, Daten zum Patienten, usw.) und dient zum anderen auch als Quelle für die Metadaten, die bei der Registrierung des Dokuments in ELGA verwendet werden. Der Header wurde über alle Anwendungsbereiche der ELGA einheitlich abgestimmt. Die medizinisch relevanten Daten, die im Rahmen von Laboruntersuchungen erfasst werden, sind im sogenannten "Body" enthalten. Die vorliegende Spezifikation der laborspezifischen inkl. mikrobiologische Inhalte eines Labordokuments in ELGA wurde von der Expertengruppe beruhend auf einer Liste mit Vorgaben der österreichischen Gesellschaft für Labormedizin und klinische Chemie (ÖGLMKC) erstellt. | + | Der sogenannte "Header" beinhaltet zum einen administrative Daten (allgemeine Angaben zum Dokument, Daten zum Patienten, usw.) und dient zum anderen auch als Quelle für die Metadaten, die bei der Registrierung des Dokuments in ELGA verwendet werden. Der Header wurde über alle Anwendungsbereiche der ELGA einheitlich abgestimmt. Die medizinisch relevanten Daten, die im Rahmen von Analysen erfasst werden, sind im sogenannten "Body" enthalten. Die vorliegende Spezifikation der laborspezifischen inkl. mikrobiologischen Inhalte eines Laborbefundes in ELGA wurde von der Expertengruppe beruhend auf einer Liste mit Vorgaben der österreichischen Gesellschaft für Labormedizin und klinische Chemie (ÖGLMKC) erstellt. |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Elemente des Headers und Bodys orientieren sich am bestehenden "[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeiner Implementierungsleitfaden für ELGA CDA Dokumente]]". |
| − | Elemente des Headers und Bodys orientieren sich am bestehenden "[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeiner Implementierungsleitfaden für ELGA CDA Dokumente]]".{{EndILFBox}} | + | {{EndILFBox}} |
| − | | |
| − | Das Verständnis eines "Laborbefundes" erstreckt sich in diesem Dokument über das gesamte Spektrum der laboranalytisch ermittelten Befunde. Die vorliegende Version definiert grundlegende Anforderungen für die Erstellung von Laborbefunden als CDA Dokumente. Insbesondere wurden Laborbefunde aus der Klinischen Chemie, Hämatologie, Immunchemie und Mikrobiologie/Bakteriologie in die Überlegungen mit einbezogen. Die gewählten Strukturen ermöglichen prinzipiell eine Übermittlung des gesamten Befundspektrums des Laborbereiches, jedoch sind die einzelnen Detailbereiche in folgenden Arbeiten detailliert zu analysieren, abzustimmen und für weitere Laborbefundarten zu definieren. Es existieren vielmehr auch dezidierte Bereiche - wie z.B. die Transfusionsmedizin – für die die Definitionen dieses Leitfadens aufgrund fehlender Strukturen und nicht definierter Codelisten nicht ausreichend sind. Dieser Leitfaden verwendet "Analysen" als Sammelbegriff für Laboruntersuchungen, Laborleistungen und Labormessgrößen.
| |
| − | | |
| − | Der "Mikrobiologiebefund" umfasst im Allgemeinen die Bereiche der Beschreibung des entnommenen Materials inklusive einer makroskopischer Beurteilung, die mikroskopische Untersuchung des Materials, kulturelle Erregernachweise (inkl. Antibiogramm), molekularer Erregernachweise sowie der Infektionsserologie.
| |
| | | | |
| | ==Zielgruppe== | | ==Zielgruppe== |
| Zeile 169: |
Zeile 194: |
| | Ein wesentlicher Schritt auf dem Weg zur Interoperabilität der IT-Systeme im Gesundheitswesen ist die Einigung auf Vorgaben für einheitliche Dokumentation und Codierung der Information. Diese durch die Arbeitsgruppen erreichte "Harmonisierung" etabliert neue nationale Qualitätsstandards der medizinischen Dokumentation. | | Ein wesentlicher Schritt auf dem Weg zur Interoperabilität der IT-Systeme im Gesundheitswesen ist die Einigung auf Vorgaben für einheitliche Dokumentation und Codierung der Information. Diese durch die Arbeitsgruppen erreichte "Harmonisierung" etabliert neue nationale Qualitätsstandards der medizinischen Dokumentation. |
| | Die Leitfäden werden über ein reguläres Standardisierungsverfahren ("Ballot") durch die HL7 Anwendergruppe Österreich (HL7 Austria) zu einem nationalen HL7 Standard. | | Die Leitfäden werden über ein reguläres Standardisierungsverfahren ("Ballot") durch die HL7 Anwendergruppe Österreich (HL7 Austria) zu einem nationalen HL7 Standard. |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Weitere Details zum Vorgehensmodell sind im [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Vorgehensmodell| Allgemeiner Leitfaden - Kapitel Leitfadenerstellungs- und Harmonisierungsprozess - Vorgehensmodell]] zu finden. |
| − | Weitere Details zum Vorgehensmodell sind im [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Vorgehensmodell| Allgemeiner Leitfaden - Kapitel Leitfadenerstellungs- und Harmonisierungsprozess - Vorgehensmodell]] zu finden. | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| | | | |
| − | Dieser Implementierungsleitfaden ist eine Weiterentwicklung des Laborbefundes 2.06.3 und entstand durch die Harmonisierungsarbeit der ''AG Labor- und Mikrobiologiebefund'', die im ''Zeitraum von [TODO] Oktober 2020 bis November 2020'' tagte. Die Teilnehmer der Arbeitsgruppe wurden durch ihre Organisation delegiert. | + | Dieser Implementierungsleitfaden ist eine Weiterentwicklung des Laborbefundes 2.06.2 und entstand durch die Harmonisierungsarbeit der ''AG Labor- und Mikrobiologiebefund'', die im ''Zeitraum von Oktober 2016 bis Oktober 2020'' tagte. Die Teilnehmer der Arbeitsgruppe wurden durch ihre Organisation delegiert. |
| | | | |
| | ''Die Arbeitsgruppe harmonisierte primär die inhaltlichen Vorgaben und soweit möglich die zu verwendenden Terminologien (Value Sets). Die Formulierung der technischen Spezifikation des CDA Implementierungsleitfadens Labor- und Mikrobiologiebefund erfolgte durch die ELGA GmbH parallel bzw. nach der inhaltlichen Festlegung.'' | | ''Die Arbeitsgruppe harmonisierte primär die inhaltlichen Vorgaben und soweit möglich die zu verwendenden Terminologien (Value Sets). Die Formulierung der technischen Spezifikation des CDA Implementierungsleitfadens Labor- und Mikrobiologiebefund erfolgte durch die ELGA GmbH parallel bzw. nach der inhaltlichen Festlegung.'' |
| | | | |
| − | Der Leitfaden wird in einem technischen Abstimmungsverfahren durch die HL7 Austria ("Ballot") zu einem österreichischen Standard. Die Verbindlichkeit zur Anwendung soll durch eine Novellierung des Gesundheitstelematikgesetzes 2012, BGBl.I Nr.111/2012 begründet werden. | + | Der Leitfaden wurde in einem technischen Abstimmungsverfahren durch die HL7 Austria (Ballot 2021-2) zu einem österreichischen HL7-Standard. Die Verbindlichkeit zur Anwendung wird durch eine Verordnung zum Gesundheitstelematikgesetz 2012, BGBl.I Nr.111/2012 begründet. |
| | ==Revision der Leitfäden== | | ==Revision der Leitfäden== |
| | Neue und geänderte Anforderungen sowie Verbesserungen können neue Versionen der bestehenden Spezifikationen notwendig machen. | | Neue und geänderte Anforderungen sowie Verbesserungen können neue Versionen der bestehenden Spezifikationen notwendig machen. |
| Zeile 187: |
Zeile 211: |
| | Dieser Implementierungsleitfaden entstand durch die Harmonisierungsarbeit der "Arbeitsgruppe Labor- und Mikrobiologiebefund" bestehend aus nachfolgend genannten Personen. Die Arbeiten für den vorliegenden Leitfaden wurden von den Autoren gemäß dem Stand der Technik und mit größtmöglicher Sorgfalt erbracht. | | Dieser Implementierungsleitfaden entstand durch die Harmonisierungsarbeit der "Arbeitsgruppe Labor- und Mikrobiologiebefund" bestehend aus nachfolgend genannten Personen. Die Arbeiten für den vorliegenden Leitfaden wurden von den Autoren gemäß dem Stand der Technik und mit größtmöglicher Sorgfalt erbracht. |
| | ===Autoren=== | | ===Autoren=== |
| − | '''Das Redaktionsteam''' bestand aus folgenden Personen: | + | '''Das Redaktionsteam''' bestand aus folgenden Personen<sup>1</sup>: |
| | {| class="wikitable" | | {| class="wikitable" |
| | ! Name | | ! Name |
| Zeile 193: |
Zeile 217: |
| | ! Rolle | | ! Rolle |
| | |- | | |- |
| − | | Mag. Dr. Stefan Sabutsch
| + | | Gabriel Kleinoscheg |
| − | | ELGA GmbH, HL7 Austria
| |
| − | | Autor, Herausgeber
| |
| − | |-
| |
| − | | Gabriel Kleinoscheg, MSc | |
| | | ELGA GmbH | | | ELGA GmbH |
| | | Autor | | | Autor |
| | |- | | |- |
| − | | DI Andrea Klostermann | + | | Andrea Klostermann |
| | | ELGA GmbH | | | ELGA GmbH |
| | | Autor | | | Autor |
| | |- | | |- |
| − | | DI Nikola Tanjga | + | | Stefan Sabutsch |
| | + | | ELGA GmbH, HL7 Austria |
| | + | | Autor, Herausgeber |
| | + | |- |
| | + | | Nikola Tanjga |
| | | ELGA GmbH | | | ELGA GmbH |
| | | Autor | | | Autor |
| | |- | | |- |
| − | | DI Oliver Kuttin | + | | Matthias Frohner |
| − | | ELGA GmbH | + | | FH Technikum Wien |
| | | Autor | | | Autor |
| | |} | | |} |
| | | | |
| | Unter Mitwirkung von: | | Unter Mitwirkung von: |
| − | Stephan Rainer-Sablatnig (ELGA GmbH), Nina Sjencic, B.A. (ELGA GmbH) | + | Oliver Kuttin (ELGA GmbH), Stephan Rainer-Sablatnig (ELGA GmbH), Nina Svec (ELGA GmbH) |
| | + | |
| | + | <sup>1</sup> Personen werden ohne Titel angegeben. |
| | + | |
| | ===Mitwirkende=== | | ===Mitwirkende=== |
| | '''Teilnehmer der Arbeitsgruppe Labor- und Mikrobiologiebefund'''<sup>1</sup> | | '''Teilnehmer der Arbeitsgruppe Labor- und Mikrobiologiebefund'''<sup>1</sup> |
| | | | |
| − | Christoph Arndt (Roche), Christoph Aspöck (Landesklinikum St. Pölten, Institut für Hygiene und Mikrobiologie), Jeroen de Bruin (Medexter Healthcare GmbH), Christa Freibauer (Institut für Pathologie LK Mistelbach; ÖGPath), Joseph Gappmayer (medilab GmbH), Milo Halabi (Institut für klinische Pathologie, Mikrobiologie und molekulare Diagnostik Krankenhaus der Barmherzigen Schwestern Ried; ÖGPath), Alexander Haushofer (ÖGLMKC; Klinikum Wels-Grieskirchen), Markus Hell (Medilab - Laboratorium Dr. Mustafa, Dr. Richter OG), Günter Igler (Analyse BioLab; KH Elisabethinen Linz), Heidrun Kerschner (Analyse BioLab; KH Elisabethinen Linz), Gabriel Kleinoscheg (ELGA GmbH), Andrea Klostermann (ELGA GmbH), Christoph Koidl (MedUni Graz, Institut für Hygiene, Mikrobiologie und Umweltmedizin; BFG Med. Mikrobiologie und Hygiene), Kristiane Kousek (NÖ Landesklinken Holding), Johannes Möst (Österreichische Gesellschaft für Hygiene, Mikrobiologie und Präventivmedizin), Johannes Möst (Österreichische Gesellschaft für Hygiene, Mikrobiologie und Präventivmedizin), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, BFG Med. u. Chem. Labordiagnostik, ÖGLMKC), Michael Nöhammer (ÖÄK), Tina Prager (NÖ Landesklinken Holding), Elisabeth Presterl (Universitätsklinik für Krankenhaushygiene & Infektionskontrolle, Medizinische Universität Wien), Stephan Rainer-Sablatnig (ELGA GmbH), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Ulrich Sagel (Universitätsklinik für Hygiene und Mikrobiologie), Tamara Savic (KAGes, Institut für Krankenhaushygiene und Mikrobiologie), Carina Seerainer (ELGA GmbH), Nina Sjencic (ELGA GmbH), Nikola Tanjga (ELGA GmbH), Gerhard Weigl (Inst. f. Labormedizin – Klinik Penzing, Wien), Birgit Willinger (AKH, MedUni Wien, Klin.Inst.f.Labormedizin)
| + | Christoph Aspöck (Klinisches Institut für Hygiene und Mikrobiologie, Universitätsklinikum St. Pölten - Lilienfeld), Jeroen S. de Bruin (docmetric GmbH), Gebhard Feierl (Diagnostik und Forschungsinstitut f. Hygiene, Mikrobiologie und Umweltmedizin, Medizinische Universität Graz), Christa Freibauer (Institut für Pathologie LK Mistelbach, ÖGPath/IAP Austria), Joseph Gappmayer (medilab - medizinisch-chemisches Labor Dr. Mustafa, Dr. Richter OG), Milo Halabi (Institut für klinische Pathologie, Mikrobiologie und molekulare Diagnostik, Krankenhaus der Barmherzigen Schwestern Ried), Alexander Haushofer (Institut für Medizinische und Chemische Labordiagnostik, Klinikum Wels-Grieskirchen GmbH), Markus Hell (Fachbereich/Abteilung für Klinische Mikrobiologie und Hygiene, medilab - medizinisch-chemisches Labor Dr. Mustafa, Dr. Richter OG), Günter Igler (analyse BioLab GmbH), Heidrun Kerschner (analyse BioLab GmbH), Gabriel Kleinoscheg (ELGA GmbH), Andrea Klostermann (ELGA GmbH), Christoph Koidl (Diagnostik und Forschungsinstitut f. Hygiene, Mikrobiologie und Umweltmedizin, Medizinische Universität Graz), Eva Leitner-Meyer (D&F Institut für Hygiene Mikrobiologie und Umweltmedizin, Medizinische Universität Graz), Michael Nöhammer (Österreichische Ärztekammer), Maximilian Ossana (Black Tusk GmbH), Tina Prager (Abteilung Med./Pfleg. Prozessmanagement, NÖ Landesgesundheitsagentur), Elisabeth Presterl (Universitätsklinik für Krankenhaushygiene und Infektionskontrolle, Medizinische Universität Wien), Stephan Rainer-Sablatnig (ELGA GmbH), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Ulrich Sagel (LADR Ihr Labor vor Ort), Carina Seerainer (FH JOANNEUM Gesellschaft mbH), Nina Svec (ELGA GmbH), Nikola Tanjga (ELGA GmbH), Gerhard Weigl (Inst. f. Labormedizin – Klinik Penzing, Wien), Birgit Willinger (Klinisches Institut für Labormedizin, Abteilung für Klinische Mikrobiologie, AKH Wien bzw. Medizinische Universität Wien) |
| − | | |
| − | [TODO fehlen noch welche? auch die erwähnen, die per E-Mail mitgeholfen haben (z.B. bei Value Sets)?]
| |
| | | | |
| | <sup>1</sup> Personen sind in alphabetischer Reihenfolge ohne Titel angegeben. | | <sup>1</sup> Personen sind in alphabetischer Reihenfolge ohne Titel angegeben. |
| Zeile 244: |
Zeile 269: |
| | '''Patronanz, Akkordierung, Ergänzungen, Zustimmung''' | | '''Patronanz, Akkordierung, Ergänzungen, Zustimmung''' |
| | | | |
| − | Clemens Auer (Bundesministerium für Gesundheit), Michael Danninger (Labene), Hubert Eisl (ELGA GmbH), Peter Fraunberger (Medizinisches Zentrallaboratorium GmbH), Josef Galler (Steiermärkische Krankenanstalten-ges.m.b.H.), Gerhard Gretzl (Solve Consulting), Ulrike Gruber-Mösenbacher (Landeskrankenhaus Feldkirch, Institut für Pathologie), Milo Halabi (Krankenhaus der Barmherzigen Schwestern Ried, Inst. f. Pathologie), Elisabeth Haschke-Becher (A.ö. Krankenhaus der Elisabethinen Linz, Institut für Medizinische und Chemische Labordiagnostik), Susanne Herbek (ELGA GmbH), Wolfgang Hiesl (Oö. Gesundheitsfonds / eHealth Management), Martin Hurch (ELGA GmbH), Stylianos Kapiotis (Med. Uni. Wien, Klinisches Institut für Medizinische und Chemische Labordiagnostik), Peter Konrath (B&S Zentrallabor), Oliver Kuttin (ELGA GmbH), Georg Lechleitner (Tilak, Abteilungsleiter Informationstechnologie/IT-Abteilung), Hubert Leitner (Steiermärkische Krankenanstalten-ges.m.b.H.), Helmut Lindorfer (Österreichische Agentur für Gesundheit und Ernährungssicherheit GmbH – AGES), Sabine Manhardt (Österreichische Ärztekammer, Sekretariat), Achim Mühlberger (GRZ IT Center Linz GmbH), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, Fachgesellschaft Labormedizin), Michael Nebel (Gibodat EDV- und Organisationsberatungs GmbH), Susan Netzl (AUVA - Unfallkrankenhaus Meidling, Labor), Claudia Perndl (A.ö. Krankenhaus der Elisabethinen Linz, Institut für Medizinische und Chemische Labordiagnostik, EDV), Sven Plattner (BKH Hall in Tirol, EDV), Thomas Pöckl (NÖ Landeskliniken-Holding), Angelika Reiner-Concin (Sozialmedizinisches Zentrum Ost – Donauspital, Pathologisch-Bakteriologisches Institut), Alexander Schanner (NÖ Landesklinikenholding), Gerhard Schobesberger (Österreichische Ärztekammer, Labor Schobesberger), Peter Schöttel (Bartelt GmbH), Christian Schweiger (Medizinische Universität Wien / AKH Wien, klinische Abteilung für Medizinisch-chemische Labordiagnostik), Herbert Stekel (AKH Linz, Institut für Laboratoriumsmedizin), Romana Thiel (HCS, Health Communication Service), Peter Uher (Telekom Austria), Michael Weidenauer (Assista), Thomas Wrba (Medizinische Universität Wien) | + | Clemens Auer (Bundesministerium für Gesundheit), Michael Danninger (Labene), Hubert Eisl (ELGA GmbH), Peter Fraunberger (Medizinisches Zentrallaboratorium GmbH), Josef Galler (Steiermärkische Krankenanstaltenges. m.b.H.), Gerhard Gretzl (Solve Consulting), Ulrike Gruber-Mösenbacher (Landeskrankenhaus Feldkirch, Institut für Pathologie), Milo Halabi (Krankenhaus der Barmherzigen Schwestern Ried, Inst. f. Pathologie), Elisabeth Haschke-Becher (A.ö. Krankenhaus der Elisabethinen Linz, Institut für Medizinische und Chemische Labordiagnostik), Susanne Herbek (ELGA GmbH), Wolfgang Hiesl (Oö. Gesundheitsfonds / eHealth Management), Martin Hurch (ELGA GmbH), Stylianos Kapiotis (Med. Uni. Wien, Klinisches Institut für Medizinische und Chemische Labordiagnostik), Peter Konrath (B&S Zentrallabor), Oliver Kuttin (ELGA GmbH), Georg Lechleitner (Tilak, Abteilungsleiter Informationstechnologie/IT-Abteilung), Hubert Leitner (Steiermärkische Krankenanstaltenges. m.b.H.), Helmut Lindorfer (Österreichische Agentur für Gesundheit und Ernährungssicherheit GmbH - AGES), Sabine Manhardt (Österreichische Ärztekammer, Sekretariat), Achim Mühlberger (GRZ IT Center Linz GmbH), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, Fachgesellschaft Labormedizin), Michael Nebel (Gibodat EDV- und Organisationsberatungs GmbH), Susan Netzl (AUVA - Unfallkrankenhaus Meidling, Labor), Claudia Perndl (A.ö. Krankenhaus der Elisabethinen Linz, Institut für Medizinische und Chemische Labordiagnostik, EDV), Sven Plattner (BKH Hall in Tirol, EDV), Thomas Pöckl (NÖ Landeskliniken-Holding), Angelika Reiner-Concin (Sozialmedizinisches Zentrum Ost – Donauspital, Pathologisch-Bakteriologisches Institut), Alexander Schanner (NÖ Landesklinikenholding), Gerhard Schobesberger (Österreichische Ärztekammer, Labor Schobesberger), Peter Schöttel (Bartelt GmbH), Christian Schweiger (Medizinische Universität Wien / AKH Wien, klinische Abteilung für Medizinisch-chemische Labordiagnostik), Herbert Stekel (AKH Linz, Institut für Laboratoriumsmedizin), Romana Thiel (HCS, Health Communication Service), Peter Uher (Telekom Austria), Michael Weidenauer (Assista), Thomas Wrba (Medizinische Universität Wien) |
| | | | |
| | ''Andere ELGA Arbeitsgruppen: '' | | ''Andere ELGA Arbeitsgruppen: '' |
| Zeile 256: |
Zeile 281: |
| | Die Änderungen für Version 2.06.2 wurde von der AG Laborbefund am 12.1.2017 abgenommen. Teilnehmer der AG Laborbefund waren: | | Die Änderungen für Version 2.06.2 wurde von der AG Laborbefund am 12.1.2017 abgenommen. Teilnehmer der AG Laborbefund waren: |
| | | | |
| − | Maria Abzieher (Wiener Krankenanstaltenverbund), Robert Alscher (Humanomed IT Solutions Gmbh), Daniel Außerdorfer (Univ.Klin. Innsbruck, Zentralinstitut für Medizinische und Chemische Labordiagnostik (ZIMCL)), René Berger (Synlab), Barbara Dall (CGM), Zeljko Drljaca (Institut für medizinische und chemische Labordiagnostik Gesellschaft m.b.H), Daniela Eisner (Salzburger Landeskliniken ), Christian Fersterer (Salzburger Landeskliniken), Matthias Frohner (FH Technikum Wien), Josef Galler (Steiermärkische Krankenanstalten-ges. m.b.H.), Andrea Griesmacher (Univ.Klin. Innsbruck, Zentralinstitut für Medizinische und Chemische Labordiagnostik (ZIMCL)), Gernot Gruber (Institut für medizinische und chemische Labordiagnostik Gesellschaft m.b.H), Sylvia Handler (biomed austria - Österreichischer Berufsverband der Biomedizinischen AnalytikerInnen), Susanne Hauptlorenz (Salzkammergut-Klinikum Vöcklabruck, Institut für Med.Chem. Labordiagnostik und Blutdepot), Alexander Haushofer (ÖGLMKC; Klinikum Wels-Grieskirchen), Wolfgang Hießl (Oö. Gesundheitsfonds / eHealth Management), Michael Hubmann (Med. Zentrallaboratorium GmbH), Günter Igler (Analyse BioLab; KH Elisabethinen Linz), Sonja Jansen-Skoupy (KAV Wien, SMZ Süd), Michael Krausenbaum (CGM LAB Deutschland GmbH), Herwig Loidl (Carecenter Software GmbH), Birgit Luxbacher (biomed austria - Österreichischer Berufsverband der Biomedizinischen AnalytikerInnen), Clemens M. Sampl (BMGF), Herbert Matzenberger (CompuGroup Medical CEE GmbH), Alexander Mense (Fachhochschule Technikum Wien, HL7 Austria), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, BFG Med. u. Chem. Labordiagnostik, ÖGLMKC), Stefan Mustafa (Labor Doz. Mag. DDr. Stefan Mustafa), Michael Nöhammer (Ärztekammer Österreich), Thomas Pöckl (NÖ Landeskliniken-Holding), Elisabeth Presterl (Universitätsklinik für Krankenhaushygiene & Infektionskontrolle, Medizinische Universität Wien), Sebastian Reimer (BMGF), Harald Rubey (LK Weinviertel Mistelbach, Laborinstitut, NÖ LK-Holding), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Ulrich Sagel (Universitätsklinik für Hygiene und Mikrobiologie), Karin Salzmann (Univ. Klinik für Innere Medizin Innsbruck), Stefan Sauermann (FH Technikum Wien, Interoperabilitätsforum Österreich), Peter Schöttel (Bartelt GmbH), Christian Schweiger (Medizinische Universität Wien / AKH Wien, klinische Abteilung für Medizinisch-chemische Labordiagnostik), Carina Seerainer (ELGA GmbH), Josef Seier (Klinikum Wels-Grieskirchen), Wolfgang Sischka (Assista Laborelectronics GmbH), Herbert Stekel (Kepler Universitätsklinikum GmbH), Michael Svizak (AUVA Hauptstelle - Ärztliche Direktion), Gerhard Weigl (Inst. f. Labormedizin – Otto-Wagner-Spital Wien) | + | Maria Abzieher (Wiener Krankenanstaltenverbund), Robert Alscher (Humanomed IT Solutions Gmbh), Daniel Außerdorfer (Univ.Klin. Innsbruck, Zentralinstitut für Medizinische und Chemische Labordiagnostik (ZIMCL)), René Berger (Synlab), Barbara Dall (CGM), Zeljko Drljaca (Institut für medizinische und chemische Labordiagnostik Gesellschaft m.b.H), Daniela Eisner (Salzburger Landeskliniken ), Christian Fersterer (Salzburger Landeskliniken), Matthias Frohner (FH Technikum Wien), Josef Galler (Steiermärkische Krankenanstaltenges. m.b.H.), Andrea Griesmacher (Univ.Klin. Innsbruck, Zentralinstitut für Medizinische und Chemische Labordiagnostik (ZIMCL)), Gernot Gruber (Institut für medizinische und chemische Labordiagnostik Gesellschaft m.b.H), Sylvia Handler (biomed austria - Österreichischer Berufsverband der Biomedizinischen AnalytikerInnen), Susanne Hauptlorenz (Salzkammergut-Klinikum Vöcklabruck, Institut für Med.Chem. Labordiagnostik und Blutdepot), Alexander Haushofer (ÖGLMKC; Klinikum Wels-Grieskirchen), Wolfgang Hießl (Oö. Gesundheitsfonds / eHealth Management), Michael Hubmann (Med. Zentrallaboratorium GmbH), Günter Igler (Analyse BioLab; KH Elisabethinen Linz), Sonja Jansen-Skoupy (KAV Wien, SMZ Süd), Michael Krausenbaum (CGM LAB Deutschland GmbH), Herwig Loidl (Carecenter Software GmbH), Birgit Luxbacher (biomed austria - Österreichischer Berufsverband der Biomedizinischen AnalytikerInnen), Herbert Matzenberger (CompuGroup Medical CEE GmbH), Alexander Mense (Fachhochschule Technikum Wien, HL7 Austria), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, BFG Med. u. Chem. Labordiagnostik, ÖGLMKC), Stefan Mustafa (Labor Doz. Mag. DDr. Stefan Mustafa), Michael Nöhammer (Ärztekammer Österreich), Thomas Pöckl (NÖ Landeskliniken-Holding), Elisabeth Presterl (Universitätsklinik für Krankenhaushygiene & Infektionskontrolle, Medizinische Universität Wien), Sebastian Reimer (BMGF), Harald Rubey (LK Weinviertel Mistelbach, Laborinstitut, NÖ LK-Holding), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Ulrich Sagel (Universitätsklinik für Hygiene und Mikrobiologie), Karin Salzmann (Univ. Klinik für Innere Medizin Innsbruck), Clemens M. Sampl (BMGF), Stefan Sauermann (FH Technikum Wien, Interoperabilitätsforum Österreich), Peter Schöttel (Bartelt GmbH), Christian Schweiger (Medizinische Universität Wien / AKH Wien, klinische Abteilung für Medizinisch-chemische Labordiagnostik), Carina Seerainer (ELGA GmbH), Josef Seier (Klinikum Wels-Grieskirchen), Wolfgang Sischka (Assista Laborelectronics GmbH), Herbert Stekel (Kepler Universitätsklinikum GmbH), Michael Svizak (AUVA Hauptstelle - Ärztliche Direktion), Gerhard Weigl (Inst. f. Labormedizin – Otto-Wagner-Spital Wien) |
| | | | |
| | <sup>2</sup> Personen sind in alphabetischer Reihenfolge ohne Titel angegeben. | | <sup>2</sup> Personen sind in alphabetischer Reihenfolge ohne Titel angegeben. |
| Zeile 262: |
Zeile 287: |
| | | | |
| | =Technischer Hintergrund= | | =Technischer Hintergrund= |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Der technische Hintergrund soll im [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Technischer_Hintergrund|allgemeinen Leitfaden]] nachgelesen werden. |
| − | Der technische Hintergrund soll im [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Technischer_Hintergrund|allgemeinen Leitfaden]] nachgelesen werden. | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| | =Allgemeine Richtlinien für ELGA CDA-Implementierungsleitfäden= | | =Allgemeine Richtlinien für ELGA CDA-Implementierungsleitfäden= |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Die [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Allgemeine_Richtlinien_f.C3.BCr_CDA-Implementierungsleitf.C3.A4den|allgemeinen Richtlinien für CDA-Implementierungsleitfäden]] sollen beachtet werden. |
| − | Die [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Allgemeine_Richtlinien_f.C3.BCr_CDA-Implementierungsleitf.C3.A4den|allgemeinen Richtlinien für CDA-Implementierungsleitfäden]] sollen beachtet werden. | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| − | =Konformitätsprüfung=
| |
| − | Ein zu diesem Implementierungsleitfaden konformes CDA-Dokument ist zunächst ein valides CDA Release 2.0 XML-Dokument mit [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#CDA_Header|Header]] und [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#CDA_Body|Body]]. Darüber hinaus erfüllt es alle in diesem Leitfaden festgelegten "Geschäftsregeln".
| |
| | | | |
| − | Dies spiegelt ein generelles Konzept im Umgang mit Dokumenten wieder: die Validierung in zwei Schritten. Im ersten Schritt stellt dies die Validierung gegen zugehörige '''W3C Schemas''' dar. Das verwendete Schema ist das geringfügig erweiterte offizielle CDA Release 2.0 Schema (siehe [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schema-Pr.C3.BCfung|Schema-Prüfung]]). Darüber hinaus existieren eine Reihe von '''Schematron''' Regeln, die für einen zweiten Validierungsschritt genutzt werden und letztlich die Detailregelungen in diesem Leitfaden wiedergeben, sowie die Einhaltung der Geschäftsregeln (Optionalität, Kardinalität/Multiplizität, Datentypen, Wertebereiche, Abhängigkeiten) sicherstellen (siehe [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schematron-Pr.C3.BCfung|Schematron-Prüfung]]). Geschäftsregeln für Abschnitte oder Elemente werden auch technisch zu '''"Templates"''' zusammengefasst. Eine XML-Instanz, die kein valides CDA-Dokument ist oder sich nicht gegen das XSD-Schema validieren lässt oder im Widerspruch zu den angegebenen Geschäftsregeln steht, ist kein gültiges CDA-Dokument im Sinne dieses Implementierungsleitfadens.
| + | =Funktionale Anforderungen= |
| − | {{BeginYellowBox}}
| |
| − | Hinweis: Nicht alle Geschäftsregeln können mit Schema oder Schematron geprüft werden (etwa Inhalte von Multimedia-Attachments, Dokumentengröße). Zusätzliche Validierungsschritte sind gegebenenfalls notwendig, um alle Regeln zu überprüfen zu können.
| |
| − | {{EndYellowBox}}
| |
| − | {{BeginILFBox}}
| |
| − | Die Kapitel zu den technischen Konformitätsprüfungen von CDA-Dokumenten, gemäß diesem Dokumentleitfadens mittels Schema und Schematron, sind im allgemeinen Leitfaden unter den folgenden Links zu finden:
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schema-Pr.C3.BCfung|Schema-Prüfung]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schematron-Pr.C3.BCfung|Schematron-Prüfung]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Online-Validation_von_CDA-Dokumenten|Online-Validation von CDA-Dokumenten]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Hinweise_zur_Konformit.C3.A4tspr.C3.BCfung|Hinweise zur Konformitätsprüfung]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Abnahmepr.C3.BCfung_f.C3.BCr_ELGA_e-Befunde|Abnahmeprüfung für ELGA e-Befunde]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Zertifizierung|Zertifizierung]]
| |
| − | {{EndILFBox}}
| |
| | | | |
| − | =Datentypen= | + | ==Voraussetzungen für den Zugriff auf e-Befunde in ELGA== |
| − | {{BeginILFBox}}
| + | Der ELGA GDA ist in ELGA angemeldet, berechtigt und besitzt eine gültige Kontaktbestätigung für den Patienten. |
| − | Im Kapitel [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Datentypen|Datentypen des allgemeinen Leitfadens]] werden nur die Datentypen beschrieben, die in ELGA CDA-Dokumenten wie diesem zur Anwendung kommen. Für weiterführende Informationen wird auf den zugrundeliegenden Standard Health Level Seven Version 3 (V3), Normative Edition verwiesen.
| + | Der Patient ist ELGA-Teilnehmer und hat keinen Widerspruch hinsichtlich ELGA eingelegt. |
| − | {{EndILFBox}}
| |
| | | | |
| − | =Vorgaben zum medizinischen Inhalt= | + | ==Anwendungsfälle des Dokumentenmanagements== |
| − | ==Labor- und Mikrobiologiebefund==
| + | Die folgenden Kapitel aus dem allgemeinen Leitfaden stellen eine Zusammenfassung der Inhalte der ELGA-Gesamtarchitektur, des Leitfadens XDS Metadaten und Usability Styleguides zum Thema e-Befunde dar. Detailinformationen sind in den entsprechenden Dokumenten nachzulesen (verfügbar auf der Homepage der [https://www.elga.gv.at/ ELGA GmbH]). Die wesentlichen Anwendungsfälle sind |
| − | Die inhaltliche Definition beruht auf den Mindestvorgaben der österreichischen Gesellschaft für Labormedizin und klinischen Chemie (ÖGLMKC) und wurden weiter verfeinert. Die folgende Tabelle zeigt einen Überblick über die inhaltlich abzubildenden medizinisch relevanten Daten. | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schreiben_und_Einbringen_von_Dokumenten|Schreiben und Einbringen von Dokumenten]] |
| − | | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Versionierung_von_Dokumenten|Versionierung von Dokumenten]] |
| − | {| class="wikitable" width="100%"
| + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Stornierung_von_Dokumenten|Stornierung von Dokumenten]] |
| − | |-
| + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Filtern_und_Suchen_von_Dokumenten|Filtern und Suchen von Dokumenten]] |
| − | ! style="text-align:left" width="20%" | Feld
| + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Lesen_von_ELGA_Dokumenten|Lesen von Dokumenten]] |
| − | ! style="text-align:left" width="40%" | Beschreibung
| |
| − | ! style="text-align:left" width="20%" | Bereich
| |
| − | |-
| |
| − | ! style="text-align:left" colspan="3" | ''Allgemeine Befundinformationen''
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Zeitpunkt der Auftragserfassung
| |
| − | | Datum und Zeitpunkt, an dem das analysierende Labor die Anforderung vom Zuweiser in der Labor-EDV erfasst hat.
| |
| − | | Header
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Auftragsdiagnose (Zuweiserdiagnose) bzw. Überweisungsgrund
| |
| − | | Vom Auftraggeber bestimmte und dem Labor übermittelte Verdachtsdiagnose bzw. der Überweisungsgrund
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Angeforderte Untersuchung bzw. Fragestellung
| |
| − | | Vom Auftraggeber angeforderte Untersuchungen, das angeforderte Analysespektrum bzw. die Fragestellung
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Befundtext
| |
| − | | Kommentar zum gesamten Befund
| |
| − | | Body
| |
| − | |-
| |
| − | ! style="text-align:left" colspan="3" | ''Spezimeninformation''
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Zeitpunkt der Spezimengewinnung
| |
| − | | Damit ist jenes Datum und Zeitpunkt gemeint, an dem das Spezimen zur Analyse gewonnen wurde. Die Dokumentation des Zeitpunkts der Spezimengewinnung ist in der Verantwortung der entnehmenden Person, die in vielen Fällen mit dem Befundersteller nicht identisch ist, weil meist Spezimen zur Analyse an Labors versendet werden. Daher ist der Zeitpunkt vielfach im Labor nicht feststellbar.
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Zeitpunkt des Einlangens des Spezimen
| |
| − | | Datum und Zeit der Probenannahme im Labor
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Art des Spezimens (Specimen Type)
| |
| − | | Art der Probe (=Materialart)
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Entnahmeort
| |
| − | | Angabe der Körperstelle, von der das Spezimen stammt
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Entnahmeart und Entnahmegerät (SpecimenCollectionProcedure) [TODO https://jira-neu.elga.gv.at/browse/ELGA-1124]
| |
| − | | Art der Gewinnung
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Specimen ID | |
| − | | Eindeutige Nummer des Spezimen
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Entnehmende Person (Performer)
| |
| − | | Person, welche die Entnahme der Probe durchgeführt hat
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Kommentar zum Spezimen | |
| − | | Präanalytik pro Spezimen zur Spezimenqualität
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Allgemeine Anmerkungen des Labors zur Spezimenqualität | |
| − | | Textinformationen zur Spezimenqualität
| |
| − | | Body
| |
| − | |-
| |
| − | ! style="text-align:left" colspan="3" | ''Allgemeine Laborergebnisse''
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Gruppierung / Befundgruppen (Organizer)
| |
| − | | Analysengruppierung
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | ID des Tests | |
| − | | Eindeutige Codierung des Tests
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Analysenbezeichnung
| |
| − | | Bezeichnung der Analyse
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Ergebnis
| |
| − | |
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Einheit
| |
| − | |
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Referenzbereiche
| |
| − | | Für die Beurteilung relevante Referenzwerte.
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Befundinterpretation
| |
| − | | Codierte Bewertung des Ergebnisses
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Ergebniskommentar
| |
| − | | Kommentar des Labors zu einem einzelnen Testergebnis
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Externes Labor
| |
| − | | Kennzeichen ob ein Ergebnis extern ermittelt wurde
| |
| − | | Body
| |
| − | |-
| |
| − | ! style="text-align:left" colspan="3" | ''Kulturergebnisse''
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Erreger<sup>1</sup> (Isolat)
| |
| − | | Der durch eine Kulturmethode ermittelte Erreger.
| |
| − | | Body
| |
| − | |- style="background:#FFFFFF"
| |
| − | | Antibiogramm (inklusive MHK)
| |
| − | | Ergebnis der Bestimmung der Empfindlichkeit bzw. Resistenz von Erregern gegenüber Antibiotika. Anlassbezogen wird das Ergebnis interpretiert (R, S, I) und/oder die minimale Hemmkonzentration (MHK) angegeben. | |
| − | | Body
| |
| − | |}
| |
| − | <ref group="Tabelle">Im Labor- und Mikrobiologiebefund abzubildende medizinische Daten</ref>
| |
| − | | |
| − | <sup>1</sup> Erreger oder Krankheitserreger sind Stoffe oder Organismen, die in anderen Organismen potenziell gesundheitsschädigende Abläufe verursachen können.
| |
| − | | |
| − | ==Allgemeiner Laborbefund==
| |
| | | | |
| − | ===Befundbereiche (Laboratory Specialty Section)=== | + | ==Verwendung in der ELGA Infrastruktur== |
| | | | |
| − | Der Laborbefund kann mehrere unterschiedliche Teilbefunde aus verschiedenen Bereichen im Body des Dokumentes beinhalten (z.B. Hämatologie oder Urindiagnostik oder beide Arten gemeinsam). Das heißt, diese Teilbefunde bilden die erste Gliederungsebene des Bodys - die "Befundbereiche" oder - in Anlehnung an die Definitionen der "IHE" - "Laboratory Specialty Section" (vgl. [TODO Referenz]). Folgende Grafik zeigt die mögliche Gliederung auf der ersten Ebene innerhalb des Bodys.
| + | ===Vorgaben zu Dokumenten-Metadaten (XDS-Metadaten)=== |
| | + | Im Folgenden werden spezifische Anforderungen für die Generierung der XDS-Metadaten dargestellt. Die allgemein gültigen Regeln für die Erstellung der XDS-Metadaten sind im "Implementierungsleitfaden XDS Metadaten" (in der jeweils gültigen Version) auf der ELGA Homepage ([https://www.elga.gv.at/technischer-hintergrund/technische-elga-leitfaeden/ www.elga.gv.at]) abrufbar. |
| | | | |
| − | [[Datei:Gliederung nach Bereiche.png|thumb|none|300px|<ref group="Abbildung">Gliederung nach Befundbereichen/Laboratory Specialties</ref>: Gliederung nach Befundbereichen/Laboratory Specialties]]
| + | Spezielle Anforderungen an das Metadaten-Mapping ergeben sich für das XDS-Metadaten-Element "'''''eventCodeList'''''". Dieses Element basiert auf dem CDA "serviceEvent"-Element und übernimmt die durchgeführten medizinischen Leistungen in die Metadaten. |
| | + | Im Kontext dieses Leitfadens wird in die ''eventCodeList'' folgende Information übernommen: |
| | + | # die im Dokument enthaltenen Sections und |
| | + | # die Codierungsgrade der einzelnen Sections. |
| | | | |
| − | Die derzeit für den Laborbefund definierten Befundbereiche werden im Rahmen des hierarchisch organisierten Value Sets "ELGA_Laborstruktur" definiert, wobei für Befundbereiche nur Einträge der Ebene 1 verwendet werden dürfen. Die folgende Tabelle gibt einen auszugsweisen Überblick über die derzeit festgelegten Befundbereiche. Bei der Verwendung der Befundbereiche ist die Reihenfolge gemäß Value Set verpflichtend einzuhalten. Die Anwendung der Befundbereiche ist optional, denn es können auch Untersuchungen verschiedener Befundbereiche in einer einzelnen "Laboratory Specialty Section" unter dem Befundbereich "Allgemeiner Laborbefund" (Ebene 0 in Value Set "ELGA_Laborstruktur") zusammengefasst werden.
| + | Das bedeutet, dass im Zuge einer Dokumentensuche erkannt werden kann, welche Inhalte ein Labor- oder Mikrobiologiebefund enthält sowie die Information, ob bzw. welche Inhalte maschinenlesbar vorliegen. Damit kann ein abrufendes System erkennen, welche Daten in codierter Form aus dem Labor- oder Mikrobiologiebefund übernommen werden können. <br> |
| − | {{BeginYellowBox}}
| + | Um dies zu erreichen, '''MUSS''' in den CDA "serviceEvent"-Elementen neben dem "code"-Element auch zwingend die ID des "serviceEvent" angegeben werden: |
| − | [TODO (GKL) Ist dieser Text obsolet, weil es nur mehr Full Support gibt?]
| + | * Das "code"-Element MUSS das "code"-Element der Section wiedergeben und |
| | + | * die ID des "serviceEvent"-Elements MUSS die OID des "templateId"-Elements der Section wiedergeben. |
| | + | * Diese Regel MUSS für jede Section wiederholt werden. Das heißt, enthält das CDA-Dokument vier Sections, so MÜSSEN im CDA-Header vier "documentationOf/serviceEvent"-Elemente enthalten sein. Diese Regel gilt nicht für die Sections "Brieftext" und "Abschließende Bemerkungen". |
| | | | |
| − | Für EIS "Enhanced" ist die Codierung der Bereiche (als unterschiedliche section-Elemente) zwingend vorgeschrieben.
| |
| − | {{EndYellowBox}}
| |
| | {| class="wikitable" | | {| class="wikitable" |
| − | ! Code | + | ! XDS-Mapping |
| − | ! Befundbereich (Laboratory Speciality Section) | + | ! Optionalität |
| | + | ! CDA-Element |
| | + | ! Mapping Vorschrift |
| | |- | | |- |
| − | | 100 | + | | eventCodeList |
| − | | Blutgruppenserologie | + | | R |
| − | |- | + | | ClinicalDocument/documentationOf/serviceEvent/id und ClincialDocument/documentationOf/serviceEvent/code |
| − | | 200
| + | | der "serviceEvent"-Code wird mit der "serviceEvent"-ID verknüpft |
| − | | Blutgasanalytik
| + | |} |
| − | |-
| |
| − | | 300
| |
| − | | Hämatologie
| |
| − | |-
| |
| − | | 400
| |
| − | | Gerinnung/Hämostaseologie
| |
| − | |-
| |
| − | | …
| |
| − | | …
| |
| − | |}<ref group="Tabelle">Liste der Befundbereiche, auszugsweise gem. ELGA Value Set "ELGA_Laborstruktur", die sich auch im ELGA Value Set "ELGA_Laborparameter" widerspiegeln.</ref>: ''Liste der Befundbereiche, auszugsweise gem. ELGA Value Set "ELGA_Laborstruktur", die sich auch im ELGA Value Set "ELGA_Laborparameter" widerspiegeln.'' | |
| − | | |
| − | ===Befundgruppen===
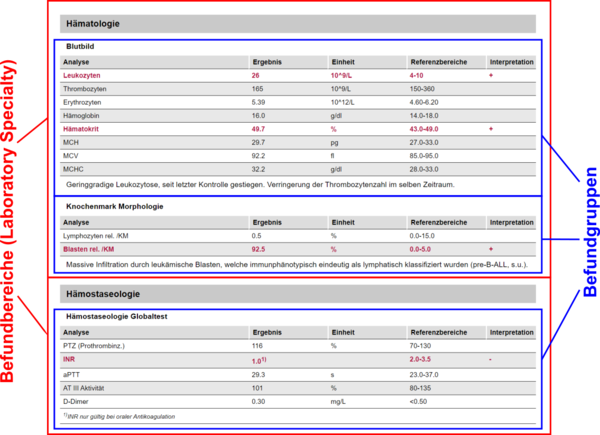
| |
| − | Innerhalb der Befundbereiche erfolgt in der Regel eine Strukturierung und Gliederung der Ergebnisse zur besseren Lesbarkeit und Auffindbarkeit in "Befundgruppen". Die dritte Ebene des ELGA Value Sets "ELGA_Laborstruktur" definiert zulässige Befundgruppen. Grundsätzlich ist die Reihenfolge der Befundgruppen gemäß Value Set verpflichtend einzuhalten. Es besteht jedoch auch die Möglichkeit, Ergebnisse ohne Befundgruppenstrukturierung zu übermitteln.
| |
| | | | |
| − | [[Datei:Strukturierungsmöglichkeiten Body.png|thumb|none|600px|<ref group="Abbildung">Strukturierungsmöglichkeiten im Laborbefund im Body</ref>: Strukturierungsmöglichkeiten im Laborbefund im Body]]
| + | Daher ergeben sich folgende Vorschriften für das Mapping von CDA-Element zu XDS-Metadaten: |
| | | | |
| − | Der folgende Ausschnitt aus einem Laborbefunden enthält die Befundbereiche "Hämatologie" und "Hämostaseologie" sowie die dazugehörigen Befundgruppen "Blutbild", "Knochenmark Morphologie" bzw. "Hämostaseologie Globaltest".
| + | '''$code''' = concat(ClincialDocument/documentationOf/serviceEvent/code[@code],"^",ClinicalDocument/documentationOf/serviceEvent/id[@root]) |
| | | | |
| − | [[Datei:Ausschnitt Beispielbefund.png|thumb|none|600px|<ref group="Abbildung">Ausschnitt aus einem Laborbefund</ref>: Ausschnitt aus einem Laborbefund]]
| + | '''$displayName''' = ClincialDocument/documentationOf/serviceEvent/code[@displayName] |
| | | | |
| − | ===Harmonisierung des Befundaufbaus – Value Set "ELGA_Laborparameter"=== | + | '''$codeSystem''' = ClincialDocument/documentationOf/serviceEvent/code[@codeSystem] |
| | | | |
| − | Im Rahmen der Arbeiten zum vorliegenden Dokument wurde in der Expertengruppe die grundsätzliche Übereinkunft getroffen, auch die Befundgruppen und die damit verbundene Testzuordnung entsprechend österreichweit abzustimmen. Die Strukturierung eines Laborbefundes wurde in Form des hierarchischen Value Sets '''"ELGA_Laborparameter"''' festgelegt.
| + | Dabei muss das Mapping für jedes serviceEvent einzeln durchgeführt und ein entsprechender rim:Classification-Block erstellt werden. |
| − | {{BeginYellowBox}}
| + | <pre class="ilfbox_code"> |
| − | Strukturierung, Reihenfolge der Parameter sowie die Bezeichnung der Parameter sind durch das Value Set "ELGA_Laborparameter" verpflichtend vorgegeben!
| + | <rim:Classification |
| − | {{EndYellowBox}}
| + | classificationScheme="urn:uuid:2c6b8cb7-8b2a-4051-b291-b1ae6a575ef" |
| − | Eine Hilfestellung zum Mapping der lokalen Codes auf die vorgeschriebenen Codes des Value Sets bietet der "Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund"<ref>Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund [https://www.elga.gv.at/fileadmin/user_upload/Dokumente_PDF_MP4/CDA/Leitfaden_LOINC_Anwendung_in_ELGA_V1.03.pdf]</ref>.
| + | classifiedObject="theDocument" |
| | + | nodeRepresentation="$code"> |
| | + | <rim:Name> |
| | + | <rim:LocalizedString value="$displayName"/> |
| | + | </rim:Name> |
| | + | <rim:Slot name="codingScheme"> |
| | + | <rim:ValueList> |
| | + | <rim:Value>urn:oid:$codeSystem</rim:Value> |
| | + | </rim:ValueList> |
| | + | </rim:Slot> |
| | + | </rim:Classification> |
| | + | </pre> |
| | | | |
| − | ==Mikrobiologiebefund==
| + | <div class="landscape"> |
| − | | |
| − | Unter den Analysen des Laborbefundes finden sich viele aus dem Bereich der Mikrobiologie. Grundsätzlich können alle Analysen auch in der "klassischen" Struktur des Laborbefundes dargestellt werden (siehe [[#Laborbefund|Laborbefund]]). Allerdings entspricht die Struktur des Laborbefundes nicht dem eines üblichen Mikrobiologiebefundes, der einem bestimmten Muster folgt, welches den Untersuchungsverlauf widerspiegelt:
| |
| − | | |
| − | # Beschreibung des entnommenen Materials (z.B. Mittelstrahlharn) inklusive makroskopischer Beurteilung
| |
| − | # Mikroskopische Untersuchung des Materials (z.B. Erythrozyten, Leukozyten, grampositive Bakterien)
| |
| − | # Kultureller Erregernachweis
| |
| − | #* Benennung der Reinkulturen (Isolate) mit Nennung der taxonomischen Bestimmung der Mikroorganismen (z.B. Streptococcus pyogenes) ggf. mit Angabe des Serovars/Pathovars.
| |
| − | #* Angabe des Antibiogramms mit Interpretation (R, S, I) und/oder minimaler Hemmkonzentration (MHK)
| |
| − | # Molekulare Erregernachweise
| |
| − | # Infektionsserologie
| |
| − | | |
| − | Die Struktur des Mikrobiologiebefundes wird durch "Laboratory Specialty Sections", die jeweils mit einem fixen Code versehen sind, vorgegeben. Details dazu sind dem Document Level Template des Mikrobiologiebefundes zu entnehmen.
| |
| − | | |
| − | [TODO vermutlich nicht mehr notwendig] [[Datei:Mikrobiologischer Befund.jpg||500px]]
| |
| − | | |
| − | [TODO überarbeiten] [[Datei:Ausschnitt Bakteriologie Beispielbefund.png|600px|Ausschnitt Bakteriologie Beispielbefund.]]
| |
| − | | |
| − | ==Hinweise für akkreditierte Laboratorien gem. ISO 15189:2012==
| |
| − | | |
| − | TODO (GKL): Aktuelle Version ist ISO 15189:2014 - ausbessern? passen Kapitel in Nummer/Überschrift dann noch zusammen?
| |
| − | | |
| − | Die Vorgaben dieses Implementierungsleitfadens erlauben die Erzeugung von Befunden gemäß ISO 15189:2012 "Medizinische Laboratorien - Anforderungen an die Qualität und Kompetenz"<ref>ISO 15189:2012 "Medizinische Laboratorien - Anforderungen an die Qualität und Kompetenz"</ref>, speziell in Hinblick auf die dort angegebenen Kapitel 5.8 (Befundberichte) und 5.9 (Freigabe der Ergebnisse). Für akkreditierte Laboratorien sind neben der ISO 15189:2012 folgende Hinweise zu beachten:
| |
| − | | |
| − | ===Angabe des Akkreditierungs-Logos===
| |
| − | | |
| − | Das Akkreditierungs-Logo kann neben dem Logo des Labors angegeben werden. Technisch erfolgt das über die Sektion "Brieftext", das Logo ist ggf. gemeinsam mit dem Logo des Labors in einer Grafikdatei anzugeben (siehe [[#Brieftext|Brieftext]]).
| |
| − | | |
| − | TODO (GKL) ggf. noch Screenshot mit aktuellem Stylesheet
| |
| − | [[Datei:Angabe des Akkreditierungs-Logos im Briefkopf.png|thumb|none|500px|<ref group="Abbildung">Angabe des Akkreditierungs-Logos im Brieftext</ref>: Angabe des Akkreditierungs-Logos im Brieftext]]
| |
| − | | |
| − | Ein Labor muss nicht zwingend für alle Analysen, die es durchführen kann, akkreditiert sein. Das Akkreditierungs-Logo kann angegeben werden, sobald es akkreditierte Analysen gibt; wenn "nicht akkreditierte Analysen" am Befund erscheinen, soll bei diesen angegeben werden können, dass das Labor für diese Analyse nicht akkreditiert ist. Für die entsprechend Markierung wird die Verwendung von Anmerkungszeichen (wie z.B. *) und Endnoten empfohlen.
| |
| − | | |
| − | ===Angabe des Untersuchungs- bzw. Messverfahrens===
| |
| − | | |
| − | Wenn der Name der Analysen keinen Rückschluss auf die Methode erlaubt, aber Untersuchungs- bzw. Messverfahren dennoch angegeben werden sollen (ISO 15189:2012, Vorgabe 5.8.3 a)), soll dies als Kommentar zur Analyse erfolgen (siehe TODO Link/Kapitel auflösen: Bemerkungen_zu_Analysen_oder_Analyseergebnissen|Bemerkungen zu Analysen oder Analyseergebnissen).
| |
| − | | |
| − | ===Vorgaben für den Befunddruck ===
| |
| − | | |
| − | Einige Vorgaben von ISO 15189:2012 beziehen sich auf Qualitätsmerkmale für gedruckte Befunde (Vorgabe 5.8.3 d) "Identifizierung des Patienten und den Aufenthaltsort des Patienten auf jeder Seite" und Vorgabe 5.8.3 p) "Seitenzahl zur Gesamtzahl der Seiten"). Dieser Leitfaden definiert ein elektronisches Format, das diese Anforderungen grundsätzlich unterstützt. Am ELGA-Portal (Zugriff für Bürger) werden Tools zur Darstellung eingesetzt, die diese Vorgaben unterstützen. Diese Tools werden auch von der ELGA GmbH zum Download bereitgestellt (Referenzstylesheet, CDA2PDF auf http://www.elga.gv.at/CDA).
| |
| − | | |
| − | =Funktionale Anforderungen=
| |
| − | | |
| − | ==Voraussetzungen für den Zugriff auf e-Befunde in ELGA==
| |
| − | Der ELGA GDA ist in ELGA angemeldet, berechtigt und besitzt eine gültige Kontaktbestätigung für den Patienten.
| |
| − | Der Patient ist ELGA-Teilnehmer und hat keinen generellen, partiellen oder situativen Widerspruch hinsichtlich ELGA eingelegt.
| |
| − | | |
| − | ==Anwendungsfälle des Dokumentenmanagements==
| |
| − | Die folgenden Kapiteln aus dem allgemeinen Leitfaden stellen eine Zusammenfassung der Inhalte der ELGA-Gesamtarchitektur, des Leitfadens XDS Metadaten und Usability Styleguides zum Thema e-Befunde dar. Detailinformationen sind in den entsprechenden Dokumenten nachzulesen (verfügbar auf der Homepage der [https://www.elga.gv.at/ ELGA GmbH]). Die wesentlichen Anwendungsfälle sind
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schreiben_und_Einbringen_von_Dokumenten|Schreiben und Einbringen von Dokumenten]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Versionierung_von_Dokumenten|Versionierung von Dokumenten]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Stornierung_von_Dokumenten|Stornierung von Dokumenten]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Filtern_und_Suchen_von_Dokumenten|Filtern und Suchen von Dokumenten]]
| |
| − | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Lesen_von_ELGA_Dokumenten|Lesen von Dokumenten]]
| |
| | | | |
| − | <div class="landscape">
| |
| | ===Dokument-Metadaten (XDS-Metadaten)=== | | ===Dokument-Metadaten (XDS-Metadaten)=== |
| | {| class="wikitable" | | {| class="wikitable" |
| Zeile 521: |
Zeile 367: |
| | ! Optionalität im XDS-Leitfaden | | ! Optionalität im XDS-Leitfaden |
| | ! CDA-Element in /ClinicalDocument | | ! CDA-Element in /ClinicalDocument |
| − | ! Werte ''mit Beispielen'' | + | ! Beispiele bzw. fixe Werte (fett) |
| | ! Erklärung | | ! Erklärung |
| | |- | | |- |
| Zeile 528: |
Zeile 374: |
| | | rowspan="2"|./id | | | rowspan="2"|./id |
| | | | | | |
| − | *@root="''1.2.40.0.34.3.1.1058.1337.999021.1''" | + | *@root="1.2.40.0.34.3.1.1058.1337.999021.1" |
| − | | rowspan="2"| Das uniqueId Element beschreibt den global eindeutigen Identifier des Dokuments und kann mit oder ohne Extension angegeben werden. | + | | rowspan="2"| Das "uniqueId"-Element beschreibt den global eindeutigen Identifier des Dokuments und kann mit oder ohne Extension angegeben werden. |
| | |- | | |- |
| | | | | | |
| − | *@root="''1.2.40.0.34.3.1.1058.1337''" | + | *@root="1.2.40.0.34.3.1.1058.1337" |
| − | *@extension="''999021.1''" | + | *@extension="999021.1" |
| | |- | | |- |
| | | rowspan="2" colspan="2"| [[ILF:XDS_Metadaten_(Version_3)#typeCode_.28und_typeCodeDisplayName.29_2|typeCode]] | | | rowspan="2" colspan="2"| [[ILF:XDS_Metadaten_(Version_3)#typeCode_.28und_typeCodeDisplayName.29_2|typeCode]] |
| Zeile 539: |
Zeile 385: |
| | | rowspan="2"| ./code | | | rowspan="2"| ./code |
| | | | | | |
| − | *@code="11502-2" | + | *@code="'''11502-2'''" |
| − | *@displayName="Laboratory report" | + | *@displayName="'''Laboratory report'''" |
| − | *@codeSystem="2.16.840.1.113883.6.1" | + | *@codeSystem="'''2.16.840.1.113883.6.1'''" |
| − | | Zur Unterscheidung des "Dokumenttyps" erhält das "code"-Elementes des '''"Laborbefundes"''' ein vorgegebenes "code"-Element. | + | | Zur Unterscheidung des "Dokumenttyps" erhält das "code"-Element des '''Laborbefundes''' einen vorgegebenen Wert. |
| | |- | | |- |
| | | | | | |
| − | *@code="18725-2" | + | *@code="'''18725-2'''" |
| − | *@displayName="Microbiology studies" | + | *@displayName="'''Microbiology studies (set)'''" |
| − | *@codeSystem="2.16.840.1.113883.6.1" | + | *@codeSystem="'''2.16.840.1.113883.6.1'''" |
| − | | Zur Unterscheidung des "Dokumenttyps" erhält das "code"-Elementes des '''"Mikrobiologiebefundes"''' ein vorgegebenes "code"-Element. | + | | Zur Unterscheidung des "Dokumenttyps" erhält das "code"-Element des '''Mikrobiologiebefundes''' einen vorgegebenen Wert. |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#classCode_.28und_classCodeDisplayName.29_2|classCode]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#classCode_.28und_classCodeDisplayName.29_2|classCode]] |
| Zeile 554: |
Zeile 400: |
| | | ./code/translation | | | ./code/translation |
| | | | | | |
| − | *@code="11502-2" | + | *@code="'''11502-2'''" |
| − | *@displayName="Laboratory report" | + | *@displayName="'''Laboratory report'''" |
| − | *@codeSystem="2.16.840.1.113883.6.1" | + | *@codeSystem="'''2.16.840.1.113883.6.1'''" |
| − | | Bezeichnet die "Dokumentklasse" in dem untergeordneten "translation"-Element. Einzig zulässige Wert für Labor- und Mikrobiologiebefund ist Laboratory report (11502-2). | + | | Bezeichnet die "Dokumentklasse" in dem untergeordneten "translation"-Element. Einzig zulässiger Wert für Labor- und Mikrobiologiebefund ist 11502-2 "Laboratory report". |
| | |- | | |- |
| | | colspan="2"| [[ILF:XDS_Metadaten_(Version_3)#title_2|title]] | | | colspan="2"| [[ILF:XDS_Metadaten_(Version_3)#title_2|title]] |
| Zeile 563: |
Zeile 409: |
| | | ./title | | | ./title |
| | | | | | |
| − | *"''Laborbefund''" | + | *"Laborbefund" |
| | | Der Titel des CDA Dokuments für den lesenden Empfänger. MUSS die Art des Dokuments (Dokumenttyp) widerspiegeln. | | | Der Titel des CDA Dokuments für den lesenden Empfänger. MUSS die Art des Dokuments (Dokumenttyp) widerspiegeln. |
| | | | |
| | Vorgeschlagene Titel wären zum Beispiel "Laborbefund" oder "Mikrobiologiebefund". | | Vorgeschlagene Titel wären zum Beispiel "Laborbefund" oder "Mikrobiologiebefund". |
| | |- | | |- |
| − | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#formatCode_.28und_formatCodeDisplayName.29_2|formatCode]] [TODO https://jira-neu.elga.gv.at/browse/ELGA-1118] | + | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#formatCode_.28und_formatCodeDisplayName.29_2|formatCode]] |
| | | M | | | M |
| | | ./hl7at:formatCode | | | ./hl7at:formatCode |
| | | | | | |
| − | *@code="<nowiki>urn:hl7-at:lab:3.0.0+YYYYYMMDD</nowiki>" | + | *@code="'''<nowiki>urn:hl7-at:lab:3.0.0+20211214</nowiki>'''" |
| − | *@displayName="ELGA Labor- und Mikrobiologiebefund 3.0.0+YYYYMMDD" | + | *@displayName="'''HL7 Austria Labor- und Mikrobiologiebefund 3.0.0+20211214'''" |
| − | *@codeSystem="1.2.40.0.34.5.37" | + | *@codeSystem="'''1.2.40.0.34.5.37'''" |
| − | | Version des vom CDA erfüllten ELGA Labor- und Mikrobiologiebefund Implementierungsleitfadens. | + | | Version des vom CDA erfüllten ELGA Implementierungsleitfaden für Labor- und Mikrobiologiebefund. |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#practiceSettingCode_.28und_practiceSettingCodeDisplayName.29_2|practiceSettingCode]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#practiceSettingCode_.28und_practiceSettingCodeDisplayName.29_2|practiceSettingCode]] |
| Zeile 581: |
Zeile 427: |
| | | ./hl7at:practiceSettingCode | | | ./hl7at:practiceSettingCode |
| | | | | | |
| − | *@code="''F028''" | + | *@code="F028" |
| − | *@displayName="''Labordiagnostik''" | + | *@displayName="Labordiagnostik" |
| | *@codeSystem="1.2.40.0.34.5.12" | | *@codeSystem="1.2.40.0.34.5.12" |
| − | | Fachliche Zuordnung des Dokuments. | + | | Fachliche Zuordnung des Dokuments aus dem Value Set "atcdabbr_PracticeSetting_VS". |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#creationTime_2|creationTime]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#creationTime_2|creationTime]] |
| Zeile 590: |
Zeile 436: |
| | | ./effectiveTime | | | ./effectiveTime |
| | | | | | |
| − | *@value="''20181213095800+0200''" | + | *@value="20181213095800+0200" |
| − | | Dokumentiert das Erstellungsdatum bzw. den Zeitpunkt, an dem das Dokument inhaltlich fertiggestellt wurde. | + | | Dokumentiert das medizinisch zutreffendste Datum - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc. |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#confidentialityCode_2|confidentialityCode]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#confidentialityCode_2|confidentialityCode]] |
| Zeile 597: |
Zeile 443: |
| | | ./confidentialityCode | | | ./confidentialityCode |
| | | | | | |
| − | *@code="''N''" | + | *@code="N" |
| − | *@displayName="''normal''" | + | *@displayName="normal" |
| | *@codeSystem="2.16.840.1.113883.5.25" | | *@codeSystem="2.16.840.1.113883.5.25" |
| | *@codeSystemName="HL7:Confidentiality" | | *@codeSystemName="HL7:Confidentiality" |
| Zeile 608: |
Zeile 454: |
| | | | | | |
| | *@code="de-AT" | | *@code="de-AT" |
| − | | Für ELGA ist in @code für CDA und Ableitungen in die XDSDocumentEntry-Metadaten derzeit ausschließlich der Wert "de-AT" zulässig. Für eHealth können in zukünftigen Versionen des Leitfadens weitere Sprachcodes erlaubt werden. | + | | Für ELGA-Dokumente ist in @code für CDA und Ableitungen in die XDSDocumentEntry-Metadaten derzeit ausschließlich der Wert "de-AT" zulässig. Für eHealth können in zukünftigen Versionen des Leitfadens weitere Sprachcodes erlaubt werden. |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#referenceIdList_2|referenceIdList]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#referenceIdList_2|referenceIdList]] |
| Zeile 614: |
Zeile 460: |
| | | ./setId | | | ./setId |
| | | | | | |
| − | *@root="''1.2.40.0.34.3.1.1058.1337''" | + | *@root="1.2.40.0.34.3.1.1058.1337" |
| − | *@extension="''999021''" | + | *@extension="999021" |
| | | Eindeutige ID des Dokumentensets. Diese bleibt über alle Versionen der Dokumente gleich (initialer Wert bleibt erhalten). Die setId SOLL unterschiedlich zu /ClinicalDocument/id sein. | | | Eindeutige ID des Dokumentensets. Diese bleibt über alle Versionen der Dokumente gleich (initialer Wert bleibt erhalten). Die setId SOLL unterschiedlich zu /ClinicalDocument/id sein. |
| | |- | | |- |
| Zeile 622: |
Zeile 468: |
| | | ./recordTarget/patientRole/id[1] | | | ./recordTarget/patientRole/id[1] |
| | | | | | |
| − | *@root="''1.2.40.0.34.99.111.1.2''" | + | *@root="1.2.40.0.34.99.111.1.2" |
| − | *@extension="''123''" | + | *@extension="123" |
| | | Patienten ID im Informationssystem des GDA, z.B.: im KIS eines Krankenhauses. | | | Patienten ID im Informationssystem des GDA, z.B.: im KIS eines Krankenhauses. |
| | |- | | |- |
| Zeile 632: |
Zeile 478: |
| | representedOrganization/id[1] | | representedOrganization/id[1] |
| | | | | | |
| − | *@root="''1.2.40.0.34.99.4.1234''" | + | *@root="1.2.40.0.34.99.4.1234" |
| | |rowspan="3"| ID und Name der Organisation (Kurzbezeichnung), der die Person angehört, wie im GDA-Index angegeben. | | |rowspan="3"| ID und Name der Organisation (Kurzbezeichnung), der die Person angehört, wie im GDA-Index angegeben. |
| | |- | | |- |
| | | | | | |
| − | *@root="''1.2.40.0.34.99.4''" | + | *@root="1.2.40.0.34.99.4" |
| − | *@extension="''1234''" | + | *@extension="1234" |
| | |- | | |- |
| | | ./author[1]/assignedAuthor/ | | | ./author[1]/assignedAuthor/ |
| | representedOrganization/name | | representedOrganization/name |
| | | | | | |
| − | *"''Krankenhaus XYZ''" | + | *"Krankenhaus XYZ" |
| | |- | | |- |
| | | authorPerson | | | authorPerson |
| Zeile 648: |
Zeile 494: |
| | | ./author[1]/assignedAuthor | | | ./author[1]/assignedAuthor |
| | | | | | |
| − | * ./id/@root="''1.2.40.0.34.99.111.1.2''" | + | * ./id/@root="1.2.40.0.34.99.111.1.2" |
| − | * ./id/@extension="''999021''" | + | * ./id/@extension="999021" |
| − | * ./assignedPerson/name/family="''Holzer''" | + | * ./assignedPerson/name/family="Holzer" |
| − | * ./assignedPerson/name/given[1]="''Daniela''" | + | * ./assignedPerson/name/given[1]="Daniela" |
| − | * ./assignedPerson/name/given[2]="''Chiara''" | + | * ./assignedPerson/name/given[2]="Chiara" |
| − | * ./assignedPerson/name/suffix="''BSc''" | + | * ./assignedPerson/name/suffix="BSc" |
| − | * ./assignedPerson/name/prefix[@qualifier="AC"]="''Dr.''" | + | * ./assignedPerson/name/prefix[@qualifier="AC"]="Dr." |
| | | Daten der Person (Name, ID, etc.) | | | Daten der Person (Name, ID, etc.) |
| | |- | | |- |
| Zeile 661: |
Zeile 507: |
| | | ./author[1]/functionCode | | | ./author[1]/functionCode |
| | | | | | |
| − | *@displayName="''Diensthabender Oberarzt''" | + | *@displayName="Diensthabender Oberarzt" |
| | | Rolle der Person. | | | Rolle der Person. |
| | |- | | |- |
| Zeile 668: |
Zeile 514: |
| | | ./author[1]/assignedAuthor/code | | | ./author[1]/assignedAuthor/code |
| | | | | | |
| − | *@displayName="''Fachärztin/Facharzt für Innere Medizin''" | + | *@displayName="Fachärztin/Facharzt für Innere Medizin" |
| − | | Fachrichtung des Verfassers des Dokuments aus ELGA_AuthorSpeciality. | + | | Fachrichtung des Verfassers des Dokuments aus dem Value Set "ELGA_AuthorSpeciality". |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#legalAuthenticator_2|legalAuthenticator]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#legalAuthenticator_2|legalAuthenticator]] |
| Zeile 675: |
Zeile 521: |
| | | ./legalAuthenticator/assignedEntity | | | ./legalAuthenticator/assignedEntity |
| | | | | | |
| − | * ./id/@root="''1.2.40.0.34.99.111.1.2''" | + | * ./id/@root="1.2.40.0.34.99.111.1.2" |
| − | * ./id/@extension="''999021''" | + | * ./id/@extension="999021" |
| − | * ./assignedPerson/name/family="''Holzer''" | + | * ./assignedPerson/name/family="Holzer" |
| − | * ./assignedPerson/name/given[1]="''Daniela''" | + | * ./assignedPerson/name/given[1]="Daniela" |
| − | * ./assignedPerson/name/given[2]="''Chiara''" | + | * ./assignedPerson/name/given[2]="Chiara" |
| − | * ./assignedPerson/name/suffix="''BSc''" | + | * ./assignedPerson/name/suffix="BSc" |
| − | * ./assignedPerson/name/prefix[@qualifier="AC"]="''Dr.''" | + | * ./assignedPerson/name/prefix[@qualifier="AC"]="Dr." |
| | | Rechtlicher Unterzeichner des Dokuments. | | | Rechtlicher Unterzeichner des Dokuments. |
| | |- | | |- |
| − | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#eventCodeList_.28und_eventCodeListDisplayName.29_2|eventCodeList]] | + | | colspan="2" rowspan="2"|[[ILF:XDS_Metadaten_(Version_3)#eventCodeList_.28und_eventCodeListDisplayName.29_2|eventCodeList]] |
| − | | R | + | | rowspan="2"|R |
| − | | ./documentationOf[n]/serviceEvent/code | + | | ./documentationOf'''[n]'''/serviceEvent/id |
| | + | | |
| | + | *@root="1.2.40.0.34.6.0.11.2.102" |
| | + | | rowspan="2" | Die ID und der Code des "serviceEvent"-Elements definieren, welche Inhalte ein Labor- oder Mikrobiologiebefund enthält sowie die Information, ob bzw. welche Inhalte maschinenlesbar vorliegen (siehe [[ILF:Labor-_und_Mikrobiologiebefund_(Version_3)#Vorgaben_zu_Dokumenten-Metadaten_.28XDS-Metadaten.29|Vorgaben zu Dokumenten-Metadaten (XDS-Metadaten)]]). |
| | + | |- |
| | + | | ./documentationOf'''[n]'''/serviceEvent/code |
| | | | | | |
| − | *@code="''100''" | + | *@code="100" |
| − | *@displayName="''Blutgruppenserologie''" | + | *@displayName="Blutgruppenserologie" |
| | *@codeSystem="1.2.40.0.34.5.11" | | *@codeSystem="1.2.40.0.34.5.11" |
| − | | Code der Gesundheitsdienstleistung.
| |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#serviceStartTime_.2F_serviceStopTime_2|serviceStartTime]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#serviceStartTime_.2F_serviceStopTime_2|serviceStartTime]] |
| | |R | | |R |
| − | |./documentationOf[1]/serviceEvent/ | + | |./documentationOf'''[1]'''/serviceEvent/ |
| | effectiveTime/low | | effectiveTime/low |
| | | | | | |
| − | *@value="''20181001082015+0200''" | + | *@value="20181001082015+0200" |
| | | Zeitpunkt des Behandlungsbeginns (erster medizinisch relevanter Behandlungstag dieser dokumentierter Gesundheitsdienstleistung) | | | Zeitpunkt des Behandlungsbeginns (erster medizinisch relevanter Behandlungstag dieser dokumentierter Gesundheitsdienstleistung) |
| | |- | | |- |
| | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#serviceStartTime_.2F_serviceStopTime_2|serviceStopTime]] | | | colspan="2"|[[ILF:XDS_Metadaten_(Version_3)#serviceStartTime_.2F_serviceStopTime_2|serviceStopTime]] |
| | |R | | |R |
| − | |./documentationOf[1]/serviceEvent/ | + | |./documentationOf'''[1]'''/serviceEvent/ |
| | effectiveTime/high | | effectiveTime/high |
| | | | | | |
| − | *@value="''20181213105900+0200''" | + | *@value="20181213105900+0200" |
| | | Zeitpunkt des Behandlungsendes (letzter medizinisch relevanter Behandlungstag dieser dokumentierter Gesundheitsdienstleistung, muss sich von Behandlungsbeginn unterscheiden) | | | Zeitpunkt des Behandlungsendes (letzter medizinisch relevanter Behandlungstag dieser dokumentierter Gesundheitsdienstleistung, muss sich von Behandlungsbeginn unterscheiden) |
| | |- | | |- |
| Zeile 714: |
Zeile 564: |
| | location/healthCareFacility/code | | location/healthCareFacility/code |
| | | | | | |
| − | *@code="''300''" | + | *@code="300" |
| − | *@displayName="''Allgemeine Krankenanstalt''" | + | *@displayName="Allgemeine Krankenanstalt" |
| | *@codeSystem="1.2.40.0.34.5.2" | | *@codeSystem="1.2.40.0.34.5.2" |
| | | Klassifizierung des GDA. | | | Klassifizierung des GDA. |
| − | |} | + | |}<ref group="Tabelle">Übersichtstabelle über die Dokument-Metadaten (XDS-Metadaten)</ref>: ''Übersichtstabelle über die Dokument-Metadaten (XDS-Metadaten)'' |
| | </div> | | </div> |
| | | | |
| − | =Anwendungsfälle= | + | =Konformitätsprüfung= |
| − | Die Einsatzszenarien für dieses Datenaustauschformat werden in Form von Anwendungsfälle beschrieben, um dem Leser den Hintergrund zu vermitteln. Diese Beschreibung der Anwendungsfälle ist nicht normativ und keine Vorentscheidung für die tatsächliche Umsetzung.
| + | Ein zu diesem Implementierungsleitfaden konformes CDA-Dokument ist zunächst ein valides CDA Release 2.0 XML-Dokument mit [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#CDA_Header|Header]] und [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#CDA_Body|Body]]. Darüber hinaus erfüllt es alle in diesem Leitfaden festgelegten "Geschäftsregeln". |
| | + | |
| | + | Dies spiegelt ein generelles Konzept im Umgang mit Dokumenten wieder: die Validierung in zwei Schritten. Im ersten Schritt stellt dies die Validierung gegen zugehörige '''W3C Schemas''' dar. Das verwendete Schema ist das geringfügig erweiterte offizielle CDA Release 2.0 Schema (siehe [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schema-Pr.C3.BCfung|Schema-Prüfung]]). Darüber hinaus existieren eine Reihe von '''Schematron''' Regeln, die für einen zweiten Validierungsschritt genutzt werden und letztlich die Detailregelungen in diesem Leitfaden wiedergeben, sowie die Einhaltung der Geschäftsregeln (Optionalität, Kardinalität/Multiplizität, Datentypen, Wertebereiche, Abhängigkeiten) sicherstellen (siehe [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schematron-Pr.C3.BCfung|Schematron-Prüfung]]). Geschäftsregeln für Abschnitte oder Elemente werden auch technisch zu '''"Templates"''' zusammengefasst. Eine XML-Instanz, die kein valides CDA-Dokument ist oder sich nicht gegen das XSD-Schema validieren lässt oder im Widerspruch zu den angegebenen Geschäftsregeln steht, ist kein gültiges CDA-Dokument im Sinne dieses Implementierungsleitfadens. |
| | + | {{BeginYellowBox}}Hinweis: Nicht alle Geschäftsregeln können mit Schema oder Schematron geprüft werden (etwa Inhalte von Multimedia-Attachments, Dokumentengröße). Zusätzliche Validierungsschritte sind gegebenenfalls notwendig, um alle Regeln überprüfen zu können. |
| | + | {{EndYellowBox}} |
| | + | {{BeginILFBox}}Die Kapitel zu den technischen Konformitätsprüfungen von CDA-Dokumenten sind im allgemeinen Leitfaden unter den folgenden Links zu finden: |
| | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schema-Pr.C3.BCfung|Schema-Prüfung]] |
| | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Schematron-Pr.C3.BCfung|Schematron-Prüfung]] |
| | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Online-Validation_von_CDA-Dokumenten|Online-Validation von CDA-Dokumenten]] |
| | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Hinweise_zur_Konformit.C3.A4tspr.C3.BCfung|Hinweise zur Konformitätsprüfung]] |
| | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Abnahmepr.C3.BCfung_f.C3.BCr_ELGA_e-Befunde|Abnahmeprüfung für ELGA e-Befunde]] |
| | + | *[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Zertifizierung|Zertifizierung]] |
| | + | {{EndILFBox}} |
| | + | |
| | + | =Datentypen= |
| | + | {{BeginILFBox}}Im Kapitel [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Datentypen|Datentypen des allgemeinen Leitfadens]] werden nur die Datentypen beschrieben, die in ELGA CDA-Dokumenten wie diesem zur Anwendung kommen. Für weiterführende Informationen wird auf den zugrundeliegenden Standard Health Level Seven Version 3 (V3), Normative Edition verwiesen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | =Vorgaben zum medizinischen Inhalt= |
| | + | ==Labor- und Mikrobiologiebefund== |
| | + | Die inhaltliche Definition beruht auf den Mindestvorgaben der österreichischen Gesellschaft für Labormedizin und klinischen Chemie (ÖGLMKC) und wurde weiter verfeinert. Die folgende Tabelle zeigt einen Überblick über die inhaltlich abzubildenden medizinisch relevanten Daten. |
| | + | |
| | + | {| class="wikitable" width="100%" |
| | + | |- |
| | + | ! style="text-align:left" width="20%" | Feld |
| | + | ! style="text-align:left" width="40%" | Beschreibung |
| | + | ! style="text-align:left" width="20%" | Bereich |
| | + | |- |
| | + | ! style="text-align:left" colspan="3" | ''Allgemeine Befundinformationen'' |
| | + | |- style="background:#FFFFFF" |
| | + | | Zeitpunkt der Auftragserfassung |
| | + | | Datum und Zeitpunkt, an dem das analysierende Labor die Anforderung vom Zuweiser in der Labor-EDV erfasst hat. |
| | + | | [[#Documentation_Of_Service_Event_-_Labor_und_Mikrobiologie|Documentation Of Service Event - Labor und Mikrobiologie]]: |
| | + | documentationOf/serviceEvent/effectiveTime/low |
| | + | |- style="background:#FFFFFF" |
| | + | | Auftragsdiagnose (Zuweiserdiagnose) bzw. Überweisungsgrund |
| | + | | Vom Auftraggeber bestimmte und dem Labor übermittelte Verdachtsdiagnose bzw. der Überweisungsgrund |
| | + | | [[#.C3.9Cberweisungsgrund_-_codiert|Überweisungsgrund - codiert]] oder [[#.C3.9Cberweisungsgrund_-_uncodiert|Überweisungsgrund - uncodiert]] |
| | + | |- style="background:#FFFFFF" |
| | + | | Angeforderte Analyse bzw. Fragestellung |
| | + | | Vom Auftraggeber angeforderte Analyse(-spektrum) bzw. die Fragestellung |
| | + | | [[#Angeforderte_Untersuchungen_-_codiert|Angeforderte Untersuchungen - codiert]] oder [[#Angeforderte_Untersuchungen_-_uncodiert|Angeforderte Untersuchungen - uncodiert]] |
| | + | |- style="background:#FFFFFF" |
| | + | | Befundtext |
| | + | | Kommentar zum gesamten Befund |
| | + | | [[#Befundbewertung|Befundbewertung]] |
| | + | |- |
| | + | ! style="text-align:left" colspan="3" | ''Information zum Untersuchungsmaterial'' |
| | + | |- style="background:#FFFFFF" |
| | + | | Zeitpunkt der Gewinnung des Untersuchungsmaterials |
| | + | | Damit ist jenes Datum und Zeitpunkt gemeint, an dem das Untersuchungsmaterial für die geplante Analyse gewonnen wurde. Die Dokumentation des Zeitpunkts der Gewinnung ist in der Verantwortung der entnehmenden Person, die in vielen Fällen mit dem Befundersteller nicht identisch ist, weil Untersuchungsmaterial meist zur Analyse an Labors versendet werden. Daher ist der Zeitpunkt vielfach im Labor nicht feststellbar. |
| | + | | [[#Specimen_Collection|Specimen Collection]] |
| | + | |
| | + | procedure/effectiveTime |
| | + | |- style="background:#FFFFFF" |
| | + | | Zeitpunkt des Einlangens des Untersuchungsmaterials |
| | + | | Datum und Zeit der Annahme des Untersuchungsmaterials im Labor |
| | + | | [[#Specimen Received|Specimen Received]] |
| | + | |
| | + | act/effectiveTime |
| | + | |- style="background:#FFFFFF" |
| | + | | Art des Untersuchungsmaterials (Specimen Type) |
| | + | | Art des Untersuchungsmaterials |
| | + | | [[#Specimen_Collection|Specimen Collection]] |
| | + | |
| | + | procedure/participant[@typeCode='PRD']/<br/> |
| | + | participantRole[@classCode='SPEC']/playingEntity/code |
| | + | |- style="background:#FFFFFF" |
| | + | | Entnahmeort |
| | + | | Angabe der Körperstelle, von der das Untersuchungsmaterial stammt |
| | + | | [[#Specimen_Collection|Specimen Collection]] |
| | + | |
| | + | procedure/targetSiteCode |
| | + | |- style="background:#FFFFFF" |
| | + | | ID des Untersuchungsmaterials |
| | + | | Eindeutige Nummer des Untersuchungsmaterials |
| | + | | [[#Specimen_Collection|Specimen Collection]] |
| | + | |
| | + | procedure/participant[@typeCode='PRD']/<br/> |
| | + | participantRole[@classCode='SPEC']/id |
| | + | |- style="background:#FFFFFF" |
| | + | | Entnehmende Person (Performer) |
| | + | | Person, welche die Entnahme des Untersuchungsmaterials durchgeführt hat |
| | + | | [[#Specimen_Collection|Specimen Collection]] |
| | + | |
| | + | procedure/performer |
| | + | |- style="background:#FFFFFF" |
| | + | | Allgemeine Anmerkungen des Labors zur Qualität des Untersuchungsmaterials |
| | + | | Präanalytik pro Untersuchungsmaterial zu dessen Qualität |
| | + | | [[#Specimen Received|Specimen Received]] |
| | + | |
| | + | act/entryRelationship |
| | + | |- |
| | + | ! style="text-align:left" colspan="3" | ''Allgemeine Laborergebnisse'' |
| | + | |- style="background:#FFFFFF" |
| | + | | Gruppierung / Befundgruppen |
| | + | | Analysengruppierung |
| | + | | [[#Laboratory Specialty Section|Laboratory Specialty Section]] bzw. [[#Laboratory Battery Organizer|Laboratory Battery Organizer]] |
| | + | |- style="background:#FFFFFF" |
| | + | | ID des Tests |
| | + | | Eindeutige Codierung des Tests |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/id |
| | + | |- style="background:#FFFFFF" |
| | + | | Analysenbezeichnung |
| | + | | Bezeichnung der Analyse |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/code |
| | + | |- style="background:#FFFFFF" |
| | + | | Ergebnis |
| | + | | |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/value[@value] |
| | + | |- style="background:#FFFFFF" |
| | + | | Einheit |
| | + | | |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/value[@unit] |
| | + | |- style="background:#FFFFFF" |
| | + | | Referenzbereich |
| | + | | Für die Beurteilung relevante Referenzwerte. |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/referenceRange |
| | + | |- style="background:#FFFFFF" |
| | + | | Befundinterpretation |
| | + | | Codierte Bewertung des Ergebnisses |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/interpretationCode |
| | + | |- style="background:#FFFFFF" |
| | + | | Ergebniskommentar |
| | + | | Kommentar des Labors zu einem einzelnen Testergebnis |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/entryRelationship/act/<br/> |
| | + | templateId[@root='1.2.40.0.34.6.0.11.3.11'] |
| | + | |- style="background:#FFFFFF" |
| | + | | Externes Labor |
| | + | | Kennzeichen ob ein Ergebnis extern ermittelt wurde |
| | + | | [[#Laboratory Observation|Laboratory Observation]] |
| | + | |
| | + | observation/performer/assignedEntity/code[@code='E'] |
| | + | |- |
| | + | ! style="text-align:left" colspan="3" | ''Kulturergebnisse'' |
| | + | |- style="background:#FFFFFF" |
| | + | | Erreger (Isolat) |
| | + | | Der durch eine Kulturmethode ermittelte Erreger. |
| | + | | [[#Laboratory Isolate Organizer|Laboratory Isolate Organizer]] |
| | + | |- style="background:#FFFFFF" |
| | + | | Antibiogramm (inklusive MHK) |
| | + | | Ergebnis der Bestimmung der Empfindlichkeit bzw. Resistenz von Erregern gegenüber Antibiotika. Anlassbezogen wird das Ergebnis interpretiert (R, S, I) und/oder die minimale Hemmkonzentration (MHK) angegeben. |
| | + | | [[#Laboratory Isolate Organizer|Laboratory Isolate Organizer]] und darin in einer [[#Laboratory Observation|Laboratory Observation]] |
| | + | |}<ref group="Tabelle">Im Labor- und Mikrobiologiebefund abzubildende medizinische Daten</ref>: ''Im Labor- und Mikrobiologiebefund abzubildende medizinische Daten'' |
| | + | |
| | + | ==Laborbefund== |
| | + | |
| | + | ===Befundbereiche (Laboratory Specialty Section)=== |
| | + | |
| | + | Der Laborbefund kann mehrere Abschnitte aus verschiedenen Laborbereichen (z.B. Hämatologie, Toxikologie, Urindiagnostik, etc.) beinhalten. Diese werden im Dokument als einzelne "Befundbereiche" umgesetzt und bilden somit die erste Gliederungsebene des Bodys. Die Befundbereiche entsprechen strukturell der "Laboratory Specialty Section" der IHE<ref name="IHEPaLMTF3"/> (siehe auch Template [[#Laboratory Specialty Section|Laboratory Specialty Section]]). Folgende Grafik zeigt die mögliche Gliederung auf der ersten Ebene innerhalb des Bodys. |
| | + | |
| | + | [[Datei:Gliederung_nach_Befundbereichen.png|thumb|none|300px|<ref group="Abbildung">Gliederung nach Befundbereichen (Laboratory Specialty Sections)</ref>: Gliederung nach Befundbereichen (Laboratory Specialty Sections)]] |
| | + | |
| | + | Die derzeit für den Laborbefund verwendbaren Befundbereiche werden im Rahmen des hierarchisch organisierten Value Sets "ELGA_Laborstruktur" definiert, wobei für Befundbereiche nur Einträge des Level 1 verwendet werden dürfen. Die folgende Tabelle gibt einen auszugsweisen Überblick über die derzeit festgelegten Befundbereiche. Bei der Verwendung der Befundbereiche ist die Reihenfolge gemäß Value Set verpflichtend einzuhalten. |
| | + | {| class="wikitable" |
| | + | ! Code |
| | + | ! Befundbereich (Laboratory Specialty Section) |
| | + | |- |
| | + | | 100 |
| | + | | Blutgruppenserologie |
| | + | |- |
| | + | | 200 |
| | + | | Blutgasanalytik |
| | + | |- |
| | + | | 300 |
| | + | | Hämatologie |
| | + | |- |
| | + | | 400 |
| | + | | Gerinnung/Hämostaseologie |
| | + | |- |
| | + | | … |
| | + | | … |
| | + | |}<ref group="Tabelle">Liste der Befundbereiche, auszugsweise gemäß Value Set "ELGA_Laborstruktur", die sich auch im Value Set "ELGA_Laborparameter" wiederfinden.</ref>: ''Liste der Befundbereiche, auszugsweise gemäß Value Set "ELGA_Laborstruktur", die sich auch im Value Set "ELGA_Laborparameter" wiederfinden.'' |
| | + | |
| | + | ===Befundgruppen=== |
| | + | Innerhalb der Befundbereiche erfolgt in der Regel eine Strukturierung und Gliederung der Ergebnisse zur besseren Lesbarkeit und Auffindbarkeit in "Befundgruppen". Für die Umsetzung der Befundgruppen im Body werden [[#Laboratory Battery Organizer|Laboratory Battery Organizer]] verwendet. Level 2 des Value Sets "ELGA_Laborstruktur" definiert die zulässigen Befundgruppen. Grundsätzlich ist die Reihenfolge der Befundgruppen gemäß Value Set verpflichtend einzuhalten. Es besteht jedoch auch die Möglichkeit, Ergebnisse ohne Befundgruppenstrukturierung zu übermitteln. |
| | + | |
| | + | [[Datei:Laborbefund_Strukturierungsmöglichkeiten_Body.png|thumb|none|600px|<ref group="Abbildung">Strukturierungsmöglichkeiten im Laborbefund im Body</ref>: Strukturierungsmöglichkeiten im Laborbefund im Body]] |
| | + | |
| | + | Der folgende Ausschnitt aus einem Laborbefunden enthält die Befundbereiche "Hämatologie" und "Hämostaseologie" sowie die dazugehörigen Befundgruppen "Blutbild", "Knochenmark Morphologie" bzw. "Hämostaseologie Globaltest". |
| | + | |
| | + | [[Datei:Laborbefund_Beispiel_Ausschnitt.png|thumb|none|600px|<ref group="Abbildung">Ausschnitt aus einem Laborbefund</ref>: Ausschnitt aus einem Laborbefund]] |
| | + | |
| | + | ===Harmonisierung des Befundaufbaus – Value Set "ELGA_Laborparameter"=== |
| | + | |
| | + | Im Rahmen der Arbeiten zum vorliegenden Dokument wurde in der Expertengruppe die grundsätzliche Übereinkunft getroffen, auch die Befundgruppen und die damit verbundene Testzuordnung entsprechend österreichweit abzustimmen. Die Strukturierung eines Laborbefundes wurde in Form des hierarchischen Value Sets '''"ELGA_Laborparameter"''' festgelegt. |
| | + | {{BeginYellowBox}}Strukturierung, Reihenfolge der Parameter sowie die Bezeichnung der Parameter sind durch das Value Set "ELGA_Laborparameter" verpflichtend vorgegeben! |
| | + | {{EndYellowBox}} |
| | + | Eine Hilfestellung zum Mapping der lokalen Codes auf die vorgeschriebenen Codes des Value Sets bietet der "Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund"<ref>Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund [https://www.elga.gv.at/fileadmin/user_upload/Dokumente_PDF_MP4/CDA/Leitfaden_LOINC_Anwendung_in_ELGA_V1.03.pdf]</ref>. |
| | + | |
| | + | ==Mikrobiologiebefund== |
| | + | |
| | + | Unter den Analysen des Laborbefundes finden sich viele aus dem Bereich der Mikrobiologie. Grundsätzlich können alle Analysen auch in der "klassischen" Struktur des Laborbefundes dargestellt werden (siehe [[#Laborbefund|Laborbefund]]). Allerdings entspricht die Struktur des Laborbefundes nicht dem eines üblichen Mikrobiologiebefundes, der einem bestimmten Muster folgt, welches den Untersuchungsverlauf widerspiegelt: |
| | + | |
| | + | # Beschreibung des entnommenen Materials (z.B. Mittelstrahlharn) inklusive makroskopischer Beurteilung |
| | + | # Mikroskopische Analyse des Materials (z.B. Erythrozyten, Leukozyten, grampositive Bakterien) |
| | + | # Kultureller Erregernachweis |
| | + | #* Benennung der Reinkulturen (Isolate) mit Nennung der taxonomischen Bestimmung der Mikroorganismen (z.B. Streptococcus pyogenes) ggf. mit Angabe des Serovars/Pathovars. |
| | + | #* Angabe des Antibiogramms mit Interpretation (R, S, I) und/oder minimaler Hemmkonzentration (MHK) |
| | + | # Molekulare Erregernachweise |
| | + | # Infektionsserologie |
| | + | |
| | + | Die Struktur des Mikrobiologiebefundes wird durch spezielle "Laboratory Specialty Sections", die jeweils mit einem fixen Code versehen sind, vorgegeben. Details dazu sind den [[#Mikrobiologiebefund_2|CDA Strukturen des Bodys des Mikrobiologiebefundes]] zu entnehmen. |
| | + | {{BeginYellowBox}}Werden mikrobiologische Analysen in einem "normalen" Laborbefund festgehalten, so sind diese dem entsprechenden Befundbereich und - wenn möglich - der passenden Befundgruppe zuzuordnen. |
| | + | {{EndYellowBox}} |
| | + | |
| | + | [[Datei:Mikrobiologiebefund_Beispiel_Ausschnitt.PNG|thumb|none|600px|<ref group="Abbildung">Ausschnitt aus einem Mikrobiologiebefund</ref>: Ausschnitt aus einem Mikrobiologiebefund]] |
| | + | |
| | + | ==Hinweise für akkreditierte Laboratorien gem. ISO 15189:2012== |
| | | | |
| − | TODO: Die Definition in passendes Kapitel verschieben.
| + | Die Vorgaben dieses Implementierungsleitfadens erlauben die Erzeugung von Befunden gemäß ISO 15189:2012 "Medizinische Laboratorien - Anforderungen an die Qualität und Kompetenz"<ref>ISO 15189:2012 "Medizinische Laboratorien - Anforderungen an die Qualität und Kompetenz" [https://www.iso.org/standard/56115.html]</ref>, speziell in Hinblick auf die dort angegebenen Kapitel 5.8 (Befundberichte) und 5.9 (Freigabe der Ergebnisse). Für akkreditierte Laboratorien sind neben der ISO 15189:2012 folgende Hinweise zu beachten: |
| | | | |
| − | '''Definition:''' Der Laborbefund wurde für die Arbeit an diesem Leitfaden wie folgt definiert:
| + | ===Angabe des Akkreditierungs-Logos=== |
| | | | |
| − | ''Ein Laborbefund (aus dem Bereich der med. u. chem. Labordiagnostik) ist der fachärztlich vidierte, kommentierte/interpretierte Befund morphologischer, biologischer, chemischer, molekularer, physikalischer und spezieller immunologischer Untersuchungsverfahren aus Körpersäften, der Beurteilung ihrer morphologischen Bestandteile sowie von ab- und ausgeschiedenem Untersuchungsmaterial zur Erkennung physiologischer Eigenschaften, krankhafter Zustände, zu Verlaufskontrollen und zur Gesundheitsvorsorge/Prophylaxe.''
| + | Das Akkreditierungs-Logo kann neben dem Logo des Labors angegeben werden. Technisch erfolgt das über die Section "Brieftext", das Logo ist ggf. gemeinsam mit dem Logo des Labors in einer Grafikdatei anzugeben (siehe [[#Brieftext|Brieftext]]). |
| | | | |
| − | ''ELGA Laborbefunde umfassen u. a. klinische Chemie und Immunchemie, Hämatologie (Erkrankungen des Blutes) und Hämostaseologie (Störungen der Blutgerinnung), Proteinchemie, Serologie, molekulare Diagnostik, Toxikologie, Drugmonitoring, Mikrobiologie, Infektionsserologie, Zytologie, Untersuchungen und die Hilfestellung für andere Fächer im Rahmen von Therapievorschlägen bei Gerinnungsstörungen, Antikoagulanzientherapien, der Impfkontrolle, Vorsorgediagnostik und Risikostratifizierung.''
| + | [[Datei:Angabe des Akkreditierungs-Logos im Briefkopf.png|thumb|none|500px|<ref group="Abbildung">Angabe des Akkreditierungs-Logos im Brieftext</ref>: Angabe des Akkreditierungs-Logos im Brieftext]] |
| | | | |
| − | ''Untersuchungen des Sonderfaches "Blutgruppenserologie und Transfusionsmedizin" werden in einer gesonderten ELGA Dokumentenklasse (geplant) abgehandelt. ''
| + | Ein Labor muss nicht zwingend für alle Analysen, die es durchführen kann, akkreditiert sein. Das Akkreditierungs-Logo kann angegeben werden, sobald es akkreditierte Analysen gibt; wenn "nicht akkreditierte Analysen" am Befund erscheinen, soll bei diesen angegeben werden können, dass das Labor für diese Analyse nicht akkreditiert ist. Für die entsprechend Markierung wird die Verwendung von Anmerkungszeichen (wie z.B. *) und Endnoten empfohlen. |
| | | | |
| − | ''Sofern keine andere Regelung zutrifft, obliegt die Entscheidung ob ein Befund in ELGA gestellt wird dem Befundersteller.''
| + | ===Angabe des Analyse- bzw. Messverfahrens=== |
| | + | |
| | + | Wenn der Name der Analysen keinen Rückschluss auf die Methode erlaubt, aber Analyse- bzw. Messverfahren dennoch angegeben werden sollen (ISO 15189:2012, Vorgabe 5.8.3 a)), soll dies als Kommentar zur Analyse erfolgen (siehe [[#Laboratory Observation|Laboratory Observation]] wie ein Kommentar zur Analyse codiert werden kann). |
| | + | |
| | + | ===Vorgaben für den Befunddruck === |
| | + | |
| | + | Einige Vorgaben von ISO 15189:2012 beziehen sich auf Qualitätsmerkmale für gedruckte Befunde (Vorgabe 5.8.3 d) "Identifizierung des Patienten und den Aufenthaltsort des Patienten auf jeder Seite" und Vorgabe 5.8.3 p) "Seitenzahl zur Gesamtzahl der Seiten"). Dieser Leitfaden definiert ein elektronisches Format, das diese Anforderungen grundsätzlich unterstützt. Am ELGA-Portal (Zugriff für Bürger) werden Tools zur Darstellung eingesetzt, die diese Vorgaben unterstützen. Diese Tools werden auch von der ELGA GmbH zum Download bereitgestellt (Referenzstylesheet, CDA2PDF auf http://www.elga.gv.at/CDA). |
| | | | |
| − | ''Im ELGA Laborbefund dürfen nur dann Ergebnisse aus genetischen Analysen enthalten sein, wenn ihre Dokumentation in Übereinstimmung mit dem Gentechnikgesetz (GTG § 71a, BGBl. I Nr. 127/2005) erfolgt. ''
| + | =Anwendungsfälle / User Stories= |
| | + | Die Einsatzszenarien für dieses Datenaustauschformat werden in Form von Anwendungsfällen beschrieben, um dem Leser den erforderlichen Hintergrund zu vermitteln. Die Beschreibung der Anwendungsfälle ist nicht normativ und keine Vorentscheidung für die tatsächliche Umsetzung. |
| | | | |
| − | Der in diesem Leitfaden beschriebene Laborbefund dient also zum Austausch von fertiggestellten, und fachärztlich vidierten Befunden innerhalb und zwischen Einrichtungen des Gesundheitswesens. Ein wesentlicher Nutzer der Befunde ist auch der Patient selbst, der die Befunde über das ELGA Bürgerportal einsehen wird. | + | Der in diesem Leitfaden beschriebene Labor- und Mikrobiologiebefund dient zum Austausch von fertiggestellten, und fachärztlich vidierten Befunden innerhalb und zwischen Einrichtungen des Gesundheitswesens. Ein wesentlicher Nutzer der Befunde ist auch der Patient selbst, der die Befunde über das ELGA Bürgerportal einsehen wird. |
| | | | |
| − | Der in diesem Leitfaden beschriebene Laborbefund ist zur Dokumentation und Kommunikation (vollständig) fertiggestellter Laborbefunde gedacht.
| + | Die Regelung, welche Befunde in ELGA einzustellen sind, ist nicht Teil dieses Leitfadens. |
| | | | |
| − | Der hier beschriebene Laborbefund ist nicht vorgesehen um Zwischenergebnisse und Nachrichten über einzelne Prozessschritte zu kommunizieren, wie etwa | + | Der in diesem Leitfaden beschriebene Labor- und Mikrobiologiebefund ist grundsätzlich zur Dokumentation und Kommunikation (vollständig) fertiggestellter Labor- und Mikrobiologiebefunde gedacht, wobei auch die Möglichkeit besteht, zu dokumentieren, wenn Analyseergebnisse noch ausständig sind. Idealerweise wird ein aktualisiertes CDA in die ELGA eingebracht, sobald die Ergebnisse vorliegen. Gleichfalls werden Ergänzungen und Korrekturen von Labor- und Mikrobiologiebefunden unterstützt. |
| − | * die Anforderung von Laboruntersuchungen
| |
| − | * das Einlangen einer Probe im Labor
| |
| − | * den Beginn, Stornierung oder die Fertigstellung einzelner Untersuchungen
| |
| − | Ergänzungen und Korrekturen von Laborbefunden werden unterstützt | |
| | | | |
| − | ==Anwendungsfall LAB01: "Laboruntersuchung eines niedergelassenen Labors"==
| + | Der hier beschriebene Labor- und Mikrobiologiebefund ist nicht als Workflow-Dokument konzipiert worden, um z.B. Zwischenergebnisse und Nachrichten über einzelne Prozessschritte zu kommunizieren. Eventuell kann ein CDA-Dokument intern als solches verwendet werden, um beispielsweise |
| − | Typischerweise entstehen Laborbefunde in medizinischen Labors. Einerseits sind das niedergelassene Labors, die als selbständige Unternehmen Laboruntersuchungen anbieten. Diese werden vielfach auf Zuweisung von Patienten durch praktische Ärzte im niedergelassenen Bereich tätig. Die Entstehung eines Laborbefundes beginnt mit einer Überweisung durch einen niedergelassenen Arzt, oder mit einer Anforderung innerhalb eines Spitals. Entweder wird das Spezimen am Patienten gleichzeitig entnommen, und dann ins Labor geschickt oder der Patient muss das Labor aufsuchen, und das Spezimen wird dann erst dort entnommen. Nach Abschluss der Untersuchung wird der Befund dem zuweisenden Arzt und/oder dem Patienten in Papierform übermittelt.
| + | * die Anforderung von Analysen, |
| | + | * das Einlangen des Untersuchungsmaterials im Labor oder |
| | + | * den Beginn, Stornierung oder die Fertigstellung einzelner Analysen abzubilden. |
| | | | |
| − | Laboruntersuchungen im Rahmen ambulanter Aufenthalte in einem Spital fallen ebenso unter diesen Anwendungsfall.
| + | ==Anwendungsfall LAB01: "Analysen aus niedergelassenen Labors und nicht-stationären Fällen"== |
| | + | Typischerweise entstehen Laborbefunde in medizinischen Labors. Das sind neben Labor-Abteilungen von Spitälern auch niedergelassene Labors, die als selbständige Unternehmen Analysen anbieten. Diese werden vielfach auf Zuweisung von Patienten durch praktische Ärzte im niedergelassenen Bereich tätig. Die Entstehung eines Laborbefundes beginnt mit einer Überweisung durch einen niedergelassenen Arzt oder mit einer Anforderung aus einem nicht-stationären Fall innerhalb eines Spitals. Entweder wird das Untersuchungsmaterial am Patienten gleichzeitig entnommen und dann ins Labor geschickt oder der Patient muss das Labor aufsuchen, und das Untersuchungsmaterial wird dann erst dort entnommen. Nach Abschluss der Analyse wird der Befund dem zuweisenden Arzt und/oder dem Patienten in Papierform übermittelt. |
| | | | |
| − | ==Anwendungsfall LAB02: "Laboruntersuchung im Rahmen eines stationären Aufenthalts in einem Spital"== | + | ==Anwendungsfall LAB02: "Analysen im Rahmen eines stationären Aufenthalts in einem Spital"== |
| − | Im Rahmen von stationären Aufenthalten von Patienten in Spitälern kommt es in der Regel zu einer Reihe von Laboruntersuchungen, die in der internen Krankengeschichte (meistens auch elektronisch) abgelegt werden. Relevante Befunde werden dem einweisenden Arzt bzw. dem Patienten im Zuge der Entlassungsdokumentation mit übermittelt. Dieses passiert oftmals als Teil des Entlassungsbriefes. Welche Werte und welche Befunde entsprechende Relevanz haben um weitergeleitet zu werden, entscheidet das jeweilige ärztliche Fachpersonal in der Klinik. | + | Im Rahmen von stationären Aufenthalten von Patienten in Spitälern kommt es in der Regel zu einer Reihe von Analysen, die in der spitalsinternen Krankengeschichte (meistens auch elektronisch) abgelegt werden. Relevante Befunde werden dem einweisenden Arzt bzw. dem Patienten im Zuge der Entlassungsdokumentation mit übermittelt. Dieses passiert oftmals als Teil des Entlassungsbriefes. Welche Werte und welche Befunde entsprechende Relevanz haben, um weitergeleitet zu werden, entscheidet das jeweilige ärztliche Fachpersonal in der Klinik. |
| | | | |
| − | ==Anwendungsfall LAB03: "Teilweise externe Vergabe von Laboruntersuchungen"== | + | ==Anwendungsfall LAB03: "Teilweise externe Vergabe von Analysen"== |
| − | In vielen Fällen kommt es zu Kooperationen zwischen Laborbefund erstellenden Organisationen. Folgende Fälle seien angeführt: | + | In vielen Fällen kommt es zu Kooperationen zwischen Laborbefund-erstellenden Organisationen. Folgende Fälle seien angeführt: |
| − | * Spitäler kooperieren mit niedergelassenen Labors. Zum Teil verfügen nicht alle Spitäler über eigene Labors, andererseits werden auch Spezialuntersuchungen, die das Spitalslabor nicht durchführt, an niedergelassene Labors vergeben. | + | * Spitäler kooperieren mit niedergelassenen Labors. Einerseits verfügen nicht alle Spitäler über eigene Labors, andererseits werden auch Spezialuntersuchungen, die das Spitalslabor nicht durchführt, an niedergelassene Labors vergeben. |
| − | * Niedergelassene Labors verfügen nicht über das volle Leistungsspektrum und senden Laboruntersuchungen an Spitallabors, welche spezielle Parameter messen können. | + | * Niedergelassene Labors verfügen nicht über das volle Leistungsspektrum und senden Analysen an Spitallabors, welche spezielle Parameter messen können. |
| − | * Es bestehen Kooperationen zwischen mehreren Spitälern. Einerseits sind das oft Spitäler, die dem gleichen Spitalsträger angehören. Teilweise bestehen auch Kooperationen zwischen Spitälern unterschiedlicher Träger, die durch die örtliche Nähe leicht Proben austauschen können. | + | * Es bestehen Kooperationen zwischen mehreren Spitälern. Das sind oft Spitäler, die dem gleichen Spitalsträger angehören. Teilweise bestehen auch Kooperationen zwischen Spitälern unterschiedlicher Träger, die durch die örtliche Nähe leicht Untersuchungsmaterial austauschen können. |
| | | | |
| − | In allen Fällen werden einzelne Labortests nicht selbst durchgeführt sondern diese Tests an ein externes kooperierendes Labor vergeben. Das externe Labor führt dann den Test durch, und übermittelt die Ergebnisse an das ursprünglich für die Untersuchungen zuständige Labor. Dort werden dann die vom externen Labor ermittelten Testergebnisse in den eigenen Laborbefund eingefügt. Das ursprünglich zuständige Labor, das den Befund erstellt, muss in diesem Fall die extern erbrachten Testergebnisse als solche erkennbar kennzeichnen. | + | In allen Fällen werden einzelne Analysen nicht selbst durchgeführt, sondern diese Tests an ein externes kooperierendes Labor vergeben. Das externe Labor führt dann die Analyse durch und übermittelt die Ergebnisse an das ursprünglich für die Analyse zuständige Labor. Dort werden dann die vom externen Labor ermittelten Testergebnisse in den eigenen Laborbefund integriert. Das ursprünglich zuständige Labor, das den Befund erstellt, muss in diesem Fall die extern erbrachten Testergebnisse als solche erkennbar kennzeichnen (siehe [[#Performer_-_Laboratory|Performer - Laboratory]]). |
| | | | |
| | ==Anwendungsfall LAB04: "Update von Laborbefunden"== | | ==Anwendungsfall LAB04: "Update von Laborbefunden"== |
| − | Ein fertiggestellter Laborbefund wird korrigiert oder ergänzt, um | + | Ein fertiggestellter Labor- oder Mikrobiologiebefund wird korrigiert oder ergänzt, um |
| | * die Inhalte des Befundes zu korrigieren (etwa das Ergebnis einer Analyse), | | * die Inhalte des Befundes zu korrigieren (etwa das Ergebnis einer Analyse), |
| | * einzelne (fehlerhafte) Analysen nachträglich aus dem Befund zu stornieren oder | | * einzelne (fehlerhafte) Analysen nachträglich aus dem Befund zu stornieren oder |
| Zeile 771: |
Zeile 849: |
| | Änderungen sollen im Text für den Leser klar kenntlich gemacht werden (eine codierte Angabe kann im narrativen Text mit Revisionsmarken erfolgen). | | Änderungen sollen im Text für den Leser klar kenntlich gemacht werden (eine codierte Angabe kann im narrativen Text mit Revisionsmarken erfolgen). |
| | | | |
| − | Eine Korrekturversion MUSS in ELGA immer alle zum Befund gehörigen Analysen enthalten, da die Vorversion als veraltet (deprecated) gekennzeichnet wird. Stornierte Analysen sind explizit mit dem entsprechenden StatusCode zu kennzeichnen. | + | Eine Korrekturversion MUSS in ELGA immer alle zum Befund gehörigen Analysen enthalten, da die Vorversion als veraltet (deprecated) gekennzeichnet wird. Stornierte Analysen sind explizit mit dem entsprechenden "statusCode" zu kennzeichnen. |
| | | | |
| | Für den Leser/Empfänger gilt: Eine neue Version ersetzt die alte Version des Befundes, alle Analysen sollen beim Import ersetzt bzw. überschrieben werden. Sollte eine Analyse in der neuen Version fehlen, soll diese als "storniert" interpretiert werden. | | Für den Leser/Empfänger gilt: Eine neue Version ersetzt die alte Version des Befundes, alle Analysen sollen beim Import ersetzt bzw. überschrieben werden. Sollte eine Analyse in der neuen Version fehlen, soll diese als "storniert" interpretiert werden. |
| | | | |
| − | =Dataset des Labor- und Mikrobiologiebefunds= | + | =Dataset= |
| | Das Dataset (auch "Datenarten" oder "Konzepte") listet alle mit der Arbeitsgruppe abgestimmten Inhalte des Leitfadens auf. Es enthält Beschreibungen der Elemente mit Synonymen. | | Das Dataset (auch "Datenarten" oder "Konzepte") listet alle mit der Arbeitsgruppe abgestimmten Inhalte des Leitfadens auf. Es enthält Beschreibungen der Elemente mit Synonymen. |
| − |
| |
| − | Dataset-Elemente können auf das CDA Datenmodell gemappt werden. In den Metadaten eines Templates sind alle assoziierten Konzepte auf einen Blick ersichtlich. Im Template-Body wird das assoziierte Konzept beim entsprechenden Datenelement angezeigt.
| |
| | | | |
| | Die Live-Version des Datasets in Art-Decor kann unter folgendem [https://art-decor.org/decor/services/RetrieveDataSet?id=1.2.40.0.34.777.11.1.1&language=de-DE&effectiveDate=2020-08-15T10:04:20&format=html&hidecolumns=3456bcdefghijklmnop Link] betrachtet werden. | | Die Live-Version des Datasets in Art-Decor kann unter folgendem [https://art-decor.org/decor/services/RetrieveDataSet?id=1.2.40.0.34.777.11.1.1&language=de-DE&effectiveDate=2020-08-15T10:04:20&format=html&hidecolumns=3456bcdefghijklmnop Link] betrachtet werden. |
| Zeile 786: |
Zeile 862: |
| | Die Struktur des CDA Austauschformats ist in den nachfolgenden Kapiteln im Detail beschrieben. | | Die Struktur des CDA Austauschformats ist in den nachfolgenden Kapiteln im Detail beschrieben. |
| | | | |
| − | Der Header entspricht mit Ausnahme der erwähnten Abweichungen den bisherigen ELGA CDA-Leitfäden ("Allgemeiner Leitfaden"). Der Body enthält die tatsächlichen (medizinischen) Inhalte des Dokuments. Dieses Dokument existiert ausschließlich in einer voll strukturierten Form, eine Unterscheidung der Interoperabilitätsstufen ist daher nicht notwendig. | + | Der Header entspricht im Wesentlichen den Vorgaben des [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)|Allgemeinen Leitfadens]]. Der Body enthält die tatsächlichen (medizinischen) Inhalte des Dokuments. Dieses Dokument existiert ausschließlich in einer voll strukturierten Form, eine Unterscheidung der Interoperabilitätsstufen ist daher nicht notwendig. |
| | | | |
| | ==Übersichtstabelle der CDA Strukturen des Headers== | | ==Übersichtstabelle der CDA Strukturen des Headers== |
| | Dieses Kapitel gibt einen Überblick über die Elemente des CDA Headers und den Vorgaben bezüglich Kardinalität und Konformität. | | Dieses Kapitel gibt einen Überblick über die Elemente des CDA Headers und den Vorgaben bezüglich Kardinalität und Konformität. |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Die jeweiligen Links in der letzten Spalte zeigen auf die einzelnen Header Elemente. Wo es zu keinen strukturellen Änderungen im Rahmen dieses Leitfadens gekommen ist, wird die Definition des Allgemeinen Implementierungsleitfadens verlinkt. |
| − | Die jeweiligen Links in der letzten Spalte zeigen auf die einzelnen Header Elemente im allgemeinen Leitfaden. | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| − | Der aktuelle Leitfaden ist für den ELGA Kontext entwickelt worden, kann jedoch auch für andere Zwecke, welche als eHealth zusammengefasst werden, verwendet werden. | + | Der aktuelle Leitfaden ist für den ELGA Kontext entwickelt worden, kann jedoch auch für andere Zwecke, welche als eHealth zusammengefasst werden, verwendet werden. |
| | + | |
| | + | {| class="wikitable" width="100%" |
| | + | |- |
| | + | ! style="text-align:left" width="20%" |Element |
| | + | ! Kard/Konf ELGA |
| | + | ! Kard/Konf eHealth |
| | + | ! style="text-align:left" width="60%" |Bedeutung / Link zum Kapitel |
| | + | |- style="background:#FFFFFF" |
| | + | |realmCode |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Hoheitsbereich_des_Dokuments_.28.22realmCode.22.29|Hoheitsbereich des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |typeId |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Dokumentformat_.28.22typeId.22.29|Kennzeichnung CDA R2]] |
| | + | |- style="background:#FFFFFF" |
| | + | |templateId |
| | + | | 3..* M |
| | + | |3..* M |
| | + | |Definition im Allgemeinen Implementierungsleitfaden: [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#ELGA_Implementierungsleitfaden-Kennzeichnung_.28.22templateId.22.29|Kennzeichnung von Strukturvorschriften]] |
| | | | |
| − | [TODO (GKL) wie funktioniert das im eHealth-Bereich, dass die Kardinalitäten/Konformitäten anders sind? Das bedeutet doch, dass im eHealth-Bereich nicht auf das Schematron bzw. Online-Validator zurückgegriffen werden kann, oder?] | + | Erlaubte Werte für den [[#Laborbefund_3|Laborbefund]] bzw. [[#Mikrobiologiebefund_3|Mikrobiologiebefund]] sind den jeweiligen Definitionen der Document Level Templates zu entnehmen. |
| | + | |- style="background:#FFFFFF" |
| | + | |id |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Dokumenten-Id_.28.22id.22.29|Dokumenten-Id]] |
| | + | |- style="background:#FFFFFF" |
| | + | |code |
| | + | translation |
| | + | | 1..1 M |
| | + | 1..1 M |
| | + | |1..1 M |
| | + | 0..* R |
| | + | |Definition im Allgemeinen Implementierungsleitfaden: [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Dokumentenklasse_.28.22code.22.29|Klassifikation des Dokuments (fein und grob)]] |
| | | | |
| − | {{ILF:Übersichtstabelle_der_CDA_Strukturen_des_Headers}}
| + | Erlaubte Werte für den [[#Laborbefund_3|Laborbefund]] bzw. [[#Mikrobiologiebefund_3|Mikrobiologiebefund]] sind den jeweiligen Definitionen der Document Level Templates zu entnehmen. |
| | + | |- style="background:#FFFFFF" |
| | + | |title |
| | + | | 1..1 M |
| | + | |1..1 M |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Titel_des_Dokuments_.28.22title.22.29|Titel des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |sdtc:statusCode |
| | + | | 0..1 C |
| | + | |0..1 O |
| | + | |Definition im Allgemeinen Implementierungsleitfaden: [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Status_des_Dokuments_.28.22sdtc:statusCode.22.29|Status des Dokuments]] |
| | | | |
| − | '''[TODO Allgemeine Anmerkungen zur Tabelle]'''
| + | Spezielle Hinweise in Bezug auf Befunde mit ausständigen Analysen ("Wert folgt") sind den Definitionen der [[#Document Level Templates|Document Level Templates]] zu entnehmen. |
| − | * [TODO documentationOf müsste 1..* R sein, weil in der Tabelle zu den Zeitangaben im Header ist es auch 1..* R]
| + | |- style="background:#FFFFFF" |
| | + | |hl7at:terminologyDate |
| | + | | 1..1 M |
| | + | |0..1 O||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Terminologiedatum_.28.22hl7at:terminologyDate.22.29|Terminologie-Datum des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |hl7at:formatCode |
| | + | | 1..1 M |
| | + | |0..1 O |
| | + | |Definition im Allgemeinen Implementierungsleitfaden: [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#FormatCode_.28.22hl7at:formatCode.22.29|FormatCode des Dokuments]] |
| | | | |
| − | === Labor- und Mikrobiologiebefund === | + | Erlaubter Wert für den Labor- bzw. Mikrobiologiebefund ist den Definitionen der [[#Document Level Templates|Document Level Templates]] zu entnehmen. |
| | + | |- style="background:#FFFFFF" |
| | + | |hl7at:practiceSettingCode |
| | + | | 1..1 M |
| | + | |0..1 O||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Fachliche_Zuordnung_des_Dokuments_.28.22hl7at:practiceSettingCode.22.29|Fachliche Zuordnung des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |effectiveTime |
| | + | | 1..1 M |
| | + | |1..1 M |
| | + | |Angabe des medizinisch zutreffendsten Datums - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc. |
| | + | |- style="background:#FFFFFF" |
| | + | |confidentialityCode |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Vertraulichkeitscode_.28.22confidentialityCode.22.29|Vertraulichkeitscode]] |
| | + | |- style="background:#FFFFFF" |
| | + | |languageCode |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Sprachcode_des_Dokuments_.28.22languageCode.22.29|Sprachcode des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |setId |
| | + | versionNumber |
| | + | | 1..1 M |
| | | | |
| − | Für den Labor- und Mikrobiologiebefund gibt es folgende Anpassungen hinsichtlich der Kardinalität/Konformität einzelner Header-Elemente.
| + | 1..1 M |
| | + | |1..1 M |
| | | | |
| − | {| class="wikitable" width="100%"
| + | 1..1 M |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Versionierung_des_Dokuments_.28.22setId.22_und_.22versionNumber.22.29|Versionierung des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |recordTarget |
| | + | | 1..1 M |
| | + | |0..1 C |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Patient_.28.22recordTarget.2FpatientRole.22.29|Patient]] |
| | |- | | |- |
| − | ! style="text-align:left" width="20%" | Element
| + | |recordTarget de-identified |
| − | ! Kard/Konf Laborbefund
| + | | 0..0 NP |
| − | ! Kard/Konf Mikrobiologiebefund
| + | |0..1 C |
| − | ! style="text-align:left" width="60%" | Bedeutung / Link zum Kapitel
| + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Alternative_Spezifikation_de-identifizierter_Patient|Anonymer oder pseudonymisierter Patient]] |
| | + | |- style="background:#FFFFFF" |
| | + | |author |
| | + | | 1..* M |
| | + | |1..* M |
| | + | |Definition im Allgemeinen Implementierungsleitfaden: [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Verfasser_des_Dokuments_.28.22author.22.29|Verfasser des Dokuments]] |
| | + | |
| | + | Für Labor- bzw. Mikrobiologiebefunde MUSS immer zumindest eine Person als Author angeführt sein. |
| | + | |- style="background:#FFFFFF" |
| | + | |dataEnterer |
| | + | | 0..1 O |
| | + | |0..1 O |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Personen_der_Dateneingabe_.28.22dataEnterer.22.29|Personen der Dateneingabe]] |
| | + | |- style="background:#FFFFFF" |
| | + | |informant |
| | + | | 0..0 NP |
| | + | |0..0 NP |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Informant_Body|Informant]] |
| | + | |- style="background:#FFFFFF" |
| | + | |custodian |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Verwahrer_des_Dokuments_.28.22custodian.22.29|Verwahrer des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |informationRecipient |
| | + | | 0..* O |
| | + | |0..* O |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Beabsichtigte_Empf.C3.A4nger_des_Dokuments_.28.22informationRecipient.22.29|Beabsichtigte Empfänger des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |legalAuthenticator |
| | + | | 1..1 M |
| | + | |1..1 M||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Rechtlicher_Unterzeichner_.28.22legalAuthenticator.22.29|Rechtlicher Unterzeichner]], wird im speziellen Leitfaden definiert. |
| | |- style="background:#FFFFFF" | | |- style="background:#FFFFFF" |
| − | | informant | + | |authenticator |
| − | | '''0..0 NP''' | + | | 0..* O |
| − | | '''0..0 NP''' | + | |0..* O |
| − | | [[ILF:Allgemeiner_Implementierungsleitfaden_2020#Informant_Body|Informant]] | + | |Definition im Allgemeinen Implementierungsleitfaden: [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Weitere_Unterzeichner_.28.22authenticator.22.29|Weitere Unterzeichner]] |
| | + | |
| | + | Definition für den Labor- bzw. Mikrobiologiebefund: [[#Laboratory_Results_Validator|Laboratory Results Validator]] |
| | + | |- style="background:#FFFFFF" |
| | + | |participant[@typeCode='REF'] |
| | + | | 1..1 R |
| | + | |0..1 O |
| | + | |[[#Participant_Auftraggeber_.2F_Ordering_Provider|Participant Auftraggeber / Ordering Provider]] |
| | + | |
| | + | '''Achtung:''' Der [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Einweisender.2FZuweisender.2F.C3.9Cberweisender_Arzt|einweisende/zuweisende/überweisende Arzt]] wie er im Allgemeinen Implementierungsleitfaden definiert ist, DARF im Labor- bzw. Mikrobiologiebefund NICHT verwendet werden! |
| | + | |- style="background:#FFFFFF" |
| | + | |participant[@typeCode='CALLBCK'] |
| | + | | 0..1 R |
| | + | |0..1 O |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Fachlicher_Ansprechpartner|Fachlicher Ansprechpartner]] |
| | + | |- style="background:#FFFFFF" |
| | + | |participant |
| | + | | 0..* O |
| | + | |0..* O |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Weitere_Beteiligte_.28.22participant.22.29|Weitere Beteiligte]], die im Labor- bzw. Mikrobiologiebefund vorkommen können. |
| | |- style="background:#FFFFFF" | | |- style="background:#FFFFFF" |
| − | | legalAuthenticator | + | |inFulfillmentOf |
| − | | '''1..1 M''' | + | | 1..* M |
| − | | '''1..1 M''' | + | |1..* M |
| − | | [[ILF:Allgemeiner_Implementierungsleitfaden_2020#Rechtlicher_Unterzeichner_.28.22legalAuthenticator.22.29|Rechtlicher Unterzeichner]] | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Zuweisung_und_Ordermanagement|Zuweisung und Ordermanagement]] |
| | |- style="background:#FFFFFF" | | |- style="background:#FFFFFF" |
| | | documentationOf | | | documentationOf |
| | + | |
| | serviceEvent | | serviceEvent |
| − | | '''1..* M''' | + | |
| | + | performer |
| | + | | 1..* M |
| | + | |
| | + | 1..1 M |
| | + | |
| | + | 0..* C |
| | + | | 1..* M |
| | + | |
| | + | 1..1 M |
| | + | |
| | + | 0..* C |
| | + | | [[#Documentation Of Service Event - Labor und Mikrobiologie|Documentation Of Service Event - Labor und Mikrobiologie]] |
| | + | |- style="background:#FFFFFF" |
| | + | |relatedDocument |
| | + | |0..1 O |
| | + | |0..1 O |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Bezug_zu_vorgehenden_Dokumenten|Bezug zu vorgehenden Dokumenten]] |
| | + | |- style="background:#FFFFFF" |
| | + | |authorization |
| | + | | 0..0 NP |
| | + | |0..* O||[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Einverst.C3.A4ndniserkl.C3.A4rung|Einverständniserklärung]] |
| | + | |- style="background:#FFFFFF" |
| | + | |componentOf |
| | + | |
| | + | encompassingEncounter |
| | + | | 0..1 O |
| | + | |
| | 1..1 M | | 1..1 M |
| − | | '''1..1 M''' | + | |0..1 O |
| | + | |
| | 1..1 M | | 1..1 M |
| − | |siehe Spezifikation der [[#Document_Level_Templates|Document Level Templates]] | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Informationen_zum_Patientenkontakt|Patientenkontakt (Aufenthalt)]] |
| − | |}<ref group="Tabelle">Übersichtstabelle der angepassten CDA Strukturen des Headers für Labor- und Mikrobiologiebefund</ref>:''Übersichtstabelle der adaptierten CDA Strukturen des Headers für Labor- und Mikrobiologiebefund'' | + | |- |
| | + | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Headers</ref>:''Übersichtstabelle der CDA Strukturen des Headers'' |
| | | | |
| | ==Übersichtstabelle der Header-Elemente für dokumenten-relevante Zeitpunkte/Zeitspannen== | | ==Übersichtstabelle der Header-Elemente für dokumenten-relevante Zeitpunkte/Zeitspannen== |
| | Dieses Kapitel gibt einen Überblick über die Elemente des CDA Headers mit Zeitangaben und ihre Zusammenhänge. | | Dieses Kapitel gibt einen Überblick über die Elemente des CDA Headers mit Zeitangaben und ihre Zusammenhänge. |
| − | {{BeginILFBox}} | + | {{BeginILFBox}}Die jeweiligen Links in der letzten Spalte zeigen auf die einzelnen Header Elemente. Wo es zu keinen strukturellen Änderungen im Rahmen dieses Leitfadens gekommen ist, wird die Definition des Allgemeinen Implementierungsleitfadens verlinkt. |
| − | Die jeweiligen Links in der letzten Spalte zeigen auf die einzelnen Header Elemente im allgemeinen Leitfaden. | |
| | {{EndILFBox}} | | {{EndILFBox}} |
| | Der aktuelle Leitfaden ist für den ELGA Kontext entwickelt worden, kann jedoch auch für andere Zwecke, welche als eHealth zusammengefasst werden, verwendet werden. | | Der aktuelle Leitfaden ist für den ELGA Kontext entwickelt worden, kann jedoch auch für andere Zwecke, welche als eHealth zusammengefasst werden, verwendet werden. |
| − | {{ILF:Übersicht_der_Zeitelemente_im_Header}}
| |
| − |
| |
| − | === Labor- und Mikrobiologiebefund ===
| |
| − |
| |
| − | Für den Labor- und Mikrobiologiebefund gibt es aufgrund der Anpassung hinsichtlich der Kardinalität/Konformität einzelner Header-Elemente folgende Änderungen.
| |
| | | | |
| | {| class="wikitable" width="100%" | | {| class="wikitable" width="100%" |
| | |- | | |- |
| − | ! style="text-align:left" width="20%" | Element | + | ! style="text-align:left" width="20%" |Element |
| − | ! Kard/Konf Laborbefund | + | ! Kard/Konf |
| − | ! Kard/Konf Mikrobiologiebefund | + | ELGA |
| | + | ! Kard/Konf |
| | + | eHealth |
| | ! style="text-align:left" width="60%" | Bedeutung | | ! style="text-align:left" width="60%" | Bedeutung |
| − | ! Link zum Kapitel | + | ! style="text-align:left" width="60%" | Link zum Kapitel |
| | + | |- style="background:#FFFFFF" |
| | + | |hl7at:terminologyDate |
| | + | | 1..1 M |
| | + | |0..1 O |
| | + | |Das Datum, an dem die lokal zur Implementierung verwendeten Value Sets mit dem österreichischen Terminologieserver abgeglichen wurden, wird hier angegeben. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Terminologiedatum_.28.22hl7at:terminologyDate.22.29|Terminologie-Datum des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |effectiveTime |
| | + | | 1..1 M |
| | + | |1..1 M |
| | + | |Angabe des medizinisch zutreffendsten Datums - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc. |
| | + | | |
| | + | |- style="background:#FFFFFF" |
| | + | |recordTarget |
| | + | |
| | + | birthTime |
| | + | | 1..1 M |
| | + | |
| | + | 1..1 R |
| | + | |0..1 C |
| | + | |
| | + | 1..1 R |
| | + | |Der Geburtstag des Patienten. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Patient_.28.22recordTarget.2FpatientRole.22.29|Patient]] |
| | + | |- style="background:#FFFFFF" |
| | + | |recordTarget |
| | + | |
| | + | deceasedTime |
| | + | | 1..1 M |
| | + | |
| | + | 0..1 R |
| | + | |0..1 C |
| | + | |
| | + | 0..1 R |
| | + | |Das Sterbedatum des Patienten. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Patient_.28.22recordTarget.2FpatientRole.22.29|Patient]] |
| | + | |- style="background:#FFFFFF" |
| | + | |author |
| | + | |
| | + | time |
| | + | | 1..* M |
| | + | |
| | + | 0..1 R |
| | + | |1..* M |
| | + | |
| | + | 0..1 R |
| | + | |Das jeweilige Datum, an welche der jeweilige Autor neue medizinische Informationen hinzugefügt hat. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Verfasser_des_Dokuments_.28.22author.22.29|Verfasser des Dokuments]] |
| | + | |- style="background:#FFFFFF" |
| | + | |dataEnterer |
| | + | |
| | + | time |
| | + | | 0..1 O |
| | + | |
| | + | 0..1 R |
| | + | |0..1 O |
| | + | |
| | + | 0..1 R |
| | + | |Das Datum, an welchem eine Schreibkraft die Informationen aus einem Medium in das CDA Dokument überträgt, ohne weitere fachliche Informationen hinzuzufügen. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Personen_der_Dateneingabe_.28.22dataEnterer.22.29|Personen der Dateneingabe]] |
| | |- style="background:#FFFFFF" | | |- style="background:#FFFFFF" |
| − | | legalAuthenticator | + | |legalAuthenticator |
| | + | |
| | time | | time |
| − | | '''1..1 M''' | + | | 1..1 M |
| | + | |
| | 1..1 R | | 1..1 R |
| − | | '''1..1 M''' | + | |1..1 M |
| | + | |
| | 1..1 R | | 1..1 R |
| − | | Die Zeitpunkte, an welchem das Dokument von der berechtigten Personen vidiert wurde. Diese Person ist der Hauptunterzeichner. Dieser Zeitpunkt sollte, wenn vorhanden, nach /ClinicalDocument/author/time und /ClinicalDocument/dataEnterer/time liegen. | + | |Die Zeitpunkte, an welchem das Dokument von der berechtigten Personen vidiert wurde. Diese Person ist der Hauptunterzeichner. Dieser Zeitpunkt sollte, wenn vorhanden, nach /ClinicalDocument/author/time und /ClinicalDocument/dataEnterer/time liegen. |
| | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Rechtlicher_Unterzeichner_.28.22legalAuthenticator.22.29|Rechtlicher Unterzeichner]] | | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Rechtlicher_Unterzeichner_.28.22legalAuthenticator.22.29|Rechtlicher Unterzeichner]] |
| | |- style="background:#FFFFFF" | | |- style="background:#FFFFFF" |
| − | | documentationOf<br/> | + | |authenticator |
| − | serviceEvent<br/> | + | |
| | + | time |
| | + | | 0..* O |
| | + | |
| | + | 1..1 R |
| | + | |0..* O |
| | + | |
| | + | 1..1 R |
| | + | |Die Zeitpunkte, an welchem das Dokument von den einzelnen berechtigten Personen vidiert wurde. Diese Personen sind die weiteren Unterzeichner. |
| | + | |[[#Laboratory Results Validator|Laboratory Results Validator]] |
| | + | |- style="background:#FFFFFF" |
| | + | |''Notfallkontakt / Auskunftsberechtigte Person'' |
| | + | participant [templateId[@root='1.2.40.0.34.6.0.11.1.27']] |
| | + | |
| | + | time |
| | + | | 0..* O |
| | + | |
| | + | 0..1 O |
| | + | |0..* O |
| | + | |
| | + | 0..1 O |
| | + | |Zeitraum, in dem der angegebene Kontakt den Notfall-Kontakt darstellt. |
| | + | Wird nur angegeben, wenn der Kontakt bereits absehbar nur in einem eingeschränkten Zeitraum zur Verfügung steht. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Weitere_Beteiligte_.28.22participant.22.29|Weitere Beteiligte]] |
| | + | |- style="background:#FFFFFF" |
| | + | |''Versicherter/Versicherungparticipant'' |
| | + | participant [templateId[@root='1.2.40.0.34.6.0.11.1.26']] |
| | + | |
| | + | time |
| | + | | 0..* O |
| | + | |
| | + | 0..1 O |
| | + | |0..* O |
| | + | |
| | + | 0..1 O |
| | + | |Gültigkeitszeitraum der Versicherungspolizze. |
| | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Weitere_Beteiligte_.28.22participant.22.29|Weitere Beteiligte]] |
| | + | |- style="background:#FFFFFF" |
| | + | |documentationOf |
| | + | |
| | + | serviceEvent |
| | + | |
| | + | effectiveTime |
| | + | | 1..* M |
| | + | |
| | + | 1..1 M |
| | + | |
| | + | 1..1 M |
| | + | |1..* M |
| | + | |
| | + | 1..1 M |
| | + | |
| | + | 1..1 M |
| | + | |Zeitraum der durchgeführten Gesundheitsdienstleistung. |
| | + | |[[#Documentation Of Service Event - Labor und Mikrobiologie|Documentation Of Service Event - Labor und Mikrobiologie]] |
| | + | |- style="background:#FFFFFF" |
| | + | |componentOf |
| | + | |
| | + | encompassingEncounter |
| | + | |
| | effectiveTime | | effectiveTime |
| − | | '''1..* M'''<br/> | + | | 0..1 O |
| − | 1..1 M<br/> | + | |
| | + | 1..1 M |
| | + | |
| | 1..1 M | | 1..1 M |
| − | | '''1..1 M'''<br/> | + | |0..1 O |
| − | 1..1 M<br/> | + | |
| | + | 1..1 M |
| | + | |
| | 1..1 M | | 1..1 M |
| − | | Zeitraum der durchgeführten Gesundheitsdienstleistung. | + | |Zeitraum des Patientenkontakts. |
| − | | siehe Spezifikation der [[#Document_Level_Templates|Document Level Templates]] | + | |[[ILF:Allgemeiner_Implementierungsleitfaden_2020#Informationen_zum_Patientenkontakt|Patientenkontakt (Aufenthalt)]] |
| − | |}<ref group="Tabelle">Übersichtstabelle der angepassten Header-Elemente für Zeitpunkte/Zeitspannen in Labor- und Mikrobiologiebefund</ref>:''Übersichtstabelle der angepassten Header-Elemente für Zeitpunkte/Zeitspannen in Labor- und Mikrobiologiebefund'' | + | |- |
| | + | |}<ref group="Tabelle">Übersichtstabelle der Header-Elemente für Zeitpunkte/Zeitspannen</ref>:''Übersichtstabelle der Header-Elemente für Zeitpunkte/Zeitspannen'' |
| | | | |
| | ==Übersichtstabelle der CDA Strukturen des Bodys== | | ==Übersichtstabelle der CDA Strukturen des Bodys== |
| | | | |
| − | Die folgenden Tabellen geben die im ELGA Labor- bzw. Mikrobiologiebefund verwendeten Sektionen und Entries wieder. Angaben über die Verwendung einzelner Elemente können - sofern nicht in dieser Tabelle aufgeführt - in den jeweiligen Sektions- oder Entry-Spezifikationen gefunden werden (aus Gründen der Übersichtlichkeit wurde auf die Darstellung aller Ebenen verzichtet). | + | Die folgenden Tabellen geben die im ELGA Labor- bzw. Mikrobiologiebefund verwendeten Sections und Entries wieder. Angaben über die Verwendung einzelner Elemente können - sofern nicht in dieser Tabelle aufgeführt - in den jeweiligen Section- oder Entry-Spezifikationen gefunden werden (aus Gründen der Übersichtlichkeit wurde auf die Darstellung aller Ebenen verzichtet). |
| | | | |
| | === Laborbefund === | | === Laborbefund === |
| | | | |
| | {| class="wikitable" width="100%" | | {| class="wikitable" width="100%" |
| − | ! colspan="4" |Sektion bzw. Entry | + | ! colspan="4" |Section bzw. Entry |
| − | !OID | + | !Template ID |
| | !Kard/Konf | | !Kard/Konf |
| | !Kapitel | | !Kapitel |
| Zeile 889: |
Zeile 1.244: |
| | | 1.2.40.0.34.6.0.11.2.69 | | | 1.2.40.0.34.6.0.11.2.69 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Brieftext|Link]] | + | | [[#Brieftext|Template-Spezifikation]] |
| | |- | | |- |
| − | | colspan="4" |Überweisungsgrund - optional codiert | + | | colspan="7" | Der "Überweisungsgrund" kann codiert oder uncodiert angegeben werden: |
| | + | |- |
| | + | | |
| | + | | colspan="3" |Überweisungsgrund - codiert |
| | | 1.2.40.0.34.6.0.11.2.6 | | | 1.2.40.0.34.6.0.11.2.6 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Überweisungsgrund - optional codiert|Link]] | + | | [[#Überweisungsgrund - codiert|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Konsultationsgrund Problem Concern Entry |
| | + | | 1.2.40.0.34.6.0.11.3.30 |
| | + | | 1..* M |
| | + | | [[#Konsultationsgrund Problem Concern Entry|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" |Überweisungsgrund - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.114 |
| | + | | 0..1 O |
| | + | | [[#Überweisungsgrund - uncodiert|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="7" | Die "Anamnese - Labor und Mikrobiologie" kann codiert oder uncodiert angegeben werden: |
| | + | |- |
| | + | | |
| | + | | colspan="3" | Anamnese - Labor und Mikrobiologie - codiert |
| | + | | 1.2.40.0.34.6.0.11.2.109 |
| | + | | 0..1 O |
| | + | | [[#Anamnese - Labor und Mikrobiologie - codiert|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Anamnese Entry - Labor und Mikrobiologie |
| | + | | 1.2.40.0.34.6.0.11.3.12 |
| | + | | 1..* M |
| | + | | [[#Anamnese Entry - Labor und Mikrobiologie|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | |
| | + | | Anamnese Observation - Labor und Mikrobiologie |
| | + | | 1.2.40.0.34.6.0.11.3.164 |
| | + | | 1..* M |
| | + | | [[#Anamnese Observation - Labor und Mikrobiologie|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" | Anamnese - Labor und Mikrobiologie - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.111 |
| | + | | 0..1 O |
| | + | | [[#Anamnese - Labor und Mikrobiologie - uncodiert|Template-Spezifikation]] |
| | |- | | |- |
| − | | colspan="4" |Angeforderte Untersuchungen - optional codiert | + | | colspan="7" | Die "Angeforderte Untersuchung" kann codiert oder uncodiert angegeben werden: |
| | + | |- |
| | + | | |
| | + | | colspan="3" |Angeforderte Untersuchungen - codiert |
| | | 1.2.40.0.34.6.0.11.2.15 | | | 1.2.40.0.34.6.0.11.2.15 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Angeforderte Untersuchungen - optional codiert|Link]] | + | | [[#Angeforderte Untersuchungen - codiert|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Angeforderte Untersuchung Entry |
| | + | | 1.2.40.0.34.6.0.11.3.169 |
| | + | | 1..* M |
| | + | | [[#Angeforderte Untersuchung Entry|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" |Angeforderte Untersuchungen - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.112 |
| | + | | 0..1 O |
| | + | | [[#Angeforderte Untersuchungen - uncodiert|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" |Probeninformation (Specimen Section) | | | colspan="4" |Probeninformation (Specimen Section) |
| | | 1.2.40.0.34.6.0.11.2.93 | | | 1.2.40.0.34.6.0.11.2.93 |
| − | | 0..1 C | + | | 0..1 C<sup>1</sup> |
| − | | [[#Probeninformation (Specimen Section)|Link]] | + | | [[#Probeninformation (Specimen Section)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 910: |
Zeile 1.326: |
| | | 1.2.40.0.34.6.0.11.3.160 | | | 1.2.40.0.34.6.0.11.3.160 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Probeninformation (Specimen Entry)|Link]] | + | | [[#Probeninformation (Specimen Entry)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 917: |
Zeile 1.333: |
| | | 1.2.40.0.34.6.0.11.3.161 | | | 1.2.40.0.34.6.0.11.3.161 |
| | | 1..* M | | | 1..* M |
| − | | [[#Specimen Collection|Link]] | + | | [[#Specimen Collection|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 925: |
Zeile 1.341: |
| | | 1.2.40.0.34.6.0.11.3.162 | | | 1.2.40.0.34.6.0.11.3.162 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Specimen Received|Link]] | + | | [[#Specimen Received|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" |Laboratory Specialty Section | | | colspan="4" |Laboratory Specialty Section |
| | | 1.2.40.0.34.6.0.11.2.102 | | | 1.2.40.0.34.6.0.11.2.102 |
| − | | 1..* M TODO: vielleicht nur 1..* R? | + | | 1..* M |
| − | | [[#Laboratory Specialty Section|Link]] | + | | [[#Laboratory Specialty Section|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 936: |
Zeile 1.352: |
| | | 1.2.40.0.34.6.0.11.3.25 | | | 1.2.40.0.34.6.0.11.3.25 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur]] | + | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur des Laboratory Report Data Processing Entries]] |
| | |- | | |- |
| | | colspan="4" | Befundbewertung | | | colspan="4" | Befundbewertung |
| | | 1.2.40.0.34.6.0.11.2.103 | | | 1.2.40.0.34.6.0.11.2.103 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Befundbewertung|Link]] | + | | [[#Befundbewertung|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 947: |
Zeile 1.363: |
| | | 1.2.40.0.34.6.0.11.3.11 | | | 1.2.40.0.34.6.0.11.3.11 |
| | | 1..1 R | | | 1..1 R |
| − | | [[#Comment Entry|Link]] | + | | [[#Comment Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" | Beilagen | | | colspan="4" | Beilagen |
| | | 1.2.40.0.34.6.0.11.2.71 | | | 1.2.40.0.34.6.0.11.2.71 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Beilagen|Link]] | + | | [[#Beilagen|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" | Abschließende Bemerkung | | | colspan="4" | Abschließende Bemerkung |
| | | 1.2.40.0.34.6.0.11.2.70 | | | 1.2.40.0.34.6.0.11.2.70 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Abschließende Bemerkung|Link]] | + | | [[#Abschließende Bemerkung|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="7" | <sup>1</sup> Die Dokumentation des Untersuchungsmaterials kann auf zwei Arten erfolgen: |
| | + | * Enthält ein Befund nur einen Befundbereich ("Laboratory Specialty Section"), so kann die Codierung innerhalb der einen Section erfolgen. |
| | + | '''ODER''' |
| | + | * Bei Verwendung von mehreren Befundbereichen in einem Befund kann es zu Überschneidungen der Untersuchungsmaterialien kommen (ein spezielles Untersuchungsmaterial kann in zwei Befundbereichen analysiert werden). Die CDA Level 3 Codierung eines Untersuchungsmaterials darf jedoch nur einmal im gesamten Befund erfolgen. Daher sind die Informationen zu den Untersuchungsmaterialien in einer eigenen, führenden "Probeninformation (Specimen Section)" zu codieren. |
| | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Bodys des Laborbefundes</ref>:''Übersichtstabelle der CDA Strukturen des Bodys des Laborbefundes'' | | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Bodys des Laborbefundes</ref>:''Übersichtstabelle der CDA Strukturen des Bodys des Laborbefundes'' |
| | | | |
| | === Mikrobiologiebefund === | | === Mikrobiologiebefund === |
| | | | |
| | + | {{BeginYellowBox}}Derzeit werden Mikrobiologiebefunde mit mehreren Untersuchungsmaterialien NICHT unterstützt. Das bedeutet, dass in einem Mikrobiologiebefund '''nur genau ein Untersuchungsmaterial''' dokumentiert werden darf. Falls mehrere Untersuchungsmaterialien zu einem Auftrag eingesendet wurden, müssen diese auf mehrere Befunde aufgeteilt werden! Die Information kann in der Section Befundbewertung festgehalten werden. |
| | + | |
| | + | ''Hintergrund: Die maschinenlesbare Angabe der Abhängigkeiten der Untersuchungsmaterialien und der zugehörigen Analysen würde sowohl für Dokumentenersteller als auch für die Nutzer der maschinenlesbaren Daten überaus komplex und daher zu fehleranfällig werden.'' |
| | + | |
| | + | Das für die Codierung des Untersuchungsmaterials erforderliche Template "[[#Specimen Collection|Specimen Collection]]" kann entweder unter dem Template "[[#Probeninformation (Specimen Entry)|Probeninformation (Specimen Entry)]]" oder dem Template "[[#Laboratory Report Data Processing Entry|Laboratory Report Data Processing Entry]]" platziert werden. |
| | + | {{EndYellowBox}} |
| | {| class="wikitable" width="100%" | | {| class="wikitable" width="100%" |
| − | ! colspan="4" |Sektion bzw. Entry | + | ! colspan="4" |Section bzw. Entry |
| − | !OID | + | !Template ID |
| | !Kard/Konf | | !Kard/Konf |
| | !Kapitel | | !Kapitel |
| Zeile 971: |
Zeile 1.398: |
| | | 1.2.40.0.34.6.0.11.2.69 | | | 1.2.40.0.34.6.0.11.2.69 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Brieftext|Link]] | + | | [[#Brieftext|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="7" | Der "Überweisungsgrund" kann codiert oder uncodiert angegeben werden: |
| | |- | | |- |
| − | | colspan="4" |Überweisungsgrund - optional codiert | + | | |
| | + | | colspan="3" |Überweisungsgrund - codiert |
| | | 1.2.40.0.34.6.0.11.2.6 | | | 1.2.40.0.34.6.0.11.2.6 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Überweisungsgrund - optional codiert|Link]] | + | | [[#Überweisungsgrund - codiert|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Konsultationsgrund Problem Concern Entry |
| | + | | 1.2.40.0.34.6.0.11.3.30 |
| | + | | 1..* M |
| | + | | [[#Konsultationsgrund Problem Concern Entry|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" |Überweisungsgrund - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.114 |
| | + | | 0..1 O |
| | + | | [[#Überweisungsgrund - uncodiert|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="7" | Die "Anamnese - Labor und Mikrobiologie" kann codiert oder uncodiert angegeben werden: |
| | + | |- |
| | + | | |
| | + | | colspan="3" | Anamnese - Labor und Mikrobiologie - codiert |
| | + | | 1.2.40.0.34.6.0.11.2.109 |
| | + | | 0..1 O |
| | + | | [[#Anamnese - Labor und Mikrobiologie - codiert|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Anamnese Entry - Labor und Mikrobiologie |
| | + | | 1.2.40.0.34.6.0.11.3.12 |
| | + | | 1..* M |
| | + | | [[#Anamnese Entry - Labor und Mikrobiologie|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | |
| | + | | Anamnese Observation - Labor und Mikrobiologie |
| | + | | 1.2.40.0.34.6.0.11.3.164 |
| | + | | 1..* M |
| | + | | [[#Anamnese Observation - Labor und Mikrobiologie|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" | Anamnese - Labor und Mikrobiologie - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.111 |
| | + | | 0..1 O |
| | + | | [[#Anamnese - Labor und Mikrobiologie - uncodiert|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="7" | Die "Angeforderte Untersuchung" kann codiert oder uncodiert angegeben werden: |
| | |- | | |- |
| − | | colspan="4" |Angeforderte Untersuchungen - optional codiert | + | | |
| | + | | colspan="3" |Angeforderte Untersuchungen - codiert |
| | | 1.2.40.0.34.6.0.11.2.15 | | | 1.2.40.0.34.6.0.11.2.15 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Angeforderte Untersuchungen - optional codiert|Link]] | + | | [[#Angeforderte Untersuchungen - codiert|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Angeforderte Untersuchung Entry |
| | + | | 1.2.40.0.34.6.0.11.3.169 |
| | + | | 1..* M |
| | + | | [[#Angeforderte Untersuchung Entry|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" |Angeforderte Untersuchungen - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.112 |
| | + | | 0..1 O |
| | + | | [[#Angeforderte Untersuchungen - uncodiert|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" |Probeninformation (Specimen Section) | | | colspan="4" |Probeninformation (Specimen Section) |
| | | 1.2.40.0.34.6.0.11.2.93 | | | 1.2.40.0.34.6.0.11.2.93 |
| − | | 0..1 C | + | | 0..1 C<sup>1</sup> |
| − | | [[#Probeninformation (Specimen Section)|Link]] | + | | [[#Probeninformation (Specimen Section)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 992: |
Zeile 1.480: |
| | | 1.2.40.0.34.6.0.11.3.160 | | | 1.2.40.0.34.6.0.11.3.160 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Probeninformation (Specimen Entry)|Link]] | + | | [[#Probeninformation (Specimen Entry)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 999: |
Zeile 1.487: |
| | | 1.2.40.0.34.6.0.11.3.161 | | | 1.2.40.0.34.6.0.11.3.161 |
| | | 1..* M | | | 1..* M |
| − | | + | | [[#Specimen Collection|Template-Spezifikation]] |
| − | '''Im Mikrobiologiebefund MUSS genau eine "Specimen Collection" vorkommen.'''
| |
| − | | [[#Specimen Collection|Link]] | |
| | |- | | |- |
| | | | | | |
| Zeile 1.009: |
Zeile 1.495: |
| | | 1.2.40.0.34.6.0.11.3.162 | | | 1.2.40.0.34.6.0.11.3.162 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Specimen Received|Link]] | + | | [[#Specimen Received|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="7" | Von den folgenden "Laboratory Specialty Sections" '''MUSS''' zumindest eine vorkommen. | | | colspan="7" | Von den folgenden "Laboratory Specialty Sections" '''MUSS''' zumindest eine vorkommen. |
| | |- | | |- |
| | | | | | |
| − | | colspan="3" |Laboratory Specialty Section (Mikroskopie) | + | | colspan="6" | Die "Mikroskopie" kann codiert oder uncodiert angegeben werden: |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Laboratory Specialty Section (Mikroskopie) - codiert |
| | | 1.2.40.0.34.6.0.11.2.105 | | | 1.2.40.0.34.6.0.11.2.105 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Laboratory Specialty Section (Mikroskopie)|Link]] | + | | [[#Laboratory Specialty Section (Mikroskopie) - codiert|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| | | | | | |
| − | | colspan="2" | Laboratory Report Data Processing Entry | + | | |
| | + | | Laboratory Report Data Processing Entry |
| | | 1.2.40.0.34.6.0.11.3.25 | | | 1.2.40.0.34.6.0.11.3.25 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur]] | + | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur des Laboratory Report Data Processing Entries]] |
| | + | |- |
| | + | | |
| | + | | |
| | + | | colspan="2" |Laboratory Specialty Section (Mikroskopie) - uncodiert |
| | + | | 1.2.40.0.34.6.0.11.2.113 |
| | + | | 0..1 O |
| | + | | [[#Laboratory Specialty Section (Mikroskopie) - uncodiert|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.030: |
Zeile 1.528: |
| | | 1.2.40.0.34.6.0.11.2.106 | | | 1.2.40.0.34.6.0.11.2.106 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Laboratory Specialty Section (Kultureller Erregernachweis)|Link]] | + | | [[#Laboratory Specialty Section (Kultureller Erregernachweis)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.037: |
Zeile 1.535: |
| | | 1.2.40.0.34.6.0.11.3.25 | | | 1.2.40.0.34.6.0.11.3.25 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur]] | + | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur des Laboratory Report Data Processing Entries]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.043: |
Zeile 1.541: |
| | | 1.2.40.0.34.6.0.11.2.107 | | | 1.2.40.0.34.6.0.11.2.107 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Laboratory Specialty Section (Molekularer Erregernachweis)|Link]] | + | | [[#Laboratory Specialty Section (Molekularer Erregernachweis)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.050: |
Zeile 1.548: |
| | | 1.2.40.0.34.6.0.11.3.25 | | | 1.2.40.0.34.6.0.11.3.25 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur]] | + | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur des Laboratory Report Data Processing Entries]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.056: |
Zeile 1.554: |
| | | 1.2.40.0.34.6.0.11.2.108 | | | 1.2.40.0.34.6.0.11.2.108 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Laboratory Specialty Section (Infektionsserologie)|Link]] | + | | [[#Laboratory Specialty Section (Infektionsserologie)|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.063: |
Zeile 1.561: |
| | | 1.2.40.0.34.6.0.11.3.25 | | | 1.2.40.0.34.6.0.11.3.25 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur]] | + | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur des Laboratory Report Data Processing Entries]] |
| | + | |- |
| | + | | colspan="4" | Laboratory Specialty Section (Weitere Analysen) |
| | + | | 1.2.40.0.34.6.0.11.2.110 |
| | + | | 0..1 O |
| | + | | [[#Laboratory Specialty Section (Weitere Analysen)|Template-Spezifikation]] |
| | + | |- |
| | + | | |
| | + | | colspan="3" | Laboratory Report Data Processing Entry |
| | + | | 1.2.40.0.34.6.0.11.3.25 |
| | + | | 1..1 M |
| | + | | [[#Struktur: Laboratory Report Data Processing Entry|Struktur des Laboratory Report Data Processing Entries]] |
| | |- | | |- |
| | | colspan="4" | Befundbewertung | | | colspan="4" | Befundbewertung |
| | | 1.2.40.0.34.6.0.11.2.103 | | | 1.2.40.0.34.6.0.11.2.103 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Befundbewertung|Link]] | + | | [[#Befundbewertung|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.074: |
Zeile 1.583: |
| | | 1.2.40.0.34.6.0.11.3.11 | | | 1.2.40.0.34.6.0.11.3.11 |
| | | 1..1 R | | | 1..1 R |
| − | | [[#Comment Entry|Link]] | + | | [[#Comment Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" | Beilagen | | | colspan="4" | Beilagen |
| | | 1.2.40.0.34.6.0.11.2.71 | | | 1.2.40.0.34.6.0.11.2.71 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Beilagen|Link]] | + | | [[#Beilagen|Template-Spezifikation]] |
| | |- | | |- |
| | | colspan="4" | Abschließende Bemerkung | | | colspan="4" | Abschließende Bemerkung |
| | | 1.2.40.0.34.6.0.11.2.70 | | | 1.2.40.0.34.6.0.11.2.70 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Abschließende Bemerkung|Link]] | + | | [[#Abschließende Bemerkung|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="7" | <sup>1</sup> Die Dokumentation des Untersuchungsmaterials kann auf zwei Arten erfolgen: |
| | + | * Enthält ein Befund nur einen Befundbereich ("Laboratory Specialty Section"), so kann die Codierung innerhalb der einen Section erfolgen. |
| | + | '''ODER''' |
| | + | * Bei Verwendung von mehreren Befundbereichen in einem Befund kann es zu Überschneidungen der Untersuchungsmaterialien kommen (ein spezielles Untersuchungsmaterial kann in zwei Befundbereichen analysiert werden). Die CDA Level 3 Codierung eines Untersuchungsmaterials darf jedoch nur einmal im gesamten Befund erfolgen. Daher sind die Informationen zu den Untersuchungsmaterialien in einer eigenen, führenden "Probeninformation (Specimen Section)" zu codieren. |
| | + | |
| | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Bodys des Mikrobiologiebefundes</ref>:''Übersichtstabelle der CDA Strukturen des Bodys des Mikrobiologiebefundes'' | | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Bodys des Mikrobiologiebefundes</ref>:''Übersichtstabelle der CDA Strukturen des Bodys des Mikrobiologiebefundes'' |
| | | | |
| Zeile 1.090: |
Zeile 1.605: |
| | | | |
| | {| class="wikitable" width="100%" | | {| class="wikitable" width="100%" |
| − | ! colspan="3" |Sektion bzw. Entry | + | ! colspan="3" |Section bzw. Entry |
| − | !OID | + | !Template ID |
| | !Kard/Konf | | !Kard/Konf |
| | !Kapitel | | !Kapitel |
| Zeile 1.098: |
Zeile 1.613: |
| | | 1.2.40.0.34.6.0.11.3.25 | | | 1.2.40.0.34.6.0.11.3.25 |
| | | 1..1 M | | | 1..1 M |
| − | | [[#Laboratory Report Data Processing Entry|Link]] | + | | [[#Laboratory Report Data Processing Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.106: |
Zeile 1.621: |
| | | colspan="2" | Specimen Collection | | | colspan="2" | Specimen Collection |
| | | 1.2.40.0.34.6.0.11.3.161 | | | 1.2.40.0.34.6.0.11.3.161 |
| − | | 0..* C | + | | 0..* C<sup>1</sup> |
| − | | + | | [[#Specimen Collection|Template-Spezifikation]] |
| − | '''Im Mikrobiologiebefund MUSS genau eine "Specimen Collection" vorkommen.'''
| |
| − | | [[#Specimen Collection|Link]] | |
| | |- | | |- |
| | | | | | |
| Zeile 1.116: |
Zeile 1.629: |
| | | 1.2.40.0.34.6.0.11.3.162 | | | 1.2.40.0.34.6.0.11.3.162 |
| | | 0..1 O | | | 0..1 O |
| − | | [[#Specimen Received|Link]] | + | | [[#Specimen Received|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| | | colspan="2" | Notification Organizer | | | colspan="2" | Notification Organizer |
| | | 1.2.40.0.34.6.0.11.3.165 | | | 1.2.40.0.34.6.0.11.3.165 |
| − | | 0..* O | + | | 0..1 O |
| − | | [[#Notification Organizer|Link]] | + | | [[#Notification Organizer|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.133: |
Zeile 1.646: |
| | | 1.2.40.0.34.6.0.11.3.166 | | | 1.2.40.0.34.6.0.11.3.166 |
| | | 0..* O | | | 0..* O |
| − | | [[#Notifiable Condition|Link]] | + | | [[#Notifiable Condition|Template-Spezifikation]] |
| − | |-
| |
| − | |
| |
| − | |
| |
| − | | Case Identification [TODO https://jira-neu.elga.gv.at/browse/ELGA-1011]
| |
| − | |
| |
| − | | 0..* O
| |
| − | |
| |
| | |- | | |- |
| | | | | | |
| | | | | | |
| − | | Outbreak Identification [TODO https://jira-neu.elga.gv.at/browse/ELGA-1011] | + | | Case Identification |
| − | |
| + | | 1.2.40.0.34.6.0.11.3.170 |
| | | 0..* O | | | 0..* O |
| − | | | + | | [[#Case Identification|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.153: |
Zeile 1.659: |
| | | 1.2.40.0.34.6.0.11.3.167 | | | 1.2.40.0.34.6.0.11.3.167 |
| | | 0..* O | | | 0..* O |
| − | | [[#Laboratory Isolate Organizer|Link]] | + | | [[#Laboratory Isolate Organizer|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.164: |
Zeile 1.670: |
| | | 1.2.40.0.34.6.0.11.3.26 | | | 1.2.40.0.34.6.0.11.3.26 |
| | | 0..* O | | | 0..* O |
| − | | [[#Laboratory Battery Organizer|Link]] | + | | [[#Laboratory Battery Organizer|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.171: |
Zeile 1.677: |
| | | 1.2.40.0.34.6.0.11.3.27 | | | 1.2.40.0.34.6.0.11.3.27 |
| | | 0..* O | | | 0..* O |
| − | | [[#Laboratory Observation|Link]] | + | | [[#Laboratory Observation|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.178: |
Zeile 1.684: |
| | | 1.2.40.0.34.6.0.11.3.19 | | | 1.2.40.0.34.6.0.11.3.19 |
| | | 0..* O | | | 0..* O |
| − | | [[#Eingebettetes Objekt Entry|Link]] | + | | [[#Eingebettetes Objekt Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.185: |
Zeile 1.691: |
| | | 1.2.40.0.34.6.0.11.3.11 | | | 1.2.40.0.34.6.0.11.3.11 |
| | | 0..* O | | | 0..* O |
| − | | [[#Comment Entry|Link]] | + | | [[#Comment Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.191: |
Zeile 1.697: |
| | | 1.2.40.0.34.6.0.11.3.26 | | | 1.2.40.0.34.6.0.11.3.26 |
| | | 0..* O | | | 0..* O |
| − | | [[#Laboratory Battery Organizer|Link]] | + | | [[#Laboratory Battery Organizer|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.198: |
Zeile 1.704: |
| | | 1.2.40.0.34.6.0.11.3.27 | | | 1.2.40.0.34.6.0.11.3.27 |
| | | 0..* O | | | 0..* O |
| − | | [[#Laboratory Observation|Link]] | + | | [[#Laboratory Observation|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.205: |
Zeile 1.711: |
| | | 1.2.40.0.34.6.0.11.3.19 | | | 1.2.40.0.34.6.0.11.3.19 |
| | | 0..* O | | | 0..* O |
| − | | [[#Eingebettetes Objekt Entry|Link]] | + | | [[#Eingebettetes Objekt Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.212: |
Zeile 1.718: |
| | | 1.2.40.0.34.6.0.11.3.11 | | | 1.2.40.0.34.6.0.11.3.11 |
| | | 0..* O | | | 0..* O |
| − | | [[#Comment Entry|Link]] | + | | [[#Comment Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.218: |
Zeile 1.724: |
| | | 1.2.40.0.34.6.0.11.3.27 | | | 1.2.40.0.34.6.0.11.3.27 |
| | | 0..* O | | | 0..* O |
| − | | [[#Laboratory Observation|Link]] | + | | [[#Laboratory Observation|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.225: |
Zeile 1.731: |
| | | 1.2.40.0.34.6.0.11.3.11 | | | 1.2.40.0.34.6.0.11.3.11 |
| | | 0..* O | | | 0..* O |
| − | | [[#Comment Entry|Link]] | + | | [[#Comment Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.231: |
Zeile 1.737: |
| | | 1.2.40.0.34.6.0.11.3.19 | | | 1.2.40.0.34.6.0.11.3.19 |
| | | 0..* O | | | 0..* O |
| − | | [[#Eingebettetes Objekt Entry|Link]] | + | | [[#Eingebettetes Objekt Entry|Template-Spezifikation]] |
| | |- | | |- |
| | | | | | |
| Zeile 1.237: |
Zeile 1.743: |
| | | 1.2.40.0.34.6.0.11.3.11 | | | 1.2.40.0.34.6.0.11.3.11 |
| | | 0..* O | | | 0..* O |
| − | | [[#Comment Entry|Link]] | + | | [[#Comment Entry|Template-Spezifikation]] |
| | + | |- |
| | + | | colspan="6" | <sup>1</sup> Die Verwendung von "Specimen Collection" innerhalb des "Laboratory Report Data Processing Entry" ist '''NUR''' dann zulässig, wenn '''NUR EIN''' Befundbereich ("Laboratory Specialty Section") im gesamten Befund vorkommt und die "Specimen Collection" nicht schon als Teil der Section "Probeninformation (Specimen Section)" codiert wurde. |
| | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Laboratory Report Data Processing Entries</ref>:''Übersichtstabelle der CDA Strukturen des Laboratory Report Data Processing Entries'' | | |}<ref group="Tabelle">Übersichtstabelle der CDA Strukturen des Laboratory Report Data Processing Entries</ref>:''Übersichtstabelle der CDA Strukturen des Laboratory Report Data Processing Entries'' |
| | | | |
| Zeile 1.248: |
Zeile 1.756: |
| | | | |
| | Der vorliegende Leitfaden baut auf den Definitionen des "IHE Pathology and Laboratory Medicine (PALM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019"<ref name="IHEPaLMTF3">IHE Pathology and Laboratory Medicine (PALM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019 [https://www.ihe.net/uploadedFiles/Documents/PaLM/IHE_PaLM_TF_Vol3.pdf]</ref> auf, welche durch diesen Leitfaden weiter eingeschränkt werden. Dadurch erhalten die entsprechenden Templates ihre Gültigkeit und sind aus Konformitätsgründen bei Komponenten, welche über eine entsprechende Definition verfügen, auch anzugeben. | | Der vorliegende Leitfaden baut auf den Definitionen des "IHE Pathology and Laboratory Medicine (PALM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019"<ref name="IHEPaLMTF3">IHE Pathology and Laboratory Medicine (PALM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019 [https://www.ihe.net/uploadedFiles/Documents/PaLM/IHE_PaLM_TF_Vol3.pdf]</ref> auf, welche durch diesen Leitfaden weiter eingeschränkt werden. Dadurch erhalten die entsprechenden Templates ihre Gültigkeit und sind aus Konformitätsgründen bei Komponenten, welche über eine entsprechende Definition verfügen, auch anzugeben. |
| − | {{BeginYellowBox}} | + | {{BeginYellowBox}}Entsprechend den Vorgaben des IHE Frameworks für Labor sind für '''Personen und Organisationen''' die Angabe eines '''Namens''' ("name"-Element), einer '''Adresse''' ("addr"-Element) und einer '''Telekom'''-Verbindung ("telecom"-Element) '''verpflichtend'''. Diese können jedoch mit einem '''nullFlavor''' versehen werden. |
| − | Entsprechend den Vorgaben des IHE Frameworks für Labor sind für '''Personen und Organisationen''' die Angabe eines '''Namens''' (name-Element), einer '''Adresse''' (addr-Element) und einer '''Telekom'''-Verbindung (telecom-Element) '''verpflichtend'''. Diese können jedoch mit einem '''nullFlavor''' versehen werden. | |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| | | | |
| Zeile 1.263: |
Zeile 1.770: |
| | | | |
| | ===Header Level Templates=== | | ===Header Level Templates=== |
| − | {{BeginILFBox}}Die Header Level Templates wurden aus dem bestehenden "Allgemeiner Implementierungsleitfaden für ELGA CDA Dokumente" übernommen. Diese sind unter [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Dokumentenstruktur| Allgemeiner Leitfaden - Kapitel Administrative Daten (CDA Header) - Dokumentenstruktur]] zu finden.{{EndILFBox}} | + | {{BeginILFBox}}Die Header Level Templates wurden aus dem bestehenden "Allgemeiner Implementierungsleitfaden für ELGA CDA Dokumente" übernommen. Diese sind unter [[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Dokumentenstruktur| Allgemeiner Leitfaden - Kapitel Administrative Daten (CDA Header) - Dokumentenstruktur]] zu finden. |
| − | {{BeginYellowBox}} | + | {{EndILFBox}} |
| − | ''Wichtiger Hinweis:'' Header-Element welche spezifisch für den Labor- und Mikrobiologiebefund angepasst wurden oder für die es spezielle Einschränkungen in Form von Asserts gibt, sind grundsätzlich der Spezifikation im Kapitel [[#Document_Level_Templates|Document Level Templates]] zu entnehmen. | + | {{BeginYellowBox}}''Wichtiger Hinweis:'' Header-Elemente welche spezifisch für den Labor- und Mikrobiologiebefund angepasst wurden oder für die es spezielle Einschränkungen in Form von Asserts gibt, sind grundsätzlich der Spezifikation im Kapitel [[#Document_Level_Templates|Document Level Templates]] zu entnehmen. |
| | | | |
| | Diese angepassten bzw. betroffenen Elemente umfassen: | | Diese angepassten bzw. betroffenen Elemente umfassen: |
| − | * ClincialDocument/templateId | + | * /ClinicalDocument/templateId |
| − | * ClincialDocument/code | + | * /ClinicalDocument/code |
| − | * ClinicalDocument/title | + | * /ClinicalDocument/title |
| − | * ClinicalDocument/formatCode | + | * /ClinicalDocument/sdtc:statusCode |
| − | * ClinicalDocument/author | + | * /ClinicalDocument/formatCode |
| − | * ClinicalDocument/authenticator | + | * /ClinicalDocument/author |
| − | * ClinicalDocument/componentOf/encompassingEncounter
| |
| | | | |
| − | Ebenso wurden folgende Header-Elemente angepasst und werden in diesem Kapitel detailliert dargestellt: | + | Ebenso wurden folgende Header-Elemente speziell für den Labor- und Mikrobiologiebefund erstellt und werden in diesem Kapitel noch einmal detailliert dargestellt: |
| − | * ClinicalDocument/participant[@typeCode='REF'] (Participant Auftraggeber / Ordering Provider) | + | * [[#Laboratory Results Validator|/ClinicalDocument/authenticator (Laboratory Results Validator)]] |
| − | * ClinicalDocument/documentationOf/serviceEvent | + | * [[#Participant Auftraggeber / Ordering Provider|/ClinicalDocument/participant[@typeCode='REF'] (Participant Auftraggeber / Ordering Provider)]] |
| | + | * [[#Documentation Of Service Event - Labor und Mikrobiologie|/ClinicalDocument/documentationOf/serviceEvent (Documentation Of Service Event - Labor und Mikrobiologie)]] |
| | + | * [[#Component Of - Encompassing Encounter with id|/ClinicalDocument/componentOf/encompassingEncounter (Component Of - Encompassing Encounter with id)]] |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| | + | |
| | + | ====Laboratory Results Validator==== |
| | + | |
| | + | {{:1.2.40.0.34.6.0.11.1.49/dynamic}} |
| | | | |
| | ====Participant Auftraggeber / Ordering Provider==== | | ====Participant Auftraggeber / Ordering Provider==== |
| Zeile 1.285: |
Zeile 1.797: |
| | {{:1.2.40.0.34.6.0.11.1.42/dynamic}} | | {{:1.2.40.0.34.6.0.11.1.42/dynamic}} |
| | | | |
| − | ====Documentation Of Service Event - Labor- und Mikrobiologie==== | + | ====Documentation Of Service Event - Labor und Mikrobiologie==== |
| | | | |
| | {{:1.2.40.0.34.6.0.11.1.48/dynamic}} | | {{:1.2.40.0.34.6.0.11.1.48/dynamic}} |
| | + | |
| | + | ====Component Of - Encompassing Encounter with id==== |
| | + | |
| | + | {{:1.2.40.0.34.6.0.11.1.50/dynamic}} |
| | | | |
| | ===Section Level Templates=== | | ===Section Level Templates=== |
| | | | |
| − | ====Brieftext==== | + | ====Überweisungsgrund - codiert==== |
| − | {{:1.2.40.0.34.6.0.11.2.69/dynamic}} | + | {{:1.2.40.0.34.6.0.11.2.6/dynamic}} |
| | + | |
| | + | ====Überweisungsgrund - uncodiert==== |
| | + | {{:1.2.40.0.34.6.0.11.2.114/dynamic}} |
| | + | |
| | + | ====Anamnese - Labor und Mikrobiologie - codiert==== |
| | + | {{:1.2.40.0.34.6.0.11.2.109/dynamic}} |
| | | | |
| − | ====Überweisungsgrund - optional codiert==== | + | ====Anamnese - Labor und Mikrobiologie - uncodiert==== |
| − | {{:1.2.40.0.34.6.0.11.2.6/dynamic}} | + | {{:1.2.40.0.34.6.0.11.2.111/dynamic}} |
| | | | |
| − | ====Angeforderte Untersuchungen - optional codiert==== | + | ====Angeforderte Untersuchungen - codiert==== |
| | {{:1.2.40.0.34.6.0.11.2.15/dynamic}} | | {{:1.2.40.0.34.6.0.11.2.15/dynamic}} |
| | + | |
| | + | ====Angeforderte Untersuchungen - uncodiert==== |
| | + | {{:1.2.40.0.34.6.0.11.2.112/dynamic}} |
| | | | |
| | ====Probeninformation (Specimen Section)==== | | ====Probeninformation (Specimen Section)==== |
| Zeile 1.306: |
Zeile 1.831: |
| | {{:1.2.40.0.34.6.0.11.2.102/dynamic}} | | {{:1.2.40.0.34.6.0.11.2.102/dynamic}} |
| | | | |
| − | ====Laboratory Specialty Section (Mikroskopie)==== | + | ====Laboratory Specialty Section (Mikroskopie) - codiert==== |
| | {{:1.2.40.0.34.6.0.11.2.105/dynamic}} | | {{:1.2.40.0.34.6.0.11.2.105/dynamic}} |
| | + | |
| | + | ====Laboratory Specialty Section (Mikroskopie) - uncodiert==== |
| | + | {{:1.2.40.0.34.6.0.11.2.113/dynamic}} |
| | | | |
| | ====Laboratory Specialty Section (Kultureller Erregernachweis)==== | | ====Laboratory Specialty Section (Kultureller Erregernachweis)==== |
| Zeile 1.317: |
Zeile 1.845: |
| | ====Laboratory Specialty Section (Infektionsserologie)==== | | ====Laboratory Specialty Section (Infektionsserologie)==== |
| | {{:1.2.40.0.34.6.0.11.2.108/dynamic}} | | {{:1.2.40.0.34.6.0.11.2.108/dynamic}} |
| | + | |
| | + | ====Laboratory Specialty Section (Weitere Analysen)==== |
| | + | {{:1.2.40.0.34.6.0.11.2.110/dynamic}} |
| | | | |
| | ====Befundbewertung==== | | ====Befundbewertung==== |
| | {{:1.2.40.0.34.6.0.11.2.103/dynamic}} | | {{:1.2.40.0.34.6.0.11.2.103/dynamic}} |
| | + | |
| | + | ====Brieftext==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Brieftext'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Brieftext|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | | | |
| | ====Beilagen==== | | ====Beilagen==== |
| − | {{:1.2.40.0.34.6.0.11.2.71/dynamic}} | + | {{BeginILFBox}}Die Spezifikation des "'''Beilagen'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Beilagen|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | {{BeginYellowBox}}ELGA Labor- und Mikrobiologiebefunde können alle Kriterien erfüllen, die für Befundberichte von der Akkreditierung für medizinische Laboratorien (ISO 15189:2012) gefordert sind. '''Ein Anhängen des PDF-Befundes ist NICHT erforderlich!''' |
| | + | |
| | + | Für die vollständige Erfüllung aller Akkreditierungsanforderungen an Befundberichte ist das erstellende Labor verantwortlich; die Akkreditierung wird von der Abt. Akkreditierung Austria im Bundesministerium für Digitalisierung und Wirtschaftsstandort durchgeführt. |
| | + | |
| | + | Mit Rücksicht auf eine einfache Verwendbarkeit der Befunde durch die Benutzer (z.B. niedergelassene Ärzte), die häufig mit einer großen Anzahl von Laborbefunden eines Patienten konfrontiert sind, ist eine Duplizierung der Daten durch Anhängen einer PDF-Ansicht daher '''NICHT gestattet'''. |
| | + | {{EndYellowBox}} |
| | | | |
| | ====Abschließende Bemerkung==== | | ====Abschließende Bemerkung==== |
| − | {{:1.2.40.0.34.6.0.11.2.70/dynamic}} | + | {{BeginILFBox}}Die Spezifikation des "'''Abschließende Bemerkung'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Abschlie.C3.9Fende_Bemerkung|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Übersetzung==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Übersetzung'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#.C3.9Cbersetzung|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ===Entry Level Templates=== |
| | + | |
| | + | ====Konsultationsgrund Problem Concern Entry==== |
| | + | {{:1.2.40.0.34.6.0.11.3.30/dynamic}} |
| | + | |
| | + | ====Konsultationsgrund Problem Entry==== |
| | + | {{:1.2.40.0.34.6.0.11.3.31/dynamic}} |
| | + | |
| | + | ====Criticality Observation==== |
| | + | {{:1.2.40.0.34.6.0.11.3.35/dynamic}} |
| | + | |
| | + | ====Anamnese Entry - Labor und Mikrobiologie==== |
| | + | {{:1.2.40.0.34.6.0.11.3.12/dynamic}} |
| | + | |
| | + | ====Anamnese Observation - Labor und Mikrobiologie==== |
| | + | {{:1.2.40.0.34.6.0.11.3.164/dynamic}} |
| | | | |
| − | ===Entry Level Template=== | + | ====Angeforderte Untersuchung Entry==== |
| | + | {{:1.2.40.0.34.6.0.11.3.169/dynamic}} |
| | | | |
| | ====Probeninformation (Specimen Entry)==== | | ====Probeninformation (Specimen Entry)==== |
| Zeile 1.346: |
Zeile 1.911: |
| | ====Notifiable Condition==== | | ====Notifiable Condition==== |
| | {{:1.2.40.0.34.6.0.11.3.166/dynamic}} | | {{:1.2.40.0.34.6.0.11.3.166/dynamic}} |
| | + | |
| | + | ====Case Identification==== |
| | + | {{:1.2.40.0.34.6.0.11.3.170/dynamic}} |
| | | | |
| | ====Laboratory Isolate Organizer==== | | ====Laboratory Isolate Organizer==== |
| Zeile 1.353: |
Zeile 1.921: |
| | {{:1.2.40.0.34.6.0.11.3.26/dynamic}} | | {{:1.2.40.0.34.6.0.11.3.26/dynamic}} |
| | | | |
| − | ====Laboratory Observation Entry==== | + | ====Laboratory Observation==== |
| | {{:1.2.40.0.34.6.0.11.3.27/dynamic}} | | {{:1.2.40.0.34.6.0.11.3.27/dynamic}} |
| | | | |
| − | ====Eingebettetes Objekt Entry==== | + | ====Logo Entry==== |
| − | {{:1.2.40.0.34.6.0.11.3.19/dynamic}} | + | {{BeginILFBox}}Die Spezifikation des "'''Logo Entry'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Logo_Entry|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Certainty Observation==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Certainty Observation'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Certainty_Observation|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Severity Observation==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Severity Observation'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Severity_Observation|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Problem Status Observation==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Problem Status Observation'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Problem_Status_Observation|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Laterality Qualifier==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Laterality Qualifier'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Laterality_Qualifier|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | | | |
| | ====Comment Entry==== | | ====Comment Entry==== |
| − | {{:1.2.40.0.34.6.0.11.3.11/dynamic}} | + | {{BeginILFBox}}Die Spezifikation des "'''Comment Entry'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Comment_Entry|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====External Document Entry==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''External Document Entry'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#External_Document_Entry|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Eingebettetes Objekt Entry==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Eingebettetes Objekt Entry'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Eingebettetes_Objekt_Entry|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | | | |
| | ===Weitere CDA Fragmente === | | ===Weitere CDA Fragmente === |
| − | {{BeginILFBox}} | + | |
| − | Die weiteren CDA Fragmente, oder auch Compilation Templates genannt, wurden aus dem bestehenden "Allgemeinen Implementierungsleitfaden für ELGA CDA Dokumente" übernommen. Diese sind auch unter [[ILF:Allgemeiner_Implementierungsleitfaden_2020#Sonstige_Templates_.28Fragmente.29 | Allgemeiner Leitfaden - Kapitel Sonstige Templates (Fragmente)]] zu finden. | + | ====Laboratory Observation Value==== |
| | + | {{:1.2.40.0.34.6.0.11.9.23/dynamic}} |
| | + | |
| | + | ====Performer - Laboratory==== |
| | + | {{:1.2.40.0.34.6.0.11.9.24/dynamic}} |
| | + | |
| | + | ====Address Compilation==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Address Compilation'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Address_Compilation|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Address Compilation Minimal==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Address Compilation Minimal'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Address_Compilation_Minimal|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Assigned Entity==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Assigned Entity'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Assigned_Entity|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Assigned Entity Body==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Assigned Entity Body'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Assigned_Entity_Body|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Assigned Entity Body with name, addr and telecom==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Assigned Entity Body with name, addr and telecom'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Assigned_Entity_Body_with_name.2C_addr_and_telecom|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Assigned Entity with id, name, addr and telecom==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Assigned Entity with id, name, addr and telecom'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Assigned_Entity_with_id.2C_name.2C_addr_and_telecom|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Author Body==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Author Body'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Author_Body|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Device Compilation==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Device Compilation'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Device_Compilation|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Informant Body==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Informant Body'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Informant_Body|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Narrative Text Reference==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Narrative Text Reference'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Narrative_Text_Reference|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Organization Compilation with id, name==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Organization Compilation with id, name'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Organization_Compilation_with_id.2C_name|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Organization Compilation with name==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Organization Compilation with name'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Organization_Compilation_with_name|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Organization Compilation with name, addr minimal==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Organization Compilation with name, addr minimal'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Organization_Compilation_with_name.2C_addr_minimal|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Organization Compilation with name, addr minimal and telecom==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Organization Compilation with name, addr minimal and telecom'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Organization_Compilation_with_name.2C_addr_minimal_and_telecom|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Organization Name Compilation==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Organization Name Compilation'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Organization_Name_Compilation|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Original Text Reference==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Original Text Reference'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Original_Text_Reference|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Participant Body==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Participant Body'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Participant_Body|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Performer Body==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Performer Body'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Performer_Body|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Person Name Compilation G1 M==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Person Name Compilation G1 M'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Person_Name_Compilation_G1_M|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Person Name Compilation G2==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Person Name Compilation G2'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Person_Name_Compilation_G2|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | |
| | + | ====Person Name Compilation G2 M==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Person Name Compilation G2 M'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Person_Name_Compilation_G2_M|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | {{EndILFBox}} | | {{EndILFBox}} |
| | + | |
| | + | ====Time Interval Information minimal==== |
| | + | {{BeginILFBox}}Die Spezifikation des "'''Time Interval Information minimal'''"-Templates ist dem '''[[ILF:Allgemeiner_Implementierungsleitfaden_(Version_3)#Time_Interval_Information_minimal|Allgemeinen Implementierungsleitfaden (Version 3)]]''' zu entnehmen. |
| | + | {{EndILFBox}} |
| | + | </div> |
| | | | |
| | ==Terminologien== | | ==Terminologien== |
| | Die erforderlichen Terminologien sind im Folgenden aufgelistet. | | Die erforderlichen Terminologien sind im Folgenden aufgelistet. |
| | | | |
| − | {{BeginYellowBox}} | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.182 atcdabbr_CriticalityObservationValue_VS] |
| − | Falls Werte in den definierten Value-Sets fehlen, dann bitten wir um die Meldung mit einem Vorschlag an [mailto:cda@elga.gv.at cda@elga.gv.at]. | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.10 atcdabbr_LanguageCode] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.211 atcdabbr_LateralityQualifierCode_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.75 atcdabbr_PracticeSetting_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.189 atcdabbr_ProblemSeverity_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.212 atcdabbr_TopographicalModifierQualifier_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.5 ELGA_ActEncounterCode] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.4 ELGA_AdministrativeGender] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.6.0.10.66 ELGA_AnamneseLaborMikrobiologie_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.6.0.10.53 ELGA_Antibiogramm_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.6 ELGA_AuthorSpeciality] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.198 ELGA_ConditionStatusCode] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.184 ELGA_ConditionVerificationStatus] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.52 ELGA_HumanActSite] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.173 ELGA_HumanLanguage] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.29 ELGA_InformationRecipientType] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.9 ELGA_InsuredAssocEntity] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.44 ELGA_Laborparameter] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.47 ELGA_Laborstruktur] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.175 ELGA_LanguageAbilityMode] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.11 ELGA_MaritalStatus] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.42 ELGA_Medientyp] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.188 ELGA_Mikroorganismen_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.186 ELGA_NachweisErgebnis_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.13 ELGA_ObservationInterpretation] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.17 ELGA_PersonalRelationship] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.187 ELGA_Probenmaterial_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.201 ELGA_Problems] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.174 ELGA_ProficiencyLevelCode] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.18 ELGA_ReligiousAffiliation] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.6.0.10.70 ELGA_RequestedProcedures_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.43 ELGA_ServiceEventPerformer] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.22 ELGA_ServiceEventsLabor] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.58 ELGA_SignificantPathogens] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.10.205 ELGA_TypeOfProblem_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.6.0.10.23 elgagab_Art_der_Diagnose_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=1.2.40.0.34.6.0.10.19 epims_meldepflichtigeKrankheiten_VS] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=2.16.840.1.113883.1.11.16040 EntityCode] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=2.16.840.1.113883.1.11.10901 ParticipationType] |
| | + | * [https://art-decor.org/art-decor/decor-valuesets--at-lab-?id=2.16.840.1.113883.1.11.10310 TargetAwareness] |
| | + | {{BeginYellowBox}}Falls Werte in den definierten Value Sets fehlen, dann bitten wir um die Meldung mit einem Vorschlag an [mailto:cda@elga.gv.at cda@elga.gv.at]. |
| | {{EndYellowBox}} | | {{EndYellowBox}} |
| − | </div>
| |
| | | | |
| − | <div class="portrait">
| |
| | =Anhang= | | =Anhang= |
| − | ==Abbildungen== | + | ==Abbildungsverzeichnis== |
| | <references group="Abbildung"/> | | <references group="Abbildung"/> |
| | | | |
| − | ==Tabellen== | + | ==Tabellenverzeichnis== |
| | <references group="Tabelle"/> | | <references group="Tabelle"/> |
| | | | |
| − | ==Referenzen== | + | ==Literatur und Weblinks== |
| − | </div> | + | <references /> |
| | + | |
| | + | ==Release-Log, Ausblick und weitere Informationen== |
| | + | Auf der [[ILF Diskussion:Labor- und Mikrobiologiebefund (Version 3)|Diskussionsseite]] zu diesem Leitfaden können das Release-Log, das einen Überblick über die in diesem Leitfaden implementierten Neuerungen gibt, und der Ausblick auf Änderungen, die in zukünftigen Leitfadenversionen geplant sind, eingesehen werden. Gegebenenfalls werden Inhalte aktueller Diskussionen, bekannte Probleme oder weitere Hinweise aufgeführt. |
Dieses Dokument bildet den
vollständigen Implementierungsleitfaden des Labor- und Mikrobiologiebefundes ab und richtet sich an Softwareentwickler und Berater. Zum besseren Verständnis empfehlen wir Ihnen, den
zusammenfassenden Guide im Vorfeld zu lesen.
1 Zusammenfassung
Der Implementierungsleitfaden "Labor- und Mikrobiologiebefund" beschreibt die Inhalte, die für den Austausch von fertiggestellten und fachärztlich vidierten Befunden, innerhalb und zwischen Einrichtungen des österreichischen Gesundheitswesens, notwendig sind.
Der Leitfaden enthält Festlegungen, Einschränkungen und Bedingungen auf Grundlage des internationalen Standards ISO/HL7 27932:2009 HL7 Clinical Document Architecture, Release 2.0 (CDA) und ist ein nationaler Standard der HL7 Austria.
Die Grundlage der Datenaustauschformate ist der internationale CDA-Standard, der sich in ELGA bewährt hat. Er erlaubt es Sender und Empfänger, sich ohne vorherige Absprache zu verstehen. Der Standard hat zum Ziel, einen umfassenden Austausch von semantisch interoperablen Informationen zwischen allen beteiligten Akteuren bei der Behandlung von Patienten zu ermöglichen. Der Datenaustausch findet hierbei nicht nur innerhalb einer Einrichtung, sondern auch zwischen kooperierenden Einrichtungen und über Sektorengrenzen hinaus statt. Die Empfänger der Dokumente sollen die Inhalte benutzen und weiterverwenden können, ohne sich vorher mit dem Ersteller absprechen zu müssen.
Der "Labor- und Mikrobiologiebefund" basiert auf den Vorgaben des Allgemeinen Implementierungsleitfadens. Darin werden die notwendigen Datentypen, Dokument-Metadaten (Header), die Möglichkeiten der Textstrukturierung, grundlegende Vorgaben für die Anwendung von Terminologien, einige allgemein genutzten Inhaltsstrukturen (Sections) sowie Codebeispiele und praktische Implementierungshilfen gezeigt. Alle weiteren, für diesen Leitfaden benötigten Elemente werden hier erklärt. Die Notation der Spezifikation der Datenaustauschformate folgt der "Art-Decor"-Schreibweise, die auf einer eigenen Seite (Art-Decor-Tabellen verstehen) erläutert wird.
Der vorgesehene Ablauf des Datenaustausches wird im Kapitel Anwendungsfälle / User Stories beschrieben.
Übersichtstabellen für Header und Body-Strukturen
Auf der Diskussionsseite werden die Fehler und Änderungswünsche an dieser Version dokumentiert.
2 Informationen über dieses Dokument
2.1 Impressum
Medieneigentümer, Herausgeber, Hersteller, Verleger:
ELGA GmbH, Treustraße 35-43, Wien, Österreich. Telefon: +43.1.2127050
Internet: www.elga.gv.at
Email: cda@elga.gv.at
Geschäftsführer: DI Dr. Günter Rauchegger, DI(FH) Dr. Franz Leisch
Redaktion, Projektleitung, Koordination:
Mag.Dr. Stefan Sabutsch, stefan.sabutsch@elga.gv.at
Abbildungen: © ELGA GmbH
Nutzung: Das Dokument enthält geistiges Eigentum der Health Level Seven® Int. und HL7® Austria, Erdbergweg 7/8, 8052 Graz; www.hl7.at.
Die Nutzung ist ohne Lizenz- und Nutzungsgebühren zum Zweck der Erstellung medizinischer Dokumente ausdrücklich erlaubt. Andere Arten der Nutzung und auch auszugsweise Wiedergabe bedürfen der Genehmigung des Medieneigentümers.
Download unter www.gesundheit.gv.at und www.elga.gv.at/cda
2.2 Haftungsausschluss
Die Arbeiten für den vorliegenden Leitfaden wurden von den Autoren gemäß dem Stand der Technik und mit größtmöglicher Sorgfalt erbracht und über ein öffentliches Kommentierungsverfahren kontrolliert. Die Nutzung des vorliegenden Leitfadens erfolgt in ausschließlicher Verantwortung der Anwender. Aus der Verwendung des vorliegenden Leitfadens können keinerlei Rechtsansprüche gegen die Autoren, Herausgeber oder Mitwirkenden erhoben und/oder abgeleitet werden. Ein allfälliger Widerspruch zum geltenden Recht ist jedenfalls nicht beabsichtigt und von den Erstellern des Dokumentes nicht gewünscht.
2.3 Sprachliche Gleichbehandlung
Soweit im Text Bezeichnungen nur im generischen Maskulinum angeführt sind, beziehen sie sich auf Männer, Frauen und andere Geschlechtsidentitäten in gleicher Weise. Unter dem Begriff "Patient" werden sowohl Bürger, Kunden und Klienten zusammengefasst, welche an einem Behandlungs- oder Pflegeprozess teilnehmen als auch gesunde Bürger, die derzeit nicht an einem solchen teilnehmen. Es wird ebenso darauf hingewiesen, dass umgekehrt der Begriff Bürger auch Patienten, Kunden und Klienten mit einbezieht.
2.4 Lizenzinformationen
Die von HL7 Austria erarbeiteten Standards und die Bearbeitungen der Standards von HL7 International stellen Werke im Sinne des österreichischen Urheberrechtsgesetzes dar und unterliegen daher urheberrechtlichem Schutz.
HL7 Austria genehmigt die Verwendung dieser Standards für die Zwecke der Erstellung, des Verkaufs und des Betriebs von Computerprogrammen, sofern nicht anders angegeben oder sich die Standards auf andere urheberrechtlich oder lizenzrechtlich geschützte Werke beziehen.
Die vollständige oder teilweise Veröffentlichung der Standards (zum Beispiel in Spezifikationen, Publikationen oder Schulungsunterlagen) ist nur mit einer ausdrücklichen Genehmigung der HL7 Austria gestattet. Mitglieder von HL7 Austria sind berechtigt, die Standards vollständig oder in Auszügen ausschließlich organisationsintern zu publizieren, zu vervielfältigen oder zu verteilen. Die Veröffentlichung eigener Anpassungen der HL7-Spezifikationen (im Sinne von Lokalisierungen) oder eigener Leitfäden erfordert eine formale Vereinbarung mit der HL7 Austria.
HL7® und CDA® sind die eingetragenen Marken von Health Level Seven International. Die vollständigen Lizenzinformationen finden sich unter https://hl7.at/nutzungsbedingungen-und-lizenzinformationen/. Die Lizenzbedingungen von HL7 International finden sich unter http://www.HL7.org/legal/ippolicy.cfm
2.4.1 Urheber- und Nutzungsrechte von anderen Quellen ("Third Party IP")
Third Party Intellectual Property
Der Nutzer dieses Dokuments (bzw. der Lizenznehmer) stimmt zu und erkennt an, dass HL7 Austria nicht alle Rechte und Ansprüche in und an den Materialien besitzt und dass die Materialien geistiges Eigentum von Dritten enthalten und / oder darauf verweisen können ("Third Party Intellectual Property (IP)").
Die Anerkennung dieser Lizenzbestimmungen gewährt dem Lizenznehmer keine Rechte in Bezug auf Third Party IP. Der Lizenznehmer allein ist für die Identifizierung und den Erhalt von notwendigen Lizenzen oder Genehmigungen zur Nutzung von Third Party IP im Zusammenhang mit den Materialien oder anderweitig verantwortlich.
Jegliche Handlungen, Ansprüche oder Klagen eines Dritten, die sich aus einer Verletzung eines Third Party IP-Rechts durch den Lizenznehmer ergeben, bleiben die Haftung des Lizenznehmers.
2.4.2 SNOMED CT
Dieser Leitfaden enthält Material, das durch SNOMED International urheberrechtlich geschützt ist.
Jede Verwendung von SNOMED CT in Österreich erfordert eine aufrechte Affiliate Lizenz oder eine Sublizenz. Die entsprechende Lizenz ist kostenlos, vorausgesetzt die Verwendung findet nur in Österreich statt und erfüllt die Bedingungen des Affiliate License Agreements. Affiliate Lizenzen können über das Member Licensing and Distribution Service (MLDS) direkt beim jeweiligen NRC beantragt werden: MLDS für Österreich.
2.4.3 Weitere Terminologien
Im Folgenden finden Sie eine nicht-exhaustive Liste von weiteren Terminologien, die eine solche separate Lizenz erfordern können:
| Terminologie
|
Eigentümer, Kontaktinformation
|
| Logical Observation Identifiers Names & Codes (LOINC) [1]
|
Regenstrief Institute, Inc. [2]
|
| Unified Code for Units of Measure (UCUM) [3]
|
Regenstrief Institute, Inc. [2]
|
| International Classification of Diseases (ICD) [4]
|
World Health Organization (WHO) [5]
|
| ICD-10 BM*G*[6]
|
Für Gesundheit zuständiges Bundesministerium www.sozialministerium.at
|
| Anatomical Therapeutic Chemical Classification System (ATC) [7]
|
World Health Organization (WHO)[5]
|
| Pharmazentralnummer (PZN)
|
ARGE Pharma im Fachverband der chemischen Industrie Österreichs (FCIO) der Wirtschaftskammern Österreichs (WKO) [8]
|
| EDQM-Codes
|
Europäisches Direktorat für die Qualität von Arzneimitteln [9]
|
| Medical Device Communications (MDC) vom ISO/IEEE 11073 Standard
|
MDC wird als Substandard 10101 "Nomenclature" in "Health informatics - Medical / health device communication standards", kurz 11073, geführt und werden mit einem Copyright bei IEEE SA am österreichischen Termserver bereitgestellt. [10], [11]
|
Die Terminologien werden am österreichischen Terminologieserver zur Verfügung gestellt.
2.5 Verwendete Grundlagen und Bezug zu anderen Standards
Grundlage dieses Implementierungsleitfadens ist der internationale Standard "HL7 Clinical Document Architecture, Release 2.0" (CDA ©), für die das Copyright © von Health Level Seven International[12] gilt. 2009 wurde die Release 2.0 als ISO-Standard ISO/HL7 27932:2009 publiziert[13].
CDA definiert die Struktur und Semantik von "medizinischen Dokumenten" zum Austausch zwischen Gesundheitsdiensteanbietern und Patienten. Es enthält alle Metadaten zur Weiterverarbeitung und einen lesbaren textuellen Inhalt und kann diese Informationen auch maschinenlesbar tragen. Das Datenmodell von CDA und seine Abbildung in XML[14] folgen dem Basisstandard HL7 Version 3[15] mit seinem Referenz-Informationsmodell (RIM). Dieser Leitfaden verwendet das HL7-Template-Austauschformat zur Definition der "Bausteine" (Templates) und ART-DECOR® [16] als Spezifikationsplattform.
- HL7 Clinical Document Architecture (CDA) [17]
- HL7 Referenz-Informationsmodell (RIM)[18]
- HL7 V3 Datentypen [19]
- HL7 Template-Austauschformat Specification and Use of Reusable Information Constraint Templates, Release 1[20]
Die HL7 Standards können über die HL7 Anwendergruppe Österreich (HL7 Austria)[21], die offizielle Vertretung von Health Level Seven International in Österreich bezogen werden (www.HL7.at). Alle auf nationale Verhältnisse angepassten und veröffentlichten HL7-Spezifikationen können ohne Lizenz- und Nutzungsgebühren in jeder Art von Anwendungssoftware verwendet werden.
Für die Modellierung der technischen Spezifikation der Inhalte des Labor- und Mikrobiologiebefundes wurde das "Pathology and Laboratory Medicine (PaLM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019"[22] der Integrating the Healthcare Enterprise (IHE) als wesentliche Grundlage gewählt.
2.6 Verbindlichkeit und rechtliche Grundlagen
Die Verbindlichkeit und die Umsetzungsfrist dieses Leitfadens sind im Gesundheitstelematikgesetz 2012, BGBl.I Nr.111/2012 sowie in den darauf fußenden ELGA-Verordnungen geregelt.
Der Leitfaden in seiner jeweils aktuell gültigen Fassung sowie die aktualisierten Terminologien sind vom zuständigen Minister auf www.gesundheit.gv.at zu veröffentlichen. Der Zeitplan zur Bereitstellung der Datenaustauschformate wird durch das Gesundheitstelematikgesetz 2012 und darauf basierenden Durchführungsverordnungen durch den zuständigen Bundesminister vorgegeben. Hauptversionen, also Aktualisierungen des Implementierungsleitfadens, die verpflichtende Änderungen in den erstellenden Systemen nach sich ziehen, sind mit ihren Fristen zur Bereitstellung per Verordnung kundzumachen. Andere Aktualisierungen (Nebenversionen) dürfen auch ohne Änderung dieser Verordnung unter www.gesundheit.gv.at veröffentlicht werden.
Die Anwendung dieses Implementierungsleitfadens hat im Einklang mit österreichischem und europäischem Recht, insbesondere mit den relevanten Materiengesetzen (z.B. Ärztegesetz 1998, Apothekenbetriebsordnung 2005, Krankenanstalten- und Kuranstaltengesetz, Gesundheits- und Krankenpflegegesetz, Rezeptpflichtgesetz, Datenschutzgesetz, Gesundheitstelematikgesetz 2012, DSGVO) zu erfolgen. Technische Möglichkeiten können gesetzliche Bestimmungen selbstverständlich nicht verändern, vielmehr sind die technischen Möglichkeiten im Einklang mit den Gesetzen zu nutzen.
Die Einhaltung der gesetzlichen Bestimmungen liegt im Verantwortungsbereich der Ersteller der CDA-Dokumente.
Bitte stellen Sie sicher, dass Sie die datenschutzrechtliche Einwilligung von dritten Personen (Probenabnehmende Person o.Ä.), die im Befund dokumentiert werden, haben.
2.7 Wichtige unterstützende Materialien
Auf der Website
Labor- und Mikrobiologiebefund Guide werden unter anderem folgende Materialien zur Verfügung gestellt:
- die PDF-Version dieses Leitfadens
- Beispieldokumente
- ein erweitertes CDA-Schema
- Schematron-Prüfregeln
Die im weiteren angeführten Templatespezifikationen wurden im Art-Decor Projektrepository Labor- und Mikrobiologiebefund erstellt und können dort eingesehen werden.
Gemeinsam mit diesem Leitfaden werden auf der Website der ELGA GmbH weitere Dateien und Dokumente zur Unterstützung bereitgestellt:
- Beispieldokumente
- Referenz-Stylesheet (Tool zur Darstellung im Browser - Konvertierung in HTML)
- CDA2PDF Suite (Tool zur Erzeugung einer PDF-Datei zur Ausgabe am Drucker)
- Schematron-Dateien für die Prüfung der Konformität ("Richtigkeit") von CDA Dateien
- Vorgaben zur Registrierung von CDA-Dokumenten (Leitfaden für XDS-Metadaten)
- Hinweise für die zu verwendenden Terminologien
- Leitfaden zur richtigen Verwendung von Terminologien
Fragen, Kommentare oder Anregungen für die Weiterentwicklung können an
cda@elga.gv.at gesendet werden.
2.8 Bedienungshinweise
2.8.1 Farbliche Hervorhebungen und Hinweise
Themenbezogene Hinweise zur besonderen Beachtung:
Hinweis:
Es dürfen keine Elemente oder Attribute verwendet werden, die nicht vom allgemeinen oder einem speziellen ELGA-Implementierungsleitfaden definiert wurden
Hinweis auf anderen Implementierungsleitfaden:
Verweis
Verweis auf den Allgemeinen Leitfaden:…
Themenbezogenes CDA Beispiel-Fragment im XML Format:
<BEISPIEL>
<languageCode code="de-AT" />
2.8.2 PDF-Navigation
Nutzen Sie die bereitgestellten Links im Dokument (z.B. im Inhaltsverzeichnis), um direkt in der PDF-Version dieses Dokuments zu navigieren. Folgende Tastenkombinationen können Ihnen die Nutzung des Leitfadens erleichtern:
- Rücksprung: Alt + Pfeil links und Retour: Alt + Pfeil rechts
- Seitenweise blättern: "Bild" Tasten
- Scrollen: Pfeil nach oben bzw. unten
- Zoomen: Strg + Mouserad drehen
- Suchen im Dokument: Strg + F
3 Begriffsdefinitionen
| Begriff
|
Definition
|
| Laborbefund
|
Ein Laborbefund (aus dem Bereich der med. u. chem. Labordiagnostik) ist der fachärztlich vidierte, kommentierte/interpretierte Befund morphologischer, biologischer, chemischer, molekularer, physikalischer und spezieller immunologischer Analyseverfahren aus Körpersäften, der Beurteilung ihrer morphologischen Bestandteile sowie von ab- und ausgeschiedenem Untersuchungsmaterial zur Erkennung physiologischer Eigenschaften, krankhafter Zustände, zu Verlaufskontrollen und zur Gesundheitsvorsorge/Prophylaxe.
Das Verständnis eines Laborbefundes im Rahmen dieses Leitfadens erstreckt sich dabei über das gesamte Spektrum der laboranalytisch ermittelten Befunde. Laborbefunde umfassen u. a. klinische Chemie und Immunchemie, Hämatologie, Hämostaseologie, Proteinchemie, Serologie, molekulare Diagnostik, Toxikologie, Drugmonitoring, Mikrobiologie, Infektionsserologie, Zytologie, Untersuchungen und die Hilfestellung für andere Fächer im Rahmen von Therapievorschlägen bei Gerinnungsstörungen, Antikoagulanzientherapien, der Impfkontrolle, Vorsorgediagnostik und Risikostratifizierung. Die gewählten Strukturen ermöglichen prinzipiell eine Übermittlung des gesamten Befundspektrums des Laborbereiches.
Analysen des Sonderfaches "Blutgruppenserologie und Transfusionsmedizin" werden in einer gesonderten ELGA Dokumentenklasse (geplant) abgehandelt.
Im ELGA Laborbefund dürfen nur dann Ergebnisse aus genetischen Analysen enthalten sein, wenn ihre Dokumentation in Übereinstimmung mit dem Gentechnikgesetz (GTG § 71a, BGBl. I Nr. 127/2005) erfolgt.
|
| Mikrobiologiebefund
|
Der Mikrobiologiebefund als spezieller Laborbefund umfasst im Allgemeinen die Bereiche der Beschreibung des entnommenen Materials inklusive einer makroskopischer Beurteilung, die mikroskopische Analyse des Materials, kulturelle Erregernachweise (inkl. Antibiogramm), molekularer Erregernachweise sowie der Infektionsserologie.
|
| Analyse
|
Sammelbegriff für Laboruntersuchung, Laborleistung und Labormessgröße.
|
| Erreger
|
Erreger oder Krankheitserreger sind Stoffe oder Organismen, die in anderen Organismen potenziell gesundheitsschädigende Abläufe verursachen können.
|
| Untersuchungsmaterial
|
Sammelbegriff für Spezimen, Probe, etc.
Die englische Bezeichnung "Specimen" wird vorwiegend dann verwendet, wenn sich der Kontext auf das IHE PaLM TF3[22] oder auf das RIM[18] bezieht.
|
4 Einleitung
4.1 Ausgangslage und Motivation
Mit dem "Implementierungsleitfaden für Laborbefunde" konnte über viele Jahre bereits Erfahrung im Bereich der CDA-basierten Labordokumentation gesammelt werden. Im Laufe der Zeit haben sich neue Anforderungen - vor allem im Bereich der Mikrobiologie - ergeben bzw. wurde es z.B. durch die österreichweite Verfügbarkeit von SNOMED CT möglich gemacht, einzelne Bereiche vollständig zu codieren. Insofern überarbeitet und erweitert der gegenständliche Implementierungsleitfaden das Spektrum der codierbaren Daten. Vor allem der "Mikrobiologiebefund" ermöglicht es, Befunde im Sinne des üblichen Untersuchungsverlaufs der Mikrobiologie abzubilden. In Summe soll dadurch die Datenqualität erhöht, der Informationsabgleich verbessert und die Interpretation der Information erleichtert werden.
Der "Labor- und Mikrobiologiebefund" basiert auf den Vorgaben des Allgemeinen Implementierungsleitfadens und aktualisiert und erweitert den bestehenden ELGA CDA Implementierungsleitfaden Labor 2.06.2.
4.2 Zweck des Dokuments
Der vorliegende "Implementierungsleitfaden für den Labor- und Mikrobiologiebefund" beschreibt die einheitliche Implementierungsvorschrift für den Informationsaustausch von Labor- und Mikrobiologiebefunden im österreichischen Gesundheitswesen. Der Leitfaden basiert auf den vorangegangenen Erfahrungen in der Erstellung von Implementierungsleitfäden für ELGA CDA Dokumente.
Der sogenannte "Header" beinhaltet zum einen administrative Daten (allgemeine Angaben zum Dokument, Daten zum Patienten, usw.) und dient zum anderen auch als Quelle für die Metadaten, die bei der Registrierung des Dokuments in ELGA verwendet werden. Der Header wurde über alle Anwendungsbereiche der ELGA einheitlich abgestimmt. Die medizinisch relevanten Daten, die im Rahmen von Analysen erfasst werden, sind im sogenannten "Body" enthalten. Die vorliegende Spezifikation der laborspezifischen inkl. mikrobiologischen Inhalte eines Laborbefundes in ELGA wurde von der Expertengruppe beruhend auf einer Liste mit Vorgaben der österreichischen Gesellschaft für Labormedizin und klinische Chemie (ÖGLMKC) erstellt.
4.3 Zielgruppe
Anwender dieses Dokuments sind Softwareentwickler und Berater, die allgemein mit Implementierungen und Integrationen im Umfeld der ELGA, insbesondere der ELGA-Gesundheitsdaten, betraut sind. Eine weitere Zielgruppe sind alle an der Erstellung von CDA-Dokumenten beteiligten Personen, einschließlich der Endbenutzer der medizinischen Softwaresysteme und der Angehörigen von Gesundheitsberufen.
5 Leitfadenerstellungs- und Harmonisierungsprozess
Für die Ausgestaltung der Inhalte von "CDA Implementierungsleitfäden" ist eine breite Beteiligung der Stakeholder wesentlich, um die praktische Nutzbarkeit und die Akzeptanz durch die ELGA-Benutzer sicherzustellen.
Für diese interdisziplinären Expertengruppen stehen nicht die technischen, sondern vor allem medizinisch-inhaltliche Aspekte im Vordergrund. Die technischen Inhalte werden großteils von den Redaktionsteams beigetragen.
Ein wesentlicher Schritt auf dem Weg zur Interoperabilität der IT-Systeme im Gesundheitswesen ist die Einigung auf Vorgaben für einheitliche Dokumentation und Codierung der Information. Diese durch die Arbeitsgruppen erreichte "Harmonisierung" etabliert neue nationale Qualitätsstandards der medizinischen Dokumentation.
Die Leitfäden werden über ein reguläres Standardisierungsverfahren ("Ballot") durch die HL7 Anwendergruppe Österreich (HL7 Austria) zu einem nationalen HL7 Standard.
Dieser Implementierungsleitfaden ist eine Weiterentwicklung des Laborbefundes 2.06.2 und entstand durch die Harmonisierungsarbeit der AG Labor- und Mikrobiologiebefund, die im Zeitraum von Oktober 2016 bis Oktober 2020 tagte. Die Teilnehmer der Arbeitsgruppe wurden durch ihre Organisation delegiert.
Die Arbeitsgruppe harmonisierte primär die inhaltlichen Vorgaben und soweit möglich die zu verwendenden Terminologien (Value Sets). Die Formulierung der technischen Spezifikation des CDA Implementierungsleitfadens Labor- und Mikrobiologiebefund erfolgte durch die ELGA GmbH parallel bzw. nach der inhaltlichen Festlegung.
Der Leitfaden wurde in einem technischen Abstimmungsverfahren durch die HL7 Austria (Ballot 2021-2) zu einem österreichischen HL7-Standard. Die Verbindlichkeit zur Anwendung wird durch eine Verordnung zum Gesundheitstelematikgesetz 2012, BGBl.I Nr.111/2012 begründet.
5.1 Revision der Leitfäden
Neue und geänderte Anforderungen sowie Verbesserungen können neue Versionen der bestehenden Spezifikationen notwendig machen.
Der CDA-Koordinator evaluiert in regelmäßigen Abständen, ob und welche Änderungen (etwa durch neue medizinische oder gesetzliche Anforderungen) notwendig sind. Aufgrund des Berichtes des CDA-Koordinators empfiehlt die ELGA GmbH die Erstellung von Revisionsversionen der bestehenden Leitfäden. Die geplanten Änderungen sollen mit den maßgeblichen Stakeholdern abgestimmt werden.
Neue Versionen, die "verpflichtende Elemente" (Sections oder Entries) neu einführen oder entfernen, sind "Hauptversionen", die jedenfalls über eine Durchführungsverordnung verbindlich gemacht und veröffentlicht werden. Andere Versionen sind "Nebenversionen". Alle verbindlichen Versionen sind auf www.gesundheit.gv.at zu veröffentlichen.
5.2 Autoren und Mitwirkende
Dieser Implementierungsleitfaden entstand durch die Harmonisierungsarbeit der "Arbeitsgruppe Labor- und Mikrobiologiebefund" bestehend aus nachfolgend genannten Personen. Die Arbeiten für den vorliegenden Leitfaden wurden von den Autoren gemäß dem Stand der Technik und mit größtmöglicher Sorgfalt erbracht.
5.2.1 Autoren
Das Redaktionsteam bestand aus folgenden Personen1:
| Name
|
Organisation
|
Rolle
|
| Gabriel Kleinoscheg
|
ELGA GmbH
|
Autor
|
| Andrea Klostermann
|
ELGA GmbH
|
Autor
|
| Stefan Sabutsch
|
ELGA GmbH, HL7 Austria
|
Autor, Herausgeber
|
| Nikola Tanjga
|
ELGA GmbH
|
Autor
|
| Matthias Frohner
|
FH Technikum Wien
|
Autor
|
Unter Mitwirkung von:
Oliver Kuttin (ELGA GmbH), Stephan Rainer-Sablatnig (ELGA GmbH), Nina Svec (ELGA GmbH)
1 Personen werden ohne Titel angegeben.
5.2.2 Mitwirkende
Teilnehmer der Arbeitsgruppe Labor- und Mikrobiologiebefund1
Christoph Aspöck (Klinisches Institut für Hygiene und Mikrobiologie, Universitätsklinikum St. Pölten - Lilienfeld), Jeroen S. de Bruin (docmetric GmbH), Gebhard Feierl (Diagnostik und Forschungsinstitut f. Hygiene, Mikrobiologie und Umweltmedizin, Medizinische Universität Graz), Christa Freibauer (Institut für Pathologie LK Mistelbach, ÖGPath/IAP Austria), Joseph Gappmayer (medilab - medizinisch-chemisches Labor Dr. Mustafa, Dr. Richter OG), Milo Halabi (Institut für klinische Pathologie, Mikrobiologie und molekulare Diagnostik, Krankenhaus der Barmherzigen Schwestern Ried), Alexander Haushofer (Institut für Medizinische und Chemische Labordiagnostik, Klinikum Wels-Grieskirchen GmbH), Markus Hell (Fachbereich/Abteilung für Klinische Mikrobiologie und Hygiene, medilab - medizinisch-chemisches Labor Dr. Mustafa, Dr. Richter OG), Günter Igler (analyse BioLab GmbH), Heidrun Kerschner (analyse BioLab GmbH), Gabriel Kleinoscheg (ELGA GmbH), Andrea Klostermann (ELGA GmbH), Christoph Koidl (Diagnostik und Forschungsinstitut f. Hygiene, Mikrobiologie und Umweltmedizin, Medizinische Universität Graz), Eva Leitner-Meyer (D&F Institut für Hygiene Mikrobiologie und Umweltmedizin, Medizinische Universität Graz), Michael Nöhammer (Österreichische Ärztekammer), Maximilian Ossana (Black Tusk GmbH), Tina Prager (Abteilung Med./Pfleg. Prozessmanagement, NÖ Landesgesundheitsagentur), Elisabeth Presterl (Universitätsklinik für Krankenhaushygiene und Infektionskontrolle, Medizinische Universität Wien), Stephan Rainer-Sablatnig (ELGA GmbH), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Ulrich Sagel (LADR Ihr Labor vor Ort), Carina Seerainer (FH JOANNEUM Gesellschaft mbH), Nina Svec (ELGA GmbH), Nikola Tanjga (ELGA GmbH), Gerhard Weigl (Inst. f. Labormedizin – Klinik Penzing, Wien), Birgit Willinger (Klinisches Institut für Labormedizin, Abteilung für Klinische Mikrobiologie, AKH Wien bzw. Medizinische Universität Wien)
1 Personen sind in alphabetischer Reihenfolge ohne Titel angegeben.
5.2.3 Autoren und Mitwirkende vergangener Leitfadenversionen
Die erste Version dieses Implementierungsleitfadens (2.06) entstand durch die Harmonisierungsarbeit der "Arbeitsgruppe Laborbefund" im Zeitraum zwischen 2008 und 2012, bestehend aus den unten genannten Personen2.
Herausgeber, Editor, CDA Koordinator
Stefan Sabutsch (ELGA GmbH)
Autoren, Fachkoordinatoren und Moderatoren
Matthias Frohner (Fachhochschule Technikum Wien), Alexander Mense (Fachhochschule Technikum Wien, HL7 Austria), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Stefan Sauermann (Fachhochschule Technikum Wien)
AG Teilnehmer
Bernhard Böhm (Vinzenz Gruppe Krankenhausbeteiligungs- und Management GmbH), Franz Burghuber (Kurienversammlung der niedergelassenen Ärzte der OÖ Ärztekammer), Christian Cebulla (KAV Wien, Generaldirektion), Georg Endler (Wilhelminenspital der Stadt Wien, Zentrallabor, KAV Wien), Manuela Födinger (KFJ – Sozialmed. Zentrum Süd, Institut für Laboratoriumsdiagnostik), Helmuth Gamper (Max management Consulting GmbH), Ferenc Gerbovics (Fachhochschule Technikum Wien), Bernhard Göbl (act Management Consulting GmbH), Andrea Griesmacher (Univ.Klin. Innsbruck, Zentralinst. Labordiagnostik), Walter-Michael Halbmayer (ÖQUASTA; KH Hietzing + NZ Rosenhügel, Institut f. Labordiagnostik), Susanne Hauptlorenz (LKH Vöcklabruck, Institut f. Med.Chem. Labordiagnostik u. Blutdepot), Alexander Haushofer (Österreichische Ärztekammer, KH St. Pölten, Inst. für Laboratoriumsmedizin), Jörg Hofmann (Österreichische Ärztekammer, Wiener KAV, Sozialmedizinisches Zentrum Ost - Donauspital, Institut für Labormedizin), Gerhard Holler (Österreichische Ärztekammer, ON-K 238), Konrad Hölzl (Wiener Krankenanstaltenverbund, KAV-IT), Wolfgang Hübl (KAV Wien, Wilhelminenspital, Zentrallabor, ÖGLMKC), Christof Jungbauer (Rotes Kreuz, Blutspendezentrale Wien), Christian Kampenhuber (GESPAG Gesundheitsinformatik-Bereichsleiter), Harald Kessler (Medizinische Universität Graz, Institut für Hygiene, Mikrobiologie und Umweltmedizin), Christian Kraml (Systema), Michael Krausenbaum (vision4health Deutschland GmbH & Co. KG), Walter Krugluger (Sozialmedizinisches Zentrum Ost), Thomas Leitha (Sozialmedizinisches Zentrum Ost), Herbert Matzenberger (Systema), Susanna Michalek (Initiative-ELGA), Helmut Mittermayer (Elisabethinen Linz), Georg Mustafa (Österreichische Ärztekammer, Bundesfachgruppe Labor), Georg Paucek (Medicon Medical Consulting), Johann Perné (Medizinisches Labor Perné), Gerald Regenfelder (KABEG), Dietmar Reiter (Tilak, Informationstechnologie/IT-Abteilung), Hans Richter (Labatech Handelsgesellschaft m.b.H.), Harald Rubey (LK Weinviertel Mistelbach, Laborinstitut, NÖ LK-Holding), Dieter Schwartz (Med. Uni. Wien, Klinik f. Blutgruppenserologie u. Transfusionsmedizin), Wolfgang Sischka (Assista Laborelectronics GmbH), Thomas Szekeres (Österreichische Ärztekammer), Beate Tiran (Kages Zentrallabor), Christoph Unfried (HCS, Health Communication Service), Philipp Urbauer (Fachhochschule Technikum Wien)
Patronanz, Akkordierung, Ergänzungen, Zustimmung
Clemens Auer (Bundesministerium für Gesundheit), Michael Danninger (Labene), Hubert Eisl (ELGA GmbH), Peter Fraunberger (Medizinisches Zentrallaboratorium GmbH), Josef Galler (Steiermärkische Krankenanstaltenges. m.b.H.), Gerhard Gretzl (Solve Consulting), Ulrike Gruber-Mösenbacher (Landeskrankenhaus Feldkirch, Institut für Pathologie), Milo Halabi (Krankenhaus der Barmherzigen Schwestern Ried, Inst. f. Pathologie), Elisabeth Haschke-Becher (A.ö. Krankenhaus der Elisabethinen Linz, Institut für Medizinische und Chemische Labordiagnostik), Susanne Herbek (ELGA GmbH), Wolfgang Hiesl (Oö. Gesundheitsfonds / eHealth Management), Martin Hurch (ELGA GmbH), Stylianos Kapiotis (Med. Uni. Wien, Klinisches Institut für Medizinische und Chemische Labordiagnostik), Peter Konrath (B&S Zentrallabor), Oliver Kuttin (ELGA GmbH), Georg Lechleitner (Tilak, Abteilungsleiter Informationstechnologie/IT-Abteilung), Hubert Leitner (Steiermärkische Krankenanstaltenges. m.b.H.), Helmut Lindorfer (Österreichische Agentur für Gesundheit und Ernährungssicherheit GmbH - AGES), Sabine Manhardt (Österreichische Ärztekammer, Sekretariat), Achim Mühlberger (GRZ IT Center Linz GmbH), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, Fachgesellschaft Labormedizin), Michael Nebel (Gibodat EDV- und Organisationsberatungs GmbH), Susan Netzl (AUVA - Unfallkrankenhaus Meidling, Labor), Claudia Perndl (A.ö. Krankenhaus der Elisabethinen Linz, Institut für Medizinische und Chemische Labordiagnostik, EDV), Sven Plattner (BKH Hall in Tirol, EDV), Thomas Pöckl (NÖ Landeskliniken-Holding), Angelika Reiner-Concin (Sozialmedizinisches Zentrum Ost – Donauspital, Pathologisch-Bakteriologisches Institut), Alexander Schanner (NÖ Landesklinikenholding), Gerhard Schobesberger (Österreichische Ärztekammer, Labor Schobesberger), Peter Schöttel (Bartelt GmbH), Christian Schweiger (Medizinische Universität Wien / AKH Wien, klinische Abteilung für Medizinisch-chemische Labordiagnostik), Herbert Stekel (AKH Linz, Institut für Laboratoriumsmedizin), Romana Thiel (HCS, Health Communication Service), Peter Uher (Telekom Austria), Michael Weidenauer (Assista), Thomas Wrba (Medizinische Universität Wien)
Andere ELGA Arbeitsgruppen:
Entlassungsbrief Arzt und Pflege: Jürgen Brandstätter (CodeWerk Software Services and Development GmbH)
Befundbericht Radiologie: Andreas Lindner (Lindner TAC), Martin Weigl (AIMC)
AG Laborbefund 2017
Die Änderungen für Version 2.06.2 wurde von der AG Laborbefund am 12.1.2017 abgenommen. Teilnehmer der AG Laborbefund waren:
Maria Abzieher (Wiener Krankenanstaltenverbund), Robert Alscher (Humanomed IT Solutions Gmbh), Daniel Außerdorfer (Univ.Klin. Innsbruck, Zentralinstitut für Medizinische und Chemische Labordiagnostik (ZIMCL)), René Berger (Synlab), Barbara Dall (CGM), Zeljko Drljaca (Institut für medizinische und chemische Labordiagnostik Gesellschaft m.b.H), Daniela Eisner (Salzburger Landeskliniken ), Christian Fersterer (Salzburger Landeskliniken), Matthias Frohner (FH Technikum Wien), Josef Galler (Steiermärkische Krankenanstaltenges. m.b.H.), Andrea Griesmacher (Univ.Klin. Innsbruck, Zentralinstitut für Medizinische und Chemische Labordiagnostik (ZIMCL)), Gernot Gruber (Institut für medizinische und chemische Labordiagnostik Gesellschaft m.b.H), Sylvia Handler (biomed austria - Österreichischer Berufsverband der Biomedizinischen AnalytikerInnen), Susanne Hauptlorenz (Salzkammergut-Klinikum Vöcklabruck, Institut für Med.Chem. Labordiagnostik und Blutdepot), Alexander Haushofer (ÖGLMKC; Klinikum Wels-Grieskirchen), Wolfgang Hießl (Oö. Gesundheitsfonds / eHealth Management), Michael Hubmann (Med. Zentrallaboratorium GmbH), Günter Igler (Analyse BioLab; KH Elisabethinen Linz), Sonja Jansen-Skoupy (KAV Wien, SMZ Süd), Michael Krausenbaum (CGM LAB Deutschland GmbH), Herwig Loidl (Carecenter Software GmbH), Birgit Luxbacher (biomed austria - Österreichischer Berufsverband der Biomedizinischen AnalytikerInnen), Herbert Matzenberger (CompuGroup Medical CEE GmbH), Alexander Mense (Fachhochschule Technikum Wien, HL7 Austria), Hans Georg Mustafa (Labor Dr. Hans Georg Mustafa, BFG Med. u. Chem. Labordiagnostik, ÖGLMKC), Stefan Mustafa (Labor Doz. Mag. DDr. Stefan Mustafa), Michael Nöhammer (Ärztekammer Österreich), Thomas Pöckl (NÖ Landeskliniken-Holding), Elisabeth Presterl (Universitätsklinik für Krankenhaushygiene & Infektionskontrolle, Medizinische Universität Wien), Sebastian Reimer (BMGF), Harald Rubey (LK Weinviertel Mistelbach, Laborinstitut, NÖ LK-Holding), Stefan Sabutsch (ELGA GmbH, HL7 Austria), Ulrich Sagel (Universitätsklinik für Hygiene und Mikrobiologie), Karin Salzmann (Univ. Klinik für Innere Medizin Innsbruck), Clemens M. Sampl (BMGF), Stefan Sauermann (FH Technikum Wien, Interoperabilitätsforum Österreich), Peter Schöttel (Bartelt GmbH), Christian Schweiger (Medizinische Universität Wien / AKH Wien, klinische Abteilung für Medizinisch-chemische Labordiagnostik), Carina Seerainer (ELGA GmbH), Josef Seier (Klinikum Wels-Grieskirchen), Wolfgang Sischka (Assista Laborelectronics GmbH), Herbert Stekel (Kepler Universitätsklinikum GmbH), Michael Svizak (AUVA Hauptstelle - Ärztliche Direktion), Gerhard Weigl (Inst. f. Labormedizin – Otto-Wagner-Spital Wien)
2 Personen sind in alphabetischer Reihenfolge ohne Titel angegeben.
6 Technischer Hintergrund
7 Allgemeine Richtlinien für ELGA CDA-Implementierungsleitfäden
8 Funktionale Anforderungen
8.1 Voraussetzungen für den Zugriff auf e-Befunde in ELGA
Der ELGA GDA ist in ELGA angemeldet, berechtigt und besitzt eine gültige Kontaktbestätigung für den Patienten.
Der Patient ist ELGA-Teilnehmer und hat keinen Widerspruch hinsichtlich ELGA eingelegt.
8.2 Anwendungsfälle des Dokumentenmanagements
Die folgenden Kapitel aus dem allgemeinen Leitfaden stellen eine Zusammenfassung der Inhalte der ELGA-Gesamtarchitektur, des Leitfadens XDS Metadaten und Usability Styleguides zum Thema e-Befunde dar. Detailinformationen sind in den entsprechenden Dokumenten nachzulesen (verfügbar auf der Homepage der ELGA GmbH). Die wesentlichen Anwendungsfälle sind
8.3 Verwendung in der ELGA Infrastruktur
8.3.1 Vorgaben zu Dokumenten-Metadaten (XDS-Metadaten)
Im Folgenden werden spezifische Anforderungen für die Generierung der XDS-Metadaten dargestellt. Die allgemein gültigen Regeln für die Erstellung der XDS-Metadaten sind im "Implementierungsleitfaden XDS Metadaten" (in der jeweils gültigen Version) auf der ELGA Homepage (www.elga.gv.at) abrufbar.
Spezielle Anforderungen an das Metadaten-Mapping ergeben sich für das XDS-Metadaten-Element "eventCodeList". Dieses Element basiert auf dem CDA "serviceEvent"-Element und übernimmt die durchgeführten medizinischen Leistungen in die Metadaten.
Im Kontext dieses Leitfadens wird in die eventCodeList folgende Information übernommen:
- die im Dokument enthaltenen Sections und
- die Codierungsgrade der einzelnen Sections.
Das bedeutet, dass im Zuge einer Dokumentensuche erkannt werden kann, welche Inhalte ein Labor- oder Mikrobiologiebefund enthält sowie die Information, ob bzw. welche Inhalte maschinenlesbar vorliegen. Damit kann ein abrufendes System erkennen, welche Daten in codierter Form aus dem Labor- oder Mikrobiologiebefund übernommen werden können.
Um dies zu erreichen, MUSS in den CDA "serviceEvent"-Elementen neben dem "code"-Element auch zwingend die ID des "serviceEvent" angegeben werden:
- Das "code"-Element MUSS das "code"-Element der Section wiedergeben und
- die ID des "serviceEvent"-Elements MUSS die OID des "templateId"-Elements der Section wiedergeben.
- Diese Regel MUSS für jede Section wiederholt werden. Das heißt, enthält das CDA-Dokument vier Sections, so MÜSSEN im CDA-Header vier "documentationOf/serviceEvent"-Elemente enthalten sein. Diese Regel gilt nicht für die Sections "Brieftext" und "Abschließende Bemerkungen".
| XDS-Mapping
|
Optionalität
|
CDA-Element
|
Mapping Vorschrift
|
| eventCodeList
|
R
|
ClinicalDocument/documentationOf/serviceEvent/id und ClincialDocument/documentationOf/serviceEvent/code
|
der "serviceEvent"-Code wird mit der "serviceEvent"-ID verknüpft
|
Daher ergeben sich folgende Vorschriften für das Mapping von CDA-Element zu XDS-Metadaten:
$code = concat(ClincialDocument/documentationOf/serviceEvent/code[@code],"^",ClinicalDocument/documentationOf/serviceEvent/id[@root])
$displayName = ClincialDocument/documentationOf/serviceEvent/code[@displayName]
$codeSystem = ClincialDocument/documentationOf/serviceEvent/code[@codeSystem]
Dabei muss das Mapping für jedes serviceEvent einzeln durchgeführt und ein entsprechender rim:Classification-Block erstellt werden.
<rim:Classification
classificationScheme="urn:uuid:2c6b8cb7-8b2a-4051-b291-b1ae6a575ef"
classifiedObject="theDocument"
nodeRepresentation="$code">
<rim:Name>
<rim:LocalizedString value="$displayName"/>
</rim:Name>
<rim:Slot name="codingScheme">
<rim:ValueList>
<rim:Value>urn:oid:$codeSystem</rim:Value>
</rim:ValueList>
</rim:Slot>
</rim:Classification>
8.3.2 Dokument-Metadaten (XDS-Metadaten)
| XDS-Element mit Link zum XDS-Leitfaden
|
Optionalität im XDS-Leitfaden
|
CDA-Element in /ClinicalDocument
|
Beispiele bzw. fixe Werte (fett)
|
Erklärung
|
| uniqueId
|
M
|
./id
|
- @root="1.2.40.0.34.3.1.1058.1337.999021.1"
|
Das "uniqueId"-Element beschreibt den global eindeutigen Identifier des Dokuments und kann mit oder ohne Extension angegeben werden.
|
- @root="1.2.40.0.34.3.1.1058.1337"
- @extension="999021.1"
|
| typeCode
|
R
|
./code
|
- @code="11502-2"
- @displayName="Laboratory report"
- @codeSystem="2.16.840.1.113883.6.1"
|
Zur Unterscheidung des "Dokumenttyps" erhält das "code"-Element des Laborbefundes einen vorgegebenen Wert.
|
- @code="18725-2"
- @displayName="Microbiology studies (set)"
- @codeSystem="2.16.840.1.113883.6.1"
|
Zur Unterscheidung des "Dokumenttyps" erhält das "code"-Element des Mikrobiologiebefundes einen vorgegebenen Wert.
|
| classCode
|
R
|
./code/translation
|
- @code="11502-2"
- @displayName="Laboratory report"
- @codeSystem="2.16.840.1.113883.6.1"
|
Bezeichnet die "Dokumentklasse" in dem untergeordneten "translation"-Element. Einzig zulässiger Wert für Labor- und Mikrobiologiebefund ist 11502-2 "Laboratory report".
|
| title
|
R
|
./title
|
|
Der Titel des CDA Dokuments für den lesenden Empfänger. MUSS die Art des Dokuments (Dokumenttyp) widerspiegeln.
Vorgeschlagene Titel wären zum Beispiel "Laborbefund" oder "Mikrobiologiebefund".
|
| formatCode
|
M
|
./hl7at:formatCode
|
- @code="urn:hl7-at:lab:3.0.0+20211214"
- @displayName="HL7 Austria Labor- und Mikrobiologiebefund 3.0.0+20211214"
- @codeSystem="1.2.40.0.34.5.37"
|
Version des vom CDA erfüllten ELGA Implementierungsleitfaden für Labor- und Mikrobiologiebefund.
|
| practiceSettingCode
|
M
|
./hl7at:practiceSettingCode
|
- @code="F028"
- @displayName="Labordiagnostik"
- @codeSystem="1.2.40.0.34.5.12"
|
Fachliche Zuordnung des Dokuments aus dem Value Set "atcdabbr_PracticeSetting_VS".
|
| creationTime
|
M
|
./effectiveTime
|
- @value="20181213095800+0200"
|
Dokumentiert das medizinisch zutreffendste Datum - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc.
|
| confidentialityCode
|
M
|
./confidentialityCode
|
- @code="N"
- @displayName="normal"
- @codeSystem="2.16.840.1.113883.5.25"
- @codeSystemName="HL7:Confidentiality"
|
Vertraulichkeitscode des Dokuments. Für ELGA-Dokumente ist ausschließlich "N" erlaubt!
|
| languageCode
|
M
|
./languageCode
|
|
Für ELGA-Dokumente ist in @code für CDA und Ableitungen in die XDSDocumentEntry-Metadaten derzeit ausschließlich der Wert "de-AT" zulässig. Für eHealth können in zukünftigen Versionen des Leitfadens weitere Sprachcodes erlaubt werden.
|
| referenceIdList
|
M
|
./setId
|
- @root="1.2.40.0.34.3.1.1058.1337"
- @extension="999021"
|
Eindeutige ID des Dokumentensets. Diese bleibt über alle Versionen der Dokumente gleich (initialer Wert bleibt erhalten). Die setId SOLL unterschiedlich zu /ClinicalDocument/id sein.
|
| sourcePatientId
|
M
|
./recordTarget/patientRole/id[1]
|
- @root="1.2.40.0.34.99.111.1.2"
- @extension="123"
|
Patienten ID im Informationssystem des GDA, z.B.: im KIS eines Krankenhauses.
|
| author
|
authorInstitution
|
M
|
./author[1]/assignedAuthor/
representedOrganization/id[1]
|
- @root="1.2.40.0.34.99.4.1234"
|
ID und Name der Organisation (Kurzbezeichnung), der die Person angehört, wie im GDA-Index angegeben.
|
- @root="1.2.40.0.34.99.4"
- @extension="1234"
|
| ./author[1]/assignedAuthor/
representedOrganization/name
|
|
| authorPerson
|
M
|
./author[1]/assignedAuthor
|
- ./id/@root="1.2.40.0.34.99.111.1.2"
- ./id/@extension="999021"
- ./assignedPerson/name/family="Holzer"
- ./assignedPerson/name/given[1]="Daniela"
- ./assignedPerson/name/given[2]="Chiara"
- ./assignedPerson/name/suffix="BSc"
- ./assignedPerson/name/prefix[@qualifier="AC"]="Dr."
|
Daten der Person (Name, ID, etc.)
|
| authorRole
|
M
|
./author[1]/functionCode
|
- @displayName="Diensthabender Oberarzt"
|
Rolle der Person.
|
| authorSpeciality
|
M
|
./author[1]/assignedAuthor/code
|
- @displayName="Fachärztin/Facharzt für Innere Medizin"
|
Fachrichtung des Verfassers des Dokuments aus dem Value Set "ELGA_AuthorSpeciality".
|
| legalAuthenticator
|
M
|
./legalAuthenticator/assignedEntity
|
- ./id/@root="1.2.40.0.34.99.111.1.2"
- ./id/@extension="999021"
- ./assignedPerson/name/family="Holzer"
- ./assignedPerson/name/given[1]="Daniela"
- ./assignedPerson/name/given[2]="Chiara"
- ./assignedPerson/name/suffix="BSc"
- ./assignedPerson/name/prefix[@qualifier="AC"]="Dr."
|
Rechtlicher Unterzeichner des Dokuments.
|
| eventCodeList
|
R
|
./documentationOf[n]/serviceEvent/id
|
- @root="1.2.40.0.34.6.0.11.2.102"
|
Die ID und der Code des "serviceEvent"-Elements definieren, welche Inhalte ein Labor- oder Mikrobiologiebefund enthält sowie die Information, ob bzw. welche Inhalte maschinenlesbar vorliegen (siehe Vorgaben zu Dokumenten-Metadaten (XDS-Metadaten)).
|
| ./documentationOf[n]/serviceEvent/code
|
- @code="100"
- @displayName="Blutgruppenserologie"
- @codeSystem="1.2.40.0.34.5.11"
|
| serviceStartTime
|
R
|
./documentationOf[1]/serviceEvent/
effectiveTime/low
|
- @value="20181001082015+0200"
|
Zeitpunkt des Behandlungsbeginns (erster medizinisch relevanter Behandlungstag dieser dokumentierter Gesundheitsdienstleistung)
|
| serviceStopTime
|
R
|
./documentationOf[1]/serviceEvent/
effectiveTime/high
|
- @value="20181213105900+0200"
|
Zeitpunkt des Behandlungsendes (letzter medizinisch relevanter Behandlungstag dieser dokumentierter Gesundheitsdienstleistung, muss sich von Behandlungsbeginn unterscheiden)
|
| healthcareFacilityTypeCode
|
R
|
./componentOf/encompassingEncounter/
location/healthCareFacility/code
|
- @code="300"
- @displayName="Allgemeine Krankenanstalt"
- @codeSystem="1.2.40.0.34.5.2"
|
Klassifizierung des GDA.
|
[Tabelle 1]:
Übersichtstabelle über die Dokument-Metadaten (XDS-Metadaten)
9 Konformitätsprüfung
Ein zu diesem Implementierungsleitfaden konformes CDA-Dokument ist zunächst ein valides CDA Release 2.0 XML-Dokument mit Header und Body. Darüber hinaus erfüllt es alle in diesem Leitfaden festgelegten "Geschäftsregeln".
Dies spiegelt ein generelles Konzept im Umgang mit Dokumenten wieder: die Validierung in zwei Schritten. Im ersten Schritt stellt dies die Validierung gegen zugehörige W3C Schemas dar. Das verwendete Schema ist das geringfügig erweiterte offizielle CDA Release 2.0 Schema (siehe Schema-Prüfung). Darüber hinaus existieren eine Reihe von Schematron Regeln, die für einen zweiten Validierungsschritt genutzt werden und letztlich die Detailregelungen in diesem Leitfaden wiedergeben, sowie die Einhaltung der Geschäftsregeln (Optionalität, Kardinalität/Multiplizität, Datentypen, Wertebereiche, Abhängigkeiten) sicherstellen (siehe Schematron-Prüfung). Geschäftsregeln für Abschnitte oder Elemente werden auch technisch zu "Templates" zusammengefasst. Eine XML-Instanz, die kein valides CDA-Dokument ist oder sich nicht gegen das XSD-Schema validieren lässt oder im Widerspruch zu den angegebenen Geschäftsregeln steht, ist kein gültiges CDA-Dokument im Sinne dieses Implementierungsleitfadens.
Hinweis: Nicht alle Geschäftsregeln können mit Schema oder Schematron geprüft werden (etwa Inhalte von Multimedia-Attachments, Dokumentengröße). Zusätzliche Validierungsschritte sind gegebenenfalls notwendig, um alle Regeln überprüfen zu können.
Die Kapitel zu den technischen Konformitätsprüfungen von CDA-Dokumenten sind im allgemeinen Leitfaden unter den folgenden Links zu finden:
10 Datentypen
Im Kapitel
Datentypen des allgemeinen Leitfadens werden nur die Datentypen beschrieben, die in ELGA CDA-Dokumenten wie diesem zur Anwendung kommen. Für weiterführende Informationen wird auf den zugrundeliegenden Standard Health Level Seven Version 3 (V3), Normative Edition verwiesen.
11 Vorgaben zum medizinischen Inhalt
11.1 Labor- und Mikrobiologiebefund
Die inhaltliche Definition beruht auf den Mindestvorgaben der österreichischen Gesellschaft für Labormedizin und klinischen Chemie (ÖGLMKC) und wurde weiter verfeinert. Die folgende Tabelle zeigt einen Überblick über die inhaltlich abzubildenden medizinisch relevanten Daten.
| Feld
|
Beschreibung
|
Bereich
|
| Allgemeine Befundinformationen
|
| Zeitpunkt der Auftragserfassung
|
Datum und Zeitpunkt, an dem das analysierende Labor die Anforderung vom Zuweiser in der Labor-EDV erfasst hat.
|
Documentation Of Service Event - Labor und Mikrobiologie:
documentationOf/serviceEvent/effectiveTime/low
|
| Auftragsdiagnose (Zuweiserdiagnose) bzw. Überweisungsgrund
|
Vom Auftraggeber bestimmte und dem Labor übermittelte Verdachtsdiagnose bzw. der Überweisungsgrund
|
Überweisungsgrund - codiert oder Überweisungsgrund - uncodiert
|
| Angeforderte Analyse bzw. Fragestellung
|
Vom Auftraggeber angeforderte Analyse(-spektrum) bzw. die Fragestellung
|
Angeforderte Untersuchungen - codiert oder Angeforderte Untersuchungen - uncodiert
|
| Befundtext
|
Kommentar zum gesamten Befund
|
Befundbewertung
|
| Information zum Untersuchungsmaterial
|
| Zeitpunkt der Gewinnung des Untersuchungsmaterials
|
Damit ist jenes Datum und Zeitpunkt gemeint, an dem das Untersuchungsmaterial für die geplante Analyse gewonnen wurde. Die Dokumentation des Zeitpunkts der Gewinnung ist in der Verantwortung der entnehmenden Person, die in vielen Fällen mit dem Befundersteller nicht identisch ist, weil Untersuchungsmaterial meist zur Analyse an Labors versendet werden. Daher ist der Zeitpunkt vielfach im Labor nicht feststellbar.
|
Specimen Collection
procedure/effectiveTime
|
| Zeitpunkt des Einlangens des Untersuchungsmaterials
|
Datum und Zeit der Annahme des Untersuchungsmaterials im Labor
|
Specimen Received
act/effectiveTime
|
| Art des Untersuchungsmaterials (Specimen Type)
|
Art des Untersuchungsmaterials
|
Specimen Collection
procedure/participant[@typeCode='PRD']/
participantRole[@classCode='SPEC']/playingEntity/code
|
| Entnahmeort
|
Angabe der Körperstelle, von der das Untersuchungsmaterial stammt
|
Specimen Collection
procedure/targetSiteCode
|
| ID des Untersuchungsmaterials
|
Eindeutige Nummer des Untersuchungsmaterials
|
Specimen Collection
procedure/participant[@typeCode='PRD']/
participantRole[@classCode='SPEC']/id
|
| Entnehmende Person (Performer)
|
Person, welche die Entnahme des Untersuchungsmaterials durchgeführt hat
|
Specimen Collection
procedure/performer
|
| Allgemeine Anmerkungen des Labors zur Qualität des Untersuchungsmaterials
|
Präanalytik pro Untersuchungsmaterial zu dessen Qualität
|
Specimen Received
act/entryRelationship
|
| Allgemeine Laborergebnisse
|
| Gruppierung / Befundgruppen
|
Analysengruppierung
|
Laboratory Specialty Section bzw. Laboratory Battery Organizer
|
| ID des Tests
|
Eindeutige Codierung des Tests
|
Laboratory Observation
observation/id
|
| Analysenbezeichnung
|
Bezeichnung der Analyse
|
Laboratory Observation
observation/code
|
| Ergebnis
|
|
Laboratory Observation
observation/value[@value]
|
| Einheit
|
|
Laboratory Observation
observation/value[@unit]
|
| Referenzbereich
|
Für die Beurteilung relevante Referenzwerte.
|
Laboratory Observation
observation/referenceRange
|
| Befundinterpretation
|
Codierte Bewertung des Ergebnisses
|
Laboratory Observation
observation/interpretationCode
|
| Ergebniskommentar
|
Kommentar des Labors zu einem einzelnen Testergebnis
|
Laboratory Observation
observation/entryRelationship/act/
templateId[@root='1.2.40.0.34.6.0.11.3.11']
|
| Externes Labor
|
Kennzeichen ob ein Ergebnis extern ermittelt wurde
|
Laboratory Observation
observation/performer/assignedEntity/code[@code='E']
|
| Kulturergebnisse
|
| Erreger (Isolat)
|
Der durch eine Kulturmethode ermittelte Erreger.
|
Laboratory Isolate Organizer
|
| Antibiogramm (inklusive MHK)
|
Ergebnis der Bestimmung der Empfindlichkeit bzw. Resistenz von Erregern gegenüber Antibiotika. Anlassbezogen wird das Ergebnis interpretiert (R, S, I) und/oder die minimale Hemmkonzentration (MHK) angegeben.
|
Laboratory Isolate Organizer und darin in einer Laboratory Observation
|
[Tabelle 2]:
Im Labor- und Mikrobiologiebefund abzubildende medizinische Daten
11.2 Laborbefund
11.2.1 Befundbereiche (Laboratory Specialty Section)
Der Laborbefund kann mehrere Abschnitte aus verschiedenen Laborbereichen (z.B. Hämatologie, Toxikologie, Urindiagnostik, etc.) beinhalten. Diese werden im Dokument als einzelne "Befundbereiche" umgesetzt und bilden somit die erste Gliederungsebene des Bodys. Die Befundbereiche entsprechen strukturell der "Laboratory Specialty Section" der IHE[22] (siehe auch Template Laboratory Specialty Section). Folgende Grafik zeigt die mögliche Gliederung auf der ersten Ebene innerhalb des Bodys.

[Abbildung 1]: Gliederung nach Befundbereichen (Laboratory Specialty Sections)
Die derzeit für den Laborbefund verwendbaren Befundbereiche werden im Rahmen des hierarchisch organisierten Value Sets "ELGA_Laborstruktur" definiert, wobei für Befundbereiche nur Einträge des Level 1 verwendet werden dürfen. Die folgende Tabelle gibt einen auszugsweisen Überblick über die derzeit festgelegten Befundbereiche. Bei der Verwendung der Befundbereiche ist die Reihenfolge gemäß Value Set verpflichtend einzuhalten.
| Code
|
Befundbereich (Laboratory Specialty Section)
|
| 100
|
Blutgruppenserologie
|
| 200
|
Blutgasanalytik
|
| 300
|
Hämatologie
|
| 400
|
Gerinnung/Hämostaseologie
|
| …
|
…
|
[Tabelle 3]:
Liste der Befundbereiche, auszugsweise gemäß Value Set "ELGA_Laborstruktur", die sich auch im Value Set "ELGA_Laborparameter" wiederfinden.
11.2.2 Befundgruppen
Innerhalb der Befundbereiche erfolgt in der Regel eine Strukturierung und Gliederung der Ergebnisse zur besseren Lesbarkeit und Auffindbarkeit in "Befundgruppen". Für die Umsetzung der Befundgruppen im Body werden Laboratory Battery Organizer verwendet. Level 2 des Value Sets "ELGA_Laborstruktur" definiert die zulässigen Befundgruppen. Grundsätzlich ist die Reihenfolge der Befundgruppen gemäß Value Set verpflichtend einzuhalten. Es besteht jedoch auch die Möglichkeit, Ergebnisse ohne Befundgruppenstrukturierung zu übermitteln.
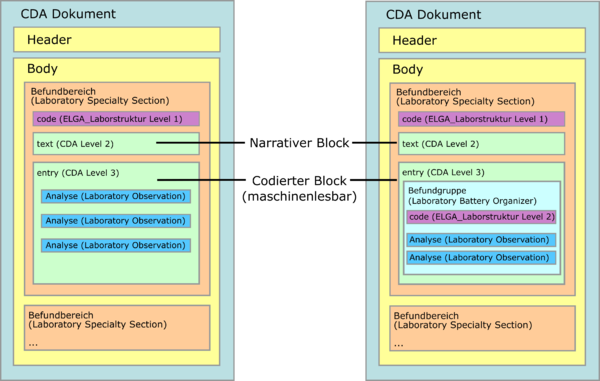
[Abbildung 2]: Strukturierungsmöglichkeiten im Laborbefund im Body
Der folgende Ausschnitt aus einem Laborbefunden enthält die Befundbereiche "Hämatologie" und "Hämostaseologie" sowie die dazugehörigen Befundgruppen "Blutbild", "Knochenmark Morphologie" bzw. "Hämostaseologie Globaltest".
11.2.3 Harmonisierung des Befundaufbaus – Value Set "ELGA_Laborparameter"
Im Rahmen der Arbeiten zum vorliegenden Dokument wurde in der Expertengruppe die grundsätzliche Übereinkunft getroffen, auch die Befundgruppen und die damit verbundene Testzuordnung entsprechend österreichweit abzustimmen. Die Strukturierung eines Laborbefundes wurde in Form des hierarchischen Value Sets "ELGA_Laborparameter" festgelegt.
Strukturierung, Reihenfolge der Parameter sowie die Bezeichnung der Parameter sind durch das Value Set "ELGA_Laborparameter" verpflichtend vorgegeben!
Eine Hilfestellung zum Mapping der lokalen Codes auf die vorgeschriebenen Codes des Value Sets bietet der "Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund"[23].
11.3 Mikrobiologiebefund
Unter den Analysen des Laborbefundes finden sich viele aus dem Bereich der Mikrobiologie. Grundsätzlich können alle Analysen auch in der "klassischen" Struktur des Laborbefundes dargestellt werden (siehe Laborbefund). Allerdings entspricht die Struktur des Laborbefundes nicht dem eines üblichen Mikrobiologiebefundes, der einem bestimmten Muster folgt, welches den Untersuchungsverlauf widerspiegelt:
- Beschreibung des entnommenen Materials (z.B. Mittelstrahlharn) inklusive makroskopischer Beurteilung
- Mikroskopische Analyse des Materials (z.B. Erythrozyten, Leukozyten, grampositive Bakterien)
- Kultureller Erregernachweis
- Benennung der Reinkulturen (Isolate) mit Nennung der taxonomischen Bestimmung der Mikroorganismen (z.B. Streptococcus pyogenes) ggf. mit Angabe des Serovars/Pathovars.
- Angabe des Antibiogramms mit Interpretation (R, S, I) und/oder minimaler Hemmkonzentration (MHK)
- Molekulare Erregernachweise
- Infektionsserologie
Die Struktur des Mikrobiologiebefundes wird durch spezielle "Laboratory Specialty Sections", die jeweils mit einem fixen Code versehen sind, vorgegeben. Details dazu sind den CDA Strukturen des Bodys des Mikrobiologiebefundes zu entnehmen.
Werden mikrobiologische Analysen in einem "normalen" Laborbefund festgehalten, so sind diese dem entsprechenden Befundbereich und - wenn möglich - der passenden Befundgruppe zuzuordnen.
11.4 Hinweise für akkreditierte Laboratorien gem. ISO 15189:2012
Die Vorgaben dieses Implementierungsleitfadens erlauben die Erzeugung von Befunden gemäß ISO 15189:2012 "Medizinische Laboratorien - Anforderungen an die Qualität und Kompetenz"[24], speziell in Hinblick auf die dort angegebenen Kapitel 5.8 (Befundberichte) und 5.9 (Freigabe der Ergebnisse). Für akkreditierte Laboratorien sind neben der ISO 15189:2012 folgende Hinweise zu beachten:
11.4.1 Angabe des Akkreditierungs-Logos
Das Akkreditierungs-Logo kann neben dem Logo des Labors angegeben werden. Technisch erfolgt das über die Section "Brieftext", das Logo ist ggf. gemeinsam mit dem Logo des Labors in einer Grafikdatei anzugeben (siehe Brieftext).
Ein Labor muss nicht zwingend für alle Analysen, die es durchführen kann, akkreditiert sein. Das Akkreditierungs-Logo kann angegeben werden, sobald es akkreditierte Analysen gibt; wenn "nicht akkreditierte Analysen" am Befund erscheinen, soll bei diesen angegeben werden können, dass das Labor für diese Analyse nicht akkreditiert ist. Für die entsprechend Markierung wird die Verwendung von Anmerkungszeichen (wie z.B. *) und Endnoten empfohlen.
11.4.2 Angabe des Analyse- bzw. Messverfahrens
Wenn der Name der Analysen keinen Rückschluss auf die Methode erlaubt, aber Analyse- bzw. Messverfahren dennoch angegeben werden sollen (ISO 15189:2012, Vorgabe 5.8.3 a)), soll dies als Kommentar zur Analyse erfolgen (siehe Laboratory Observation wie ein Kommentar zur Analyse codiert werden kann).
11.4.3 Vorgaben für den Befunddruck
Einige Vorgaben von ISO 15189:2012 beziehen sich auf Qualitätsmerkmale für gedruckte Befunde (Vorgabe 5.8.3 d) "Identifizierung des Patienten und den Aufenthaltsort des Patienten auf jeder Seite" und Vorgabe 5.8.3 p) "Seitenzahl zur Gesamtzahl der Seiten"). Dieser Leitfaden definiert ein elektronisches Format, das diese Anforderungen grundsätzlich unterstützt. Am ELGA-Portal (Zugriff für Bürger) werden Tools zur Darstellung eingesetzt, die diese Vorgaben unterstützen. Diese Tools werden auch von der ELGA GmbH zum Download bereitgestellt (Referenzstylesheet, CDA2PDF auf http://www.elga.gv.at/CDA).
12 Anwendungsfälle / User Stories
Die Einsatzszenarien für dieses Datenaustauschformat werden in Form von Anwendungsfällen beschrieben, um dem Leser den erforderlichen Hintergrund zu vermitteln. Die Beschreibung der Anwendungsfälle ist nicht normativ und keine Vorentscheidung für die tatsächliche Umsetzung.
Der in diesem Leitfaden beschriebene Labor- und Mikrobiologiebefund dient zum Austausch von fertiggestellten, und fachärztlich vidierten Befunden innerhalb und zwischen Einrichtungen des Gesundheitswesens. Ein wesentlicher Nutzer der Befunde ist auch der Patient selbst, der die Befunde über das ELGA Bürgerportal einsehen wird.
Die Regelung, welche Befunde in ELGA einzustellen sind, ist nicht Teil dieses Leitfadens.
Der in diesem Leitfaden beschriebene Labor- und Mikrobiologiebefund ist grundsätzlich zur Dokumentation und Kommunikation (vollständig) fertiggestellter Labor- und Mikrobiologiebefunde gedacht, wobei auch die Möglichkeit besteht, zu dokumentieren, wenn Analyseergebnisse noch ausständig sind. Idealerweise wird ein aktualisiertes CDA in die ELGA eingebracht, sobald die Ergebnisse vorliegen. Gleichfalls werden Ergänzungen und Korrekturen von Labor- und Mikrobiologiebefunden unterstützt.
Der hier beschriebene Labor- und Mikrobiologiebefund ist nicht als Workflow-Dokument konzipiert worden, um z.B. Zwischenergebnisse und Nachrichten über einzelne Prozessschritte zu kommunizieren. Eventuell kann ein CDA-Dokument intern als solches verwendet werden, um beispielsweise
- die Anforderung von Analysen,
- das Einlangen des Untersuchungsmaterials im Labor oder
- den Beginn, Stornierung oder die Fertigstellung einzelner Analysen abzubilden.
12.1 Anwendungsfall LAB01: "Analysen aus niedergelassenen Labors und nicht-stationären Fällen"
Typischerweise entstehen Laborbefunde in medizinischen Labors. Das sind neben Labor-Abteilungen von Spitälern auch niedergelassene Labors, die als selbständige Unternehmen Analysen anbieten. Diese werden vielfach auf Zuweisung von Patienten durch praktische Ärzte im niedergelassenen Bereich tätig. Die Entstehung eines Laborbefundes beginnt mit einer Überweisung durch einen niedergelassenen Arzt oder mit einer Anforderung aus einem nicht-stationären Fall innerhalb eines Spitals. Entweder wird das Untersuchungsmaterial am Patienten gleichzeitig entnommen und dann ins Labor geschickt oder der Patient muss das Labor aufsuchen, und das Untersuchungsmaterial wird dann erst dort entnommen. Nach Abschluss der Analyse wird der Befund dem zuweisenden Arzt und/oder dem Patienten in Papierform übermittelt.
12.2 Anwendungsfall LAB02: "Analysen im Rahmen eines stationären Aufenthalts in einem Spital"
Im Rahmen von stationären Aufenthalten von Patienten in Spitälern kommt es in der Regel zu einer Reihe von Analysen, die in der spitalsinternen Krankengeschichte (meistens auch elektronisch) abgelegt werden. Relevante Befunde werden dem einweisenden Arzt bzw. dem Patienten im Zuge der Entlassungsdokumentation mit übermittelt. Dieses passiert oftmals als Teil des Entlassungsbriefes. Welche Werte und welche Befunde entsprechende Relevanz haben, um weitergeleitet zu werden, entscheidet das jeweilige ärztliche Fachpersonal in der Klinik.
12.3 Anwendungsfall LAB03: "Teilweise externe Vergabe von Analysen"
In vielen Fällen kommt es zu Kooperationen zwischen Laborbefund-erstellenden Organisationen. Folgende Fälle seien angeführt:
- Spitäler kooperieren mit niedergelassenen Labors. Einerseits verfügen nicht alle Spitäler über eigene Labors, andererseits werden auch Spezialuntersuchungen, die das Spitalslabor nicht durchführt, an niedergelassene Labors vergeben.
- Niedergelassene Labors verfügen nicht über das volle Leistungsspektrum und senden Analysen an Spitallabors, welche spezielle Parameter messen können.
- Es bestehen Kooperationen zwischen mehreren Spitälern. Das sind oft Spitäler, die dem gleichen Spitalsträger angehören. Teilweise bestehen auch Kooperationen zwischen Spitälern unterschiedlicher Träger, die durch die örtliche Nähe leicht Untersuchungsmaterial austauschen können.
In allen Fällen werden einzelne Analysen nicht selbst durchgeführt, sondern diese Tests an ein externes kooperierendes Labor vergeben. Das externe Labor führt dann die Analyse durch und übermittelt die Ergebnisse an das ursprünglich für die Analyse zuständige Labor. Dort werden dann die vom externen Labor ermittelten Testergebnisse in den eigenen Laborbefund integriert. Das ursprünglich zuständige Labor, das den Befund erstellt, muss in diesem Fall die extern erbrachten Testergebnisse als solche erkennbar kennzeichnen (siehe Performer - Laboratory).
12.4 Anwendungsfall LAB04: "Update von Laborbefunden"
Ein fertiggestellter Labor- oder Mikrobiologiebefund wird korrigiert oder ergänzt, um
- die Inhalte des Befundes zu korrigieren (etwa das Ergebnis einer Analyse),
- einzelne (fehlerhafte) Analysen nachträglich aus dem Befund zu stornieren oder
- fehlende Analysen zu ergänzen (etwa besonders lang dauernde Analysen).
Änderungen sollen im Text für den Leser klar kenntlich gemacht werden (eine codierte Angabe kann im narrativen Text mit Revisionsmarken erfolgen).
Eine Korrekturversion MUSS in ELGA immer alle zum Befund gehörigen Analysen enthalten, da die Vorversion als veraltet (deprecated) gekennzeichnet wird. Stornierte Analysen sind explizit mit dem entsprechenden "statusCode" zu kennzeichnen.
Für den Leser/Empfänger gilt: Eine neue Version ersetzt die alte Version des Befundes, alle Analysen sollen beim Import ersetzt bzw. überschrieben werden. Sollte eine Analyse in der neuen Version fehlen, soll diese als "storniert" interpretiert werden.
13 Dataset
Das Dataset (auch "Datenarten" oder "Konzepte") listet alle mit der Arbeitsgruppe abgestimmten Inhalte des Leitfadens auf. Es enthält Beschreibungen der Elemente mit Synonymen.
Die Live-Version des Datasets in Art-Decor kann unter folgendem Link betrachtet werden.
14 Technische Spezifikation
Die Struktur des CDA Austauschformats ist in den nachfolgenden Kapiteln im Detail beschrieben.
Der Header entspricht im Wesentlichen den Vorgaben des Allgemeinen Leitfadens. Der Body enthält die tatsächlichen (medizinischen) Inhalte des Dokuments. Dieses Dokument existiert ausschließlich in einer voll strukturierten Form, eine Unterscheidung der Interoperabilitätsstufen ist daher nicht notwendig.
Dieses Kapitel gibt einen Überblick über die Elemente des CDA Headers und den Vorgaben bezüglich Kardinalität und Konformität.
Die jeweiligen Links in der letzten Spalte zeigen auf die einzelnen Header Elemente. Wo es zu keinen strukturellen Änderungen im Rahmen dieses Leitfadens gekommen ist, wird die Definition des Allgemeinen Implementierungsleitfadens verlinkt.
Der aktuelle Leitfaden ist für den ELGA Kontext entwickelt worden, kann jedoch auch für andere Zwecke, welche als eHealth zusammengefasst werden, verwendet werden.
[Tabelle 4]:
Übersichtstabelle der CDA Strukturen des Headers
Dieses Kapitel gibt einen Überblick über die Elemente des CDA Headers mit Zeitangaben und ihre Zusammenhänge.
Die jeweiligen Links in der letzten Spalte zeigen auf die einzelnen Header Elemente. Wo es zu keinen strukturellen Änderungen im Rahmen dieses Leitfadens gekommen ist, wird die Definition des Allgemeinen Implementierungsleitfadens verlinkt.
Der aktuelle Leitfaden ist für den ELGA Kontext entwickelt worden, kann jedoch auch für andere Zwecke, welche als eHealth zusammengefasst werden, verwendet werden.
| Element
|
Kard/Konf
ELGA
|
Kard/Konf
eHealth
|
Bedeutung
|
Link zum Kapitel
|
| hl7at:terminologyDate
|
1..1 M
|
0..1 O
|
Das Datum, an dem die lokal zur Implementierung verwendeten Value Sets mit dem österreichischen Terminologieserver abgeglichen wurden, wird hier angegeben.
|
Terminologie-Datum des Dokuments
|
| effectiveTime
|
1..1 M
|
1..1 M
|
Angabe des medizinisch zutreffendsten Datums - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc.
|
|
| recordTarget
birthTime
|
1..1 M
1..1 R
|
0..1 C
1..1 R
|
Der Geburtstag des Patienten.
|
Patient
|
| recordTarget
deceasedTime
|
1..1 M
0..1 R
|
0..1 C
0..1 R
|
Das Sterbedatum des Patienten.
|
Patient
|
| author
time
|
1..* M
0..1 R
|
1..* M
0..1 R
|
Das jeweilige Datum, an welche der jeweilige Autor neue medizinische Informationen hinzugefügt hat.
|
Verfasser des Dokuments
|
| dataEnterer
time
|
0..1 O
0..1 R
|
0..1 O
0..1 R
|
Das Datum, an welchem eine Schreibkraft die Informationen aus einem Medium in das CDA Dokument überträgt, ohne weitere fachliche Informationen hinzuzufügen.
|
Personen der Dateneingabe
|
| legalAuthenticator
time
|
1..1 M
1..1 R
|
1..1 M
1..1 R
|
Die Zeitpunkte, an welchem das Dokument von der berechtigten Personen vidiert wurde. Diese Person ist der Hauptunterzeichner. Dieser Zeitpunkt sollte, wenn vorhanden, nach /ClinicalDocument/author/time und /ClinicalDocument/dataEnterer/time liegen.
|
Rechtlicher Unterzeichner
|
| authenticator
time
|
0..* O
1..1 R
|
0..* O
1..1 R
|
Die Zeitpunkte, an welchem das Dokument von den einzelnen berechtigten Personen vidiert wurde. Diese Personen sind die weiteren Unterzeichner.
|
Laboratory Results Validator
|
| Notfallkontakt / Auskunftsberechtigte Person
participant [templateId[@root='1.2.40.0.34.6.0.11.1.27']]
time
|
0..* O
0..1 O
|
0..* O
0..1 O
|
Zeitraum, in dem der angegebene Kontakt den Notfall-Kontakt darstellt.
Wird nur angegeben, wenn der Kontakt bereits absehbar nur in einem eingeschränkten Zeitraum zur Verfügung steht.
|
Weitere Beteiligte
|
| Versicherter/Versicherungparticipant
participant [templateId[@root='1.2.40.0.34.6.0.11.1.26']]
time
|
0..* O
0..1 O
|
0..* O
0..1 O
|
Gültigkeitszeitraum der Versicherungspolizze.
|
Weitere Beteiligte
|
| documentationOf
serviceEvent
effectiveTime
|
1..* M
1..1 M
1..1 M
|
1..* M
1..1 M
1..1 M
|
Zeitraum der durchgeführten Gesundheitsdienstleistung.
|
Documentation Of Service Event - Labor und Mikrobiologie
|
| componentOf
encompassingEncounter
effectiveTime
|
0..1 O
1..1 M
1..1 M
|
0..1 O
1..1 M
1..1 M
|
Zeitraum des Patientenkontakts.
|
Patientenkontakt (Aufenthalt)
|
[Tabelle 5]:
Übersichtstabelle der Header-Elemente für Zeitpunkte/Zeitspannen
14.3 Übersichtstabelle der CDA Strukturen des Bodys
Die folgenden Tabellen geben die im ELGA Labor- bzw. Mikrobiologiebefund verwendeten Sections und Entries wieder. Angaben über die Verwendung einzelner Elemente können - sofern nicht in dieser Tabelle aufgeführt - in den jeweiligen Section- oder Entry-Spezifikationen gefunden werden (aus Gründen der Übersichtlichkeit wurde auf die Darstellung aller Ebenen verzichtet).
14.3.1 Laborbefund
| Section bzw. Entry
|
Template ID
|
Kard/Konf
|
Kapitel
|
| Brieftext
|
1.2.40.0.34.6.0.11.2.69
|
0..1 O
|
Template-Spezifikation
|
| Der "Überweisungsgrund" kann codiert oder uncodiert angegeben werden:
|
|
|
Überweisungsgrund - codiert
|
1.2.40.0.34.6.0.11.2.6
|
0..1 O
|
Template-Spezifikation
|
|
|
|
Konsultationsgrund Problem Concern Entry
|
1.2.40.0.34.6.0.11.3.30
|
1..* M
|
Template-Spezifikation
|
|
|
Überweisungsgrund - uncodiert
|
1.2.40.0.34.6.0.11.2.114
|
0..1 O
|
Template-Spezifikation
|
| Die "Anamnese - Labor und Mikrobiologie" kann codiert oder uncodiert angegeben werden:
|
|
|
Anamnese - Labor und Mikrobiologie - codiert
|
1.2.40.0.34.6.0.11.2.109
|
0..1 O
|
Template-Spezifikation
|
|
|
|
Anamnese Entry - Labor und Mikrobiologie
|
1.2.40.0.34.6.0.11.3.12
|
1..* M
|
Template-Spezifikation
|
|
|
|
|
Anamnese Observation - Labor und Mikrobiologie
|
1.2.40.0.34.6.0.11.3.164
|
1..* M
|
Template-Spezifikation
|
|
|
Anamnese - Labor und Mikrobiologie - uncodiert
|
1.2.40.0.34.6.0.11.2.111
|
0..1 O
|
Template-Spezifikation
|
| Die "Angeforderte Untersuchung" kann codiert oder uncodiert angegeben werden:
|
|
|
Angeforderte Untersuchungen - codiert
|
1.2.40.0.34.6.0.11.2.15
|
0..1 O
|
Template-Spezifikation
|
|
|
|
Angeforderte Untersuchung Entry
|
1.2.40.0.34.6.0.11.3.169
|
1..* M
|
Template-Spezifikation
|
|
|
Angeforderte Untersuchungen - uncodiert
|
1.2.40.0.34.6.0.11.2.112
|
0..1 O
|
Template-Spezifikation
|
| Probeninformation (Specimen Section)
|
1.2.40.0.34.6.0.11.2.93
|
0..1 C1
|
Template-Spezifikation
|
|
|
Probeninformation (Specimen Entry)
|
1.2.40.0.34.6.0.11.3.160
|
1..1 M
|
Template-Spezifikation
|
|
|
|
Specimen Collection
|
1.2.40.0.34.6.0.11.3.161
|
1..* M
|
Template-Spezifikation
|
|
|
|
|
Specimen Received
|
1.2.40.0.34.6.0.11.3.162
|
0..1 O
|
Template-Spezifikation
|
| Laboratory Specialty Section
|
1.2.40.0.34.6.0.11.2.102
|
1..* M
|
Template-Spezifikation
|
|
|
Laboratory Report Data Processing Entry
|
1.2.40.0.34.6.0.11.3.25
|
1..1 M
|
Struktur des Laboratory Report Data Processing Entries
|
| Befundbewertung
|
1.2.40.0.34.6.0.11.2.103
|
0..1 O
|
Template-Spezifikation
|
|
|
Comment Entry
|
1.2.40.0.34.6.0.11.3.11
|
1..1 R
|
Template-Spezifikation
|
| Beilagen
|
1.2.40.0.34.6.0.11.2.71
|
0..1 O
|
Template-Spezifikation
|
| Abschließende Bemerkung
|
1.2.40.0.34.6.0.11.2.70
|
0..1 O
|
Template-Spezifikation
|
1 Die Dokumentation des Untersuchungsmaterials kann auf zwei Arten erfolgen:
- Enthält ein Befund nur einen Befundbereich ("Laboratory Specialty Section"), so kann die Codierung innerhalb der einen Section erfolgen.
ODER
- Bei Verwendung von mehreren Befundbereichen in einem Befund kann es zu Überschneidungen der Untersuchungsmaterialien kommen (ein spezielles Untersuchungsmaterial kann in zwei Befundbereichen analysiert werden). Die CDA Level 3 Codierung eines Untersuchungsmaterials darf jedoch nur einmal im gesamten Befund erfolgen. Daher sind die Informationen zu den Untersuchungsmaterialien in einer eigenen, führenden "Probeninformation (Specimen Section)" zu codieren.
|
[Tabelle 6]:
Übersichtstabelle der CDA Strukturen des Bodys des Laborbefundes
14.3.2 Mikrobiologiebefund
Derzeit werden Mikrobiologiebefunde mit mehreren Untersuchungsmaterialien NICHT unterstützt. Das bedeutet, dass in einem Mikrobiologiebefund
nur genau ein Untersuchungsmaterial dokumentiert werden darf. Falls mehrere Untersuchungsmaterialien zu einem Auftrag eingesendet wurden, müssen diese auf mehrere Befunde aufgeteilt werden! Die Information kann in der Section Befundbewertung festgehalten werden.
Hintergrund: Die maschinenlesbare Angabe der Abhängigkeiten der Untersuchungsmaterialien und der zugehörigen Analysen würde sowohl für Dokumentenersteller als auch für die Nutzer der maschinenlesbaren Daten überaus komplex und daher zu fehleranfällig werden.
Das für die Codierung des Untersuchungsmaterials erforderliche Template "Specimen Collection" kann entweder unter dem Template "Probeninformation (Specimen Entry)" oder dem Template "Laboratory Report Data Processing Entry" platziert werden.
:
Übersichtstabelle der CDA Strukturen des Bodys des Mikrobiologiebefundes
14.3.3 Struktur: Laboratory Report Data Processing Entry
[Tabelle 8]:
Übersichtstabelle der CDA Strukturen des Laboratory Report Data Processing Entries
14.4 CDA Templates
14.4.1 IHE PaLM TF-3 Konformität
Der vorliegende Leitfaden baut auf den Definitionen des "IHE Pathology and Laboratory Medicine (PALM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019"[22] auf, welche durch diesen Leitfaden weiter eingeschränkt werden. Dadurch erhalten die entsprechenden Templates ihre Gültigkeit und sind aus Konformitätsgründen bei Komponenten, welche über eine entsprechende Definition verfügen, auch anzugeben.
Entsprechend den Vorgaben des IHE Frameworks für Labor sind für Personen und Organisationen die Angabe eines Namens ("name"-Element), einer Adresse ("addr"-Element) und einer Telekom-Verbindung ("telecom"-Element) verpflichtend. Diese können jedoch mit einem nullFlavor versehen werden.
14.4.2 Document Level Templates
14.4.2.1 Laborbefund
| Id | 1.2.40.0.34.6.0.11.0.11 | Gültigkeit ab | 2020‑08‑25 14:35:13 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 3.0.0+20211214 |
|---|
| Name | atlab_document_Laborbefund | Bezeichnung | Laborbefund |
|---|
| Beschreibung | Der Laborbefund erlaubt es, beliebige Befundbereiche, Befundgruppen und deren Ergebnisse im Rahmen eines Dokumentes zu übermitteln. Dabei kann es vorkommen, dass der Befund auch nur einen bestimmten Befundbereich (z.B. Hämatologie) oder verschiedene Befundbereiche enthält. Diesem Umstand wird durch die Angabe der enthaltenen Befundbereiche bei der Registrierung
des Laborbefundes in den XDS-Metadaten Rechnung getragen. Durch die Registrierung der in dem Laborbefund enthaltenen Befundbereiche über die serviceEvents in den XDS-Metadaten ("eventCodeList") sind auch Detailbefunde in der ELGA einfach auffindbar. |
|
| Kontext | Pfadname / |
|---|
| Klassifikation | CDA Document Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 42 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.1.10 | Inklusion |  Document Realm (1.0.1+20230717) Document Realm (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.30 | Inklusion |  Document TypeId (1.0.0+20210219) Document TypeId (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.1 | Inklusion |  Document Id (1.0.0+20210219) Document Id (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.45 | Inklusion |  Document StatusCode (1.0.1+20210624) Document StatusCode (1.0.1+20210624) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.46 | Inklusion |  Document TerminologyDate (1.0.0+20210219) Document TerminologyDate (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.44 | Inklusion |  Document PracticeSettingCode (1.1.0+20210303) Document PracticeSettingCode (1.1.0+20210303) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.11 | Inklusion |  Document Effective Time (1.0.1+20230717) Document Effective Time (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.12 | Inklusion |  Document Confidentiality Code (1.0.2+20230717) Document Confidentiality Code (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.13 | Inklusion |  Document Language (1.0.0+20210219) Document Language (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.15 | Inklusion |  Document Set Id and Version Number (1.0.0+20210219) Document Set Id and Version Number (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.3 | Inklusion |  Record Target (1.2.1) Record Target (1.2.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.2 | Inklusion |  Author (1.0.3+20230717) Author (1.0.3+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.22 | Inklusion |  Data Enterer (1.0.1+20230717) Data Enterer (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.4 | Inklusion |  Custodian (1.0.1+20211213) Custodian (1.0.1+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.24 | Inklusion |  Information Recipient (1.0.0+20210219) Information Recipient (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.5 | Inklusion |  Legal Authenticator (1.0.0+20210219) Legal Authenticator (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.49 | Inklusion |  Laboratory Results Validator (1.0.0+20211213) Laboratory Results Validator (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.42 | Inklusion |  Participant Auftraggeber / Ordering Provider (1.1.0+20211213) Participant Auftraggeber / Ordering Provider (1.1.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.20 | Inklusion |  Participant Fachlicher Ansprechpartner (1.0.2+20210803) Participant Fachlicher Ansprechpartner (1.0.2+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.23 | Inklusion |  Participant Hausarzt (1.0.1+20210803) Participant Hausarzt (1.0.1+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.27 | Inklusion |  Participant Auskunftsberechtigte Person (Notfallkontakt) (1.0.2+20210803) Participant Auskunftsberechtigte Person (Notfallkontakt) (1.0.2+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.25 | Inklusion |  Participant Angehoerige (1.0.1+20210803) Participant Angehoerige (1.0.1+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.26 | Inklusion |  Participant Versicherung (1.0.0+20210219) Participant Versicherung (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.29 | Inklusion |  Participant Betreuungsorganisation (1.0.0+20210219) Participant Betreuungsorganisation (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.28 | Inklusion |  Participant Weitere Behandler (1.0.0+20210219) Participant Weitere Behandler (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.9 | Inklusion |  In Fulfillment Of (1.0.1+20210628) In Fulfillment Of (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.48 | Inklusion |  Documentation Of Service Event - Labor und Mikrobiologie (1.0.0+20211213) Documentation Of Service Event - Labor und Mikrobiologie (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.14 | Inklusion |  Document Replacement - Related Document (1.0.1+20210628) Document Replacement - Related Document (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.50 | Inklusion |  Component Of - Encompassing Encounter with id (1.0.1+20230717) Component Of - Encompassing Encounter with id (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.69 | Containment |  Brieftext (1.0.1+20210628) Brieftext (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.6 | Containment |  Überweisungsgrund - codiert (1.0.0+20211213) Überweisungsgrund - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.114 | Containment |  Überweisungsgrund - uncodiert (1.0.0+20211213) Überweisungsgrund - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.109 | Containment |  Anamnese - Labor und Mikrobiologie - codiert (1.0.0+20211213) Anamnese - Labor und Mikrobiologie - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.111 | Containment |  Anamnese - Labor und Mikrobiologie - uncodiert (1.0.0+20211213) Anamnese - Labor und Mikrobiologie - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.15 | Containment |  Angeforderte Untersuchungen - codiert (1.0.0+20211213) Angeforderte Untersuchungen - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.112 | Containment |  Angeforderte Untersuchungen - uncodiert (1.0.0+20211213) Angeforderte Untersuchungen - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.93 | Containment |  Probeninformation (Specimen Section) (1.0.0+20211213) Probeninformation (Specimen Section) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.102 | Containment |  Laboratory Specialty Section (1.0.0+20211213) Laboratory Specialty Section (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.103 | Containment |  Befundbewertung (1.0.0+20211213) Befundbewertung (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.71 | Containment |  Beilagen (1.0.2+20230717) Beilagen (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.70 | Containment |  Abschließende Bemerkung (1.0.1+20210628) Abschließende Bemerkung (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.33 | Inklusion |  Stylesheet Test eBefund (1.0.1+20210628) Stylesheet Test eBefund (1.0.1+20210628) | DYNAMIC |
|
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCCLIN |  | @moodCode
|
| cs | 0 … 1 | F | EVN | | | Constraint | Entsprechend den Vorgaben des IHE Frameworks für Labor sind für Personen und Organisationen die Angabe eines Namens ("name"-Element), einer Adresse ("addr"-Element) und einer Telekom-Verbindung ("telecom"-Element) verpflichtend. Diese können jedoch mit einem nullFlavor versehen werden. | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.10 Document Realm (DYNAMIC) |  | hl7:realmCode
|
| CS | 1 … 1 | M | Hoheitsbereich des Dokuments.
Fester Wert: @code = AT (aus Value Set „ELGA_RealmCode“) | |  |  | @code
|
| | 1 … 1 | F | AT | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.30 Document TypeId (DYNAMIC) |  | hl7:typeId
|
| II | 1 … 1 | M | Dokumentformat CDA R2
| |  |  | @root
|
| uid | 1 … 1 | F | 2.16.840.1.113883.1.3 |  |  | @extension
|
| st | 1 … 1 | F | POCD_HD000040 |  | hl7:templateId
|
| II | 1 … 1 | M | Fixe OID für alle Dokumente, die in der Governance-Gruppe "eHealth Austria" abgestimmt werden und von einem zentralen Art-Decor-Repository abgeleitet werden (AT-CDA-BBR). | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.0.1 |  | hl7:templateId
|
| II | 1 … 1 | M | OID des Implementierungsleitfadens "Labor- und Mikrobiologiebefund" (Dokument-OID). Dient als informative Referenz. | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.7.4.9.3 |  | hl7:templateId
|
| II | 1 … 1 | M | OID des Art-Decor-Templates für das Dokument "Laborbefund" (Document Level Template für Schematron) | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.0.11 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.2.3 templateId
Im Grunde folgt dieser Leitfaden den Vorgaben der IHE. Die Codierung der "Laboratory Specialty Section" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3 | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.1 Document Id (DYNAMIC) |  | hl7:id
|
| II | 1 … 1 | M | Dokumenten-Id des CDA-Dokuments.
Es MUSS eine gültige und innerhalb des ID-Pools eindeutige Dokumenten-ID angegeben werden.
Grundsätzlich sind die Vorgaben gemäß „Identifikations-Elemente“ zu befolgen.
| |  |  | @root
|
| uid | 1 … 1 | R | |  | hl7:code
|
| CE | 1 … 1 | M | Für den Laborbefund ist sowohl als Dokumententyp (/ClinicalDocument/code) als auch als Dokumentenklasse (/ClinicalDocument/code/translation) "11502-2 - Laboratory report" anzugeben.
↔ Hinweis zum XDS-Mapping:
- Das code-Element wird in das XDS-Metadaten-Attribut XDSDocumentEntry.typeCode übernommen.
- Das translation-Element wird in das XDS-Metadaten-Attribut XDSDocumentEntry.classCode übernommen.
| |  |  | @codeSystemName
|
| st | 0 … 1 | F | LOINC |  |  | @code
|
| CONF | 1 … 1 | F | 11502-2 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.1 (LOINC) |  |  | @displayName
|
| 1 … 1 | F | Laboratory report |  |  | hl7:translation
|
| CD | 1 … 1 | M | Fixe Dokumentenklasse "11502-2 - Laboratory report" | | | st | 0 … 1 | F | LOINC | | CONF | 1 … 1 | F | 11502-2 | | 1 … 1 | F | 2.16.840.1.113883.6.1 (LOINC) | | 1 … 1 | F | Laboratory report |  | hl7:title
|
| ST | 1 … 1 | M | Der Titel des CDA Dokuments für den lesenden Empfänger. MUSS die Art des Dokuments (Dokumenttyp) widerspiegeln.
Zum Beispiel "Laborbefund". | | | Eingefügt | 0 … 1 | C | von 1.2.40.0.34.6.0.11.1.45 Document StatusCode (DYNAMIC) | | | Constraint | Labor- und Mikrobiologiebefunde sind grundsätzlich abgeschlossene bzw. "fertige" Dokumente - in diesen Fällen erübrigt sich die Angabe eines Status.
Befunde, in denen die Ergebnisse bestimmter Analysen noch ausständig sind ("Wert folgt"), MÜSSEN den Dokumentenstatus "active" erhalten und das Ergebnis der ausständigen Analyse MUSS den SNOMED CT Code "255599008 - Incomplete (qualifier value)" erhalten. |  | sdtc:statusCode
|
| CS | 0 … 1 | C |
Status eines Dokuments.
e-Befunde sind grundsätzlich abgeschlossene bzw. "fertige" ("completed") Dokumente, daher entfällt die Angabe eines Status. In folgenden Ausnahmen SOLL der Status eines Dokuments wie folgt angegeben werden:
- “active”: z.B. wenn bekannt ist, dass Updates folgen werden: Etwa für "vorläufige ärztliche Entlassungsbriefe" oder Laborbefunde, für die noch Ergebnisse einzelner Analysen ausständig sind
- “nullified”: z.B. für Dokumente, die gemäß Anwendungsfall "Storno von ELGA-Dokumenten" storniert werden, wobei zusätzlich ein letztes Dokument mit Storniert-Status in der Versionskette registriert wird.
↔ Hinweis zum XDS-Mapping: Der Status wird nicht in die XDS-Metadaten übernommen!
| | | | Constraint |
Zulässige Werte für sdtc:statusCode/@code sind "active" und "nullified"
| | | CONF | | @code muss "nullified" sein | | oder | | @code muss "active" sein |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.46 Document TerminologyDate (DYNAMIC) |  | hl7at:terminologyDate
|
| TS.DATE.FULL | 1 … 1 | M | Das Terminologie-Datum des Dokumentes
Das Datum, an dem die lokal zur Implementierung verwendeten Value Sets mit dem österreichischen Terminologieserver abgeglichen wurden, wird hier angegeben.
| | | | Constraint | Das Datum der letzten Terminologie-Aktualisierung MUSS entsprechend klassischer HL7 V3 Notation im Format "YYYYMMDD" angegeben werden.
Beispiel: 20200527
|  | hl7at:formatCode
|
| CD | 1 … 1 | M | ↔ Hinweis zum XDS-Mapping:
@code wird in das XDS-Attribut XDSDocumentEntry.formatCode übernommen. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | ELGA_FormatCode |  |  | @code
|
| CONF | 1 … 1 | F | urn:hl7-at:lab:3.0.0+20211214 |  |  | @codeSystem
|
| 1 … 1 | F | 1.2.40.0.34.5.37 |  |  | @displayName
|
| 1 … 1 | F | HL7 Austria Labor- und Mikrobiologiebefund 3.0.0+20211214 | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.44 Document PracticeSettingCode (DYNAMIC) |  | hl7at:practiceSettingCode
|
| CD | 1 … 1 | M | Die fachliche Zuordnung des Dokumentes | |  |  | @displayName
|
| | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.75 ELGA_PracticeSetting (DYNAMIC) |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.11 Document Effective Time (DYNAMIC)
Angabe des medizinisch zutreffendsten Datums - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc. |  | hl7:effectiveTime
|
| TS.AT.TZ | 1 … 1 | M |
Relevantes Datum des Dokuments.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
| | | |  | | at-cda-bbr-dataelement-11 |  Erstellungsdatum Erstellungsdatum |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.12 Document Confidentiality Code (DYNAMIC) |  | hl7:confidentialityCode
|
| CE | 1 … 1 | M | Vertraulichkeitscode des Dokuments aus Value Set „ELGA_Confidentiality“. | | | |  | | at-cda-bbr-dataelement-13 |  Vertraulichkeitscode Vertraulichkeitscode |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @codeSystemName
|
| st | 1 … 1 | F | HL7:Confidentiality | | | Constraint | Für ELGA-Dokumente ist ausschließlich "N" erlaubt!
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.13 Document Language (DYNAMIC) |  | hl7:languageCode
|
| CS.LANG | 1 … 1 | M | Sprachcode des Dokuments.
| | | |  | | at-cda-bbr-dataelement-14 |  Sprachcode Sprachcode |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @code
|
| cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.10 ELGA_LanguageCode (DYNAMIC) |
| | | Constraint | Für ELGA ist in @code für CDA und Ableitungen in die XDSDocumentEntry-Metadaten derzeit ausschließlich der Wert "de-AT" zulässig.
Für eHealth und zukünftige Versionen der ELGA Leitfäden können weitere Sprachcodes erlaubt werden. | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.15 Document Set Id and Version Number (DYNAMIC) |  | hl7:setId
|
| II | 1 … 1 | M |
Eindeutige Id des Dokumentensets. Diese bleibt über alle Versionen der Dokumente gleich (initialer Wert bleibt erhalten). Die setId SOLL unterschiedlich zur clinicalDocument.id sein. ↔ Hinweis zum XDS-Mapping: Dieses Element wird ins XDS-Attribut referenceIdList
("urn:elga:iti:xds:2014:ownDocument_setId") gemappt. Hinweis: Bestimmte Systeme, die bei der Übernahme der setId in die XDS-Metadaten mit dem V2-Datentyp CX arbeiten, könnten ein Problem mit @extension-Attributen haben, die länger als 15 Zeichen sind. | |  | hl7:versionNumber
|
| INT.NONNEG | 1 … 1 | M | Versionsnummer des Dokuments, wird bei neuen Dokumenten mit 1 festgelegt.
Die versionNumber ist eine natürliche Zahl für die fortlaufende Versionszählung. Mit einer neuen Version wird diese Zahl hochgezählt, während die setId gleich bleibt.
| |  |  | @value
|
| int | 1 … 1 | R | Versionsnummer als positive ganze Zahl.
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.3 Record Target (DYNAMIC) |  | hl7:recordTarget
|
| | 1 … 1 | M | Komponente für die Patientendaten. | | | |  | | at-cda-bbr-dataelement-64 |  Patient Patient |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | RCT |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:patientRole
|
| | 1 … 1 | M | Patientendaten.
| | | cs | 0 … 1 | F | PAT | | II | 2 … * | R | Patientenidentifikatoren | | | |  | | at-cda-bbr-dataelement-193 |  EKVK EKVK |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-65 |  LokaleID LokaleID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-66 |  SVNr SVNr |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-67 |  bPK-GH bPK-GH |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Hinweis: Die Reihenfolge der id-Elemente MUSS unbedingt eingehalten werden!
* id[1] Identifikation des Patienten im lokalen System (1..1 M)
↔ Hinweis zum XDS-Mapping: Das Element id[1] wird ins XDS-Attribut sourcePatientId gemappt.
* id[2] Sozialversicherungsnummer des Patienten (1..1 R):
- @root: OID der Liste aller österreichischen Sozialversicherungen, fester Wert: 1.2.40.0.10.1.4.3.1 (1..1 M)
- @extension: Vollständige Sozialversicherungsnummer des Patienten (10 Stellen) (1..1 M)
- @assigningAuthorityName: Fester Wert: Österreichische Sozialversicherung (0..1 O)
Zugelassene nullFlavor:
- NI … Patient hat keine Sozialversicherungsnummer (z.B. Ausländer)
- UNK … Patient hat eine Sozialversicherungsnummer, diese ist jedoch unbekannt
* id[@root="1.2.40.0.10.2.1.1.149"] Bereichsspezifisches Personenkennzeichen (0..1 O):
- @root : OID der österreichischen bPK, fester Wert: 1.2.40.0.10.2.1.1.149 (1..1 M)
- @extension : bPK des Patienten: concat(Bereichskürzel, ":", bPK) (Base64,28 Zeichen). Typischerweise bPK-GH (Gesundheit). Kann im Zusammenhang mit E-ID auch andere Bereichskürzel tragen.
Anmerkung : Das bPK dient ausschließlich technisch der Zuordnung der elektronischen Identität und darf daher weder angezeigt werden noch am Ausdruck erscheinen noch in allfälligen Downloads enthalten sein (1..1 M)
- @assigningAuthorityName : Fester Wert: Österreichische Stammzahlenregisterbehörde (0..1 O)
* id[@root="1.2.40.0.34.4.21"] Europäische Krankenversicherungskarte kurz (0..1 O):
- @root: OID der EKVK, fester Wert: 1.2.40.0.34.4.21 (1..1 M) - @extension: Datenfeld 3 der EKVK - @assigningAuthorityName : Fester Wert: Nationaler Krankenversicherungsträger (0..1 O) * id[@root="1.2.40.0.10.1.4.3.8"] Europäische Krankenversicherungskarte lang (0..1 O):
- @root: OID der EKVK, fester Wert: 1.2.40.0.34.4.21 (1..1 M) - @extension: Datenfelder der EKVK nach folgender Bildungsvorschrift: concat(Feld 6,"^",Feld 7,"^",Feld 8,"^",Feld 9) wobei Feld 6 "Persönliche Kennnummer" angegeben sein MUSS (1..1 M). Die übrigen Datenfelder sind optional (0..1 O). In Feld 9 MUSS die Datumsangabe im Format YYYMMDD erfolgen.
- @assigningAuthorityName : Fester Wert: Nationaler Krankenversicherungsträger (0..1 O)
Grundsätzlich sind die Vorgaben gemäß „Identifikations-Elemente“ zu befolgen. | | | Beispiel | EKVK Beispiel-Max <id root="1.2.40.0.34.4.21" extension="123456789^1100-OEGK^800400010016^20251231"/> | | | Beispiel | EKVK Beispiel-Min <id root="1.2.40.0.34.4.21" extension="123456789"/> | | | 0 … 2 | R | Adresse des Patienten.
Es MUSS eine mögliche Adresse unterstützt werden. Spezielle Leitfäden (z.B. Entlassungsbrief Pflege) können es erforderlich machen, dass mehr als eine Adresse unterstützt werden muss.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | |  | | at-cda-bbr-dataelement-68 |  Adresse Adresse |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Werden mehrere gleichartige address-Elemente strukturiert (z.B. Home, Pflege), MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … * | R | Kontakt-Element. Grundsätzlich sind die Vorgaben gemäß „Kontaktdaten-Element“ zu befolgen.
| | | |  | | at-cda-bbr-dataelement-72 |  Kontaktdaten Kontaktdaten |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | url | 1 … 1 | R |
Formatkonvention siehe „telecom-Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß Value-Set „ELGA_URLScheme“
| | cs | 0 … 1 | | Bedeutung des angegebenen Kontakts (z.B Heim, Arbeitsplatz), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | | 1 … 1 | M | Name des Patienten.
Für den Namen ist verpflichtend Granularitätsstufe 2 („strukturierte Angabe des Namens‘‘) anzuwenden!
Grundsätzlich sind die Vorgaben gemäß „Namen-Elemente von Personen PN“ zu befolgen. | | | |  | | at-cda-bbr-dataelement-70 |  Name Name |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | cs | 0 … 1 | F | PSN | | cs | 0 … 1 | F | INSTANCE | | PN | 1 … 1 | M | Namen-Element (Person) | | | cs | 0 … 1 | |
Die genaue Bedeutung des angegebenen Namens, z.B. Angabe eines Künstlernamens mit „A" für „Artist“.
Zulässige Werte gemäß Value Set „ELGA_EntityNameUse“.
Wird kein @use Attribut angegeben, gilt der Name als rechtlicher Name („L“).
| | ENXP | 0 … * | |
Beliebig viele Präfixe zum Namen, z.B. Akademische Titel
Achtung: Die Angabe der Anrede („Frau“, „Herr“), ist im CDA nicht vorgesehen!
| | | cs | 0 … 1 | |
Bedeutung eines prefix-Elements, z.B. Angabe eines akademischen mit "AC" für „Academic“.
Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier".
| | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | ENXP | 1 … * | M | Mindestens ein Hauptname (Nachname). | | | cs | 0 … 1 | | Bedeutung eines family-Elements, z.B. Angabe eines Geburtsnamen mit „BR" für „Birth“. Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier“. | | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | ENXP | 1 … * | M | Mindestens ein Vorname | | | cs | 0 … 1 | |
Die genaue Bedeutung eines given-Elements, beispielsweise dass das angegebene Element einen Geburtsnamen bezeichnet, z.B. BR („Birth“).
Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier“
| | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | ENXP | 0 … * | | Beliebig viele Suffixe zum Namen | | | cs | 0 … 1 | | Die genaue Bedeutung eines suffix-Elements, beispielsweise dass das angegebene Suffix einen akademischen Titel darstellt, z.B.: AC („Academic“).
Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier“. | | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | Auswahl | 1 … 1 | | Das "administrative Geschlecht" ist das soziale oder gesellschaftliche Geschlecht ("Gender"). Das administrative Geschlecht ist daher grundsätzlich getrennt von den biologischen Merkmalen der Person zu sehen. Grundsätzlich soll das administrative Geschlecht dem im
Zentralen Melderegister (ZMR) eingetragenen Geschlecht entsprechen. Über ein Translation-Element können weitere Angaben zum Geschlecht gemacht werden, wenn diese abweichend vom administrativen Geschlecht sind, z.B.: - Biologisches Geschlecht
- Geschlecht in der
Sozialversicherung
- Geschlecht für die Stations-/Bettenbelegung im Krankenhaus
Codierung des Geschlechts des Patienten aus ValueSet "ELGA_AdministrativeGender". - hl7:administrativeGenderCode[not(@nullFlavor)]
- hl7:administrativeGenderCode[@nullFlavor='UNK']
|  |  |  |  |  | hl7:administrativeGenderCode
|
| CE | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-74 |  Geschlecht Geschlecht |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | st | 1 … 1 | R | | | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.1 | | st | 0 … 1 | F | HL7:AdministrativeGender | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.4 ELGA_AdministrativeGender (DYNAMIC) |
| | CD | 0 … * | R | Über ein Translation-Element können weitere Angaben zum Geschlecht gemacht werden, wenn diese abweichend vom administrativen Geschlecht sind, z.B.: Biologisches Geschlecht, Geschlecht in der Sozialversicherung, Geschlecht für die Stations-/Bettenbelegung im Krankenhaus | | | st | 1 … 1 | R | | | | Beispiel | Beispiel für eine SNOMED CT Angabe <translation code="772004004" codeSystem="2.16.840.1.113883.6.96" displayName="Non-binary gender"/> |  |  |  |  |  | hl7:administrativeGenderCode
|
| CE | 0 … 1 | | Mittels nullFlavor="UNK" wird "Unbekannt" abgebildet. Dies schließt die Ausprägung "Keine Angabe" mit ein. | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … 1 | | Geburtsdatum des Patienten.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
- hl7:birthTime
- hl7:birthTime[@nullFlavor='UNK']
| | TS.AT.VAR | 0 … 1 | | | | | |  | | at-cda-bbr-dataelement-75 |  Geburtsdatum Geburtsdatum |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | TS.AT.VAR | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | BL | 0 … 1 | R | Kennzeichen, dass die Person verstorben ist. Kann alternativ zum Todesdatum angegeben werden, v.a. wenn der Todeszeitpunkt nicht bekannt ist. | | | |  | | at-cda-bbr-dataelement-192 |  Verstorben-Kennzeichen Verstorben-Kennzeichen |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | TS.AT.TZ | 0 … 1 | R | Todesdatum der Person. | | | |  | | at-cda-bbr-dataelement-191 |  Todesdatum Todesdatum |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | CE | 0 … 1 | R | Codierung des Familienstands des Patienten.
Zulässige Werte gemäß Value-Set „ELGA_MaritalStatus“ | | | |  | | at-cda-bbr-dataelement-98 |  Familienstand Familienstand |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.2 | | st | 1 … 1 | F | HL7:MaritalStatus | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.11 ELGA_MaritalStatus (DYNAMIC) |
|  |  |  |  | hl7:religiousAffiliationCode
|
| CE | 0 … 1 | R | Codierung des Religionsbekenntnisses des Patienten.
Zulässige Werte gemäß Value-Set „ELGA_ReligiousAffiliation“ | | | |  | | at-cda-bbr-dataelement-99 |  Religionsbekenntnis Religionsbekenntnis |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.2.16.1.4.1 | | st | 1 … 1 | F | HL7.AT:ReligionAustria | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.18 ELGA_ReligiousAffiliation (DYNAMIC) |
| | | | NP | Rasse des Patienten.
Darf nicht verwendet werden! | | | | | NP | Ethnische Zugehörigkeit des Patienten.
Darf nicht verwendet werden! | | | | 0 … * | R | Gesetzlicher Vertreter:
- Vorsorgebevollmächtigte/r (Bevollmächtigte/r durch Vorsorgevollmacht)
- Gewählte/r ErwachsenenvertreterIn
- Gesetzliche/r ErwachsenenvertreterIn
- Gerichtliche/r ErwachsenenvertreterIn (Sachwalter)
Der gesetzliche Vetreter kann entweder eine Person (guardianPerson) oder eine Organisation (guardianOrganization) sein.
Beim Patienten können optional ein oder mehrere gesetzliche Vetreter angegeben werden. Wenn ein gesetzliche Vetreter bekannt ist, SOLL diese Information auch angegeben werden.
| | | |  | | at-cda-bbr-dataelement-88 |  Gesetzlicher Vertreter Gesetzlicher Vertreter |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 0 … 1 | F | GUARD | | | 0 … 1 | R | Die Adresse des gesetzlichen Vertreters oder der Organisation.
Grundsätzlich sind die Vorgaben für „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | TEL.AT | 0 … * | R | Beliebig viele Kontaktdaten des gesetzlichen Vertreters als Person oder Organisation.
Grundsätzlich sind die Vorgaben gemäß „Kontaktdaten-Element“ zu befolgen.
| | | st | 1 … 1 | R |
Formatkonvention siehe „telecom-Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß Value-Set „ELGA_URLScheme“
| | set_cs | 0 … 1 | | Bedeutung des angegebenen Kontakts (z.B. Heim, Arbeitsplatz) Bsp: WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Angabe des gesetzlichen Vertreters als Person (guardianPerson in Granularitätsstufe 1 oder 2) ODER als Organisation (guardianOrganization)
Elemente in der Auswahl:- hl7:guardianPerson welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:guardianPerson welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
- hl7:guardianOrganization welches enthält Template 1.2.40.0.34.6.0.11.9.27 Organization Name Compilation (DYNAMIC)
| | | 0 … 1 | | Name des gesetzlichen Vertreters: Angabe in Granularitätsstufe 1
Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | | 0 … 1 | | Name des gesetzlichen Vertreters: Angabe in Granularitätsstufe 2
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | |  |  |  |  |  |  | hl7:guardianOrganization
|
| | 0 … 1 | R | Name des gesetzlichen Vertreters (Organisation)
Beinhaltet 1.2.40.0.34.6.0.11.9.27 Organization Name Compilation (DYNAMIC) | | | | 0 … 1 | R | Geburtsort des Patienten. | | | |  | | at-cda-bbr-dataelement-76 |  Geburtsort Geburtsort |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 0 … 1 | F | BIRTHPL | | | 1 … 1 | M | | | | cs | 0 … 1 | F | PLC | | cs | 0 … 1 | F | INSTANCE | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:addr welches enthält Template 1.2.40.0.34.6.0.11.9.10 Address Compilation Minimal (DYNAMIC)
- hl7:addr welches enthält Template 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC)
| | AD | 0 … 1 | | Die Adresse des Geburtsorts. Minimalangabe. Alle Elemente optional.
Beinhaltet 1.2.40.0.34.6.0.11.9.10 Address Compilation Minimal (DYNAMIC) | | | AD | 0 … 1 | | Die Adresse des Geburtsorts, struktuiert.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | |  |  |  |  | hl7:languageCommunication
|
| | 0 … * | R | Informationen bezüglich der Sprachfähigkeiten und Ausdrucksform des Patienten.
| | | |  | | at-cda-bbr-dataelement-100 |  Sprachfähigkeit Sprachfähigkeit |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | CS | 1 … 1 | M | Sprache, die vom Patienten zu einem bestimmten Grad beherrscht wird (geschrieben oder gesprochen).
In der Klasse languageCommunication können Informationen bezüglich der Sprachfähigkeiten und Ausdrucksform (z.B. gesprochen oder geschrieben) des Patienten angegeben werden.
Dieser Leitfaden schränkt die möglichen Werte für die Sprache auf Werte aus dem Value Set ELGA_HumanLanguage ein.
Die Gebärdensprache ist als eigene Sprache inkl. Ländercode anzugeben, mit der Ergänzung des Länder-/Regional-Codes (z.B. sgn-at), die Ausdrucksweise (MoodCode) wird in diesem Fall nicht angegeben (denn expressed / received signed wären redundant). | | | |  | | at-cda-bbr-dataelement-101 |  Sprache Sprache |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | Zulässige Werte gemäß Value-Set „ELGA_HumanLanguage“ aus Code-System „HL7:HumanLanguage 2.16.840.1.113883.6.121“
Gemäß IETF / RFC 3066 enthält es ein bestimmtes Subset von Codes aus ISO 639-1 und ISO 639-2 (also zwei- und dreistellige Sprachcodes). Gemäß RFC 3066 ist es zulässig, eine Angabe der landestypischen Ausprägung der Sprache nach einem Bindestrich anzufügen. Das Land wird dabei nach ISO 3166-1 Alpha 2 angegeben. Dies MUSS bei der Auswertung des languageCodes berücksichtigt und toleriert werden.
| | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.173 ELGA_HumanLanguage (DYNAMIC) |
| | CE | 0 … 1 | C | Ausdrucksform der Sprache.
Zulässige Werte gemäß Value-Set „ELGA_LanguageAbilityMode“
| | | cs | 1 … 1 | R | | | st | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.60 | | st | 0 … 1 | F | HL7:LanguageAbilityMode | | | Constraint | Bei Strukturierung einer Gebärdensprache ist dieses Element NICHT ERLAUBT, NP [0..0] und MUSS daher komplett entfallen | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.175 ELGA_LanguageAbilityMode (DYNAMIC) |
|  |  |  |  |  | hl7:proficiencyLevelCode
|
| CE | 0 … 1 | R | Grad der Sprachkenntnis in der Sprache.
Zulässige Werte gemäß Value-Set „ELGA_ProficiencyLevelCode“
| | | |  | | at-cda-bbr-dataelement-102 |  Grad der Sprachkenntnis Grad der Sprachkenntnis |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | st | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.61 | | st | 0 … 1 | F | HL7:LanguageAbilityProficiency | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.174 ELGA_ProficiencyLevelCode (DYNAMIC) |
| | BL | 0 … 1 | R | Kennzeichnung, ob die Sprache in der angegebenen Ausdrucksform vom Patienten bevorzugt wird. | | | |  | | at-cda-bbr-dataelement-103 |  Sprachpräferenz Sprachpräferenz |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Schematron assert | role | error | | | | test | not(hl7:id[1]/@nullFlavor) | | | | Meldung | Die Verwendung von id/@nullFlavor ist an dieser Stelle NICHT ERLAUBT. | | | | Schematron assert | role | error | | | | test | not(hl7:id[2]/@nullFlavor) or (hl7:id[2][@nullFlavor='UNK'] or hl7:id[2][@nullFlavor='NI']) | | | | Meldung | Zugelassene nullFlavor sind "NI" und "UNK" | | | Eingefügt | 1 … * | M | von 1.2.40.0.34.6.0.11.1.2 Author (DYNAMIC) |  | hl7:author
|
| | 1 … * | M | Verfasser des Dokuments.
| |  |  | @typeCode
|
| cs | 0 … 1 | F | AUT |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:functionCode
|
| CE (extensible) | 0 … 1 | R |
Funktionscode des Verfassers des Dokuments, z.B: „Diensthabender Oberarzt“, „Verantwortlicher Arzt für Dokumentation“,„Stationsschwester“.
Eigene Codes und Bezeichnungen können verwendet werden.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | Auswahl | 1 … 1 | |
Der Zeitpunkt, zu dem das Dokument verfasst bzw. inhaltlich fertiggestellt wurde.
Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='UNK']
| | TS.AT.TZ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | TS.AT.TZ | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK |  |  | hl7:assignedAuthor
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ASSIGNED | | Auswahl | 1 … * | |
Identifikation des Verfassers des Dokuments im lokalen System des/der datenerstellenden Gerätes/Software.
ODER Identifikation des/der datenerstellenden Gerätes/Software.
Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | | Constraint | Zugelassene nullFlavor:
- NI ….... Person hat keine ID / Gerät/Software hat keine ID
- UNK … Person hat eine ID, diese ist jedoch unbekannt / Gerät/Software hat eine ID, diese ist jedoch unbekannt
| | II | 0 … * | |
Identifikation des Verfassers des Dokuments im lokalen System des/der datenerstellenden Gerätes/Software.
ODER Identifikation des/der datenerstellenden Gerätes/Software.
| | | wo [not(@nullFlavor)] | | | II | 0 … 1 | | | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | CE | 0 … 1 | R |
Angabe der Fachrichtung des Verfassers des Dokuments („Sonderfach“ gem. Ausbildungsordnung), z.B: „Facharzt/Fachärztin für Gynäkologie“.
Wenn ein Autor mehreren ärztlichen Sonderfächern zugeordnet ist, kann das anzugebende Sonderfach gewählt werden. Additivfächer werden nicht angegeben.
| | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.6 ELGA_AuthorSpeciality (DYNAMIC) |
| | TEL.AT | 0 … * | |
Kontaktdaten des Verfassers des Dokuments.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:assignedPerson welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
- hl7:assignedAuthoringDevice welches enthält Template 1.2.40.0.34.6.0.11.9.18 Device Compilation (DYNAMIC)
| | | 0 … 1 | |
Personendaten des Verfassers des Dokuments.
Grundsätzlich sind die Vorgaben für „Personen-Element“ zu befolgen, name-Element ist hier Mandatory.
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | |  |  |  |  | hl7:assignedAuthoringDevice
|
| | 0 … 1 | | Datenerstellende/s Software/Gerät
Beinhaltet 1.2.40.0.34.6.0.11.9.18 Device Compilation (DYNAMIC) | |  |  |  | hl7:representedOrganization
|
| | 1 … 1 | M | Organisation, in deren Auftrag der Verfasser des Dokuments die Dokumentation verfasst hat.
↔ Hinweis zum XDS-Mapping: Da manche offiziellen Bezeichnungen von GDA sehr lang werden können, SOLL das name Element einer möglichst eindeutigen Kurzbezeichnung der Organisation entsprechen (im GDA-I im Tag description enthalten). Bei größeren Organisationen SOLL zusätzlich die Abteilung angegeben werden, damit die Zuordnung für den Leser einfacher wird.
Beispiel: Statt "Allgemeines Krankenhaus der Stadt Wien-Medizinischer Universitätscampus" --> "Wien AKH" bzw. "Wien AKH - Augenambulanz"
Beinhaltet 1.2.40.0.34.6.0.11.9.5 Organization Compilation with id, name (DYNAMIC) | | | | Constraint |
- id MUSS der OID der Organisation aus dem GDA-Index entsprechen.
- name SOLL der Kurzbezeichnung im GDA-I entsprechen (sofern vorhanden)
- Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden., z.B.: „Amadeus Spital, Chirurgische Abteilung“
-
Ausnahme: Wenn als Author ein/e Software/Gerät fungiert und keine OID aus dem GDA-I angegeben werden kann, MÜSSEN die Angaben der Organisation des Geräte-/Software-Betreibers oder Herstellers entsprechen.
| | | Schematron assert | role | error | | | | test | count(hl7:author/hl7:assignedAuthor/hl7:assignedPerson)>0 | | | | Meldung | Es MUSS immer zumindest eine Person als Autor angeführt sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.22 Data Enterer (DYNAMIC) |  | hl7:dataEnterer
|
| | 0 … 1 | |
z.B. Schreibkraft, Medizinische Dokumentationsassistenz
| | | |  | | at-cda-bbr-dataelement-16 |  Schreibkraft Schreibkraft |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | ENT |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:time
|
| TS.AT.TZ | 0 … 1 | R |
Der Zeitpunkt zu dem die Daten dokumentiert wurden.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-17 |  Zeitpunkt des Schreibens Zeitpunkt des Schreibens |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | hl7:assignedEntity
|
| | 1 … 1 | M | Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.4 Custodian (DYNAMIC) |  | hl7:custodian
|
| | 1 … 1 | M | Verwahrer des Dokuments. | | | |  | | at-cda-bbr-dataelement-24 |  Verwahrer Verwahrer |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | CST |  |  | hl7:assignedCustodian
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ASSIGNED |  |  |  | hl7:representedCustodianOrganization
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 1 … * | M | Identifikation des Verwahrers des Dokuments. Wenn dieser im GDA-I angeführt ist, ist die entsprechende OID zu verwenden.
Grundsätzlich sind die Vorgaben für „Identifikations-Elemente“ zu befolgen. | | | ON | 1 … 1 | M | Name des Verwahrers des Dokuments (Organisation). Grundsätzlich sind die Vorgaben für „Namen-Elemente von Organisationen ON“ zu befolgen. | | | TEL.AT | 0 … * | | Kontaktdaten des Verwahrers des originalen Dokuments (Organisation). Grundsätzlich sind die Vorgaben für „Kontaktdaten-Elemente“ zu befolgen. | | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R | | | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 1 … 1 | M | Adresse des Verwahrers des Dokuments (Organisation). Grundsätzlich sind die Vorgaben für „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.24 Information Recipient (DYNAMIC) |  | hl7:informationRecipient
|
| | 0 … * | | Beabsichtiger Empfänger des Dokuments.
| | | |  | | at-cda-bbr-dataelement-26 |  Empfänger Empfänger |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | | Typ des Informationsempfängers, z.B: PRCP „Primärer Empfänger“.
Werden mehrere Empfänger angegeben, MUSS der primäre Empfänger über den typeCode definiert werden.
Hinweis: Das ist relevant, wenn Funktionen aus dem gerichteten Befundversand oder für den Briefdruck auf das Dokument angewendet werden. | | | CONF | | Der Wert von @typeCode muss gewählt werden aus dem Value Set 1.2.40.0.34.10.29 ELGA_InformationRecipientType (DYNAMIC) |
| | |  | | at-cda-bbr-dataelement-27 |  Empfänger Typ Empfänger Typ |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | hl7:intendedRecipient
|
| | 1 … 1 | M | | | | cs | 0 … 1 | | | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | II | 0 … * | | Identifikation des beabsichtigten Empfängers (Person).
Empfohlene Information für einen Empfänger ist die ID aus dem GDA-Index.
Grundsätzlich sind die Vorgaben für „Identifikations-Elemente“ zu befolgen. | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-28 |  ID des Empfängers ID des Empfängers |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | II | 0 … 1 | | NI … Person hat keine ID | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | UNK ... Person hat eine ID, diese ist jedoch unbekannt | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … 1 | | Personendaten des beabsichtigten Empfängers.
Empfehlung: Der Name des Empfängers und die Organisation, der er angehört, sollen in möglichst hoher Granularität angegeben werden. Aufgrund der gängigen Praxis kann als minimale Information für den Empfänger der unstrukturierte Name angegeben werden.
Grundsätzlich sind die Vorgaben gemäß Kapitel „Personen-Element“ zu befolgen. Elemente in der Auswahl:- hl7:informationRecipient[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:informationRecipient[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
|  |  |  |  | hl7:informationRecipient
|
| | … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | |  | | at-cda-bbr-dataelement-29 |  Name Name |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  |  |  | hl7:informationRecipient
|
| | … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | |  |  |  | hl7:receivedOrganization
|
| | 0 … 1 | R | Organisation, der der beabsichtigte Empfänger angehört, z.B.: „Ordination des empfangenden Arztes“.
Grundsätzlich sind die Vorgaben gemäß Kapitel „Organisations-Element“ zu befolgen. | | | |  | | at-cda-bbr-dataelement-30 |  Organisation Organisation |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.5 Legal Authenticator (DYNAMIC)
"Medizinischer Validator" oder der laborverantwortliche Arzt |  | hl7:legalAuthenticator
|
| | 1 … 1 | M | Hauptunterzeichner, Rechtlicher Unterzeichner
| | | |  | | at-cda-bbr-dataelement-1 |  Rechtlicher Unterzeichner Rechtlicher Unterzeichner |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | @typeCode
|
| cs | 0 … 1 | F | LA | | Auswahl | 1 … 1 | | Der Zeitpunkt, an dem das Dokument unterzeichnet wurde.
Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='UNK']
| | TS.AT.TZ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-5 |  Zeitpunkt der Unterzeichnung Zeitpunkt der Unterzeichnung |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | TS.AT.TZ | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK |  |  | hl7:signatureCode
|
| CS | 1 … 1 | M | Signaturcode gibt an, dass das Originaldokument unterzeichnet wurde.
| | | |  | | at-cda-bbr-dataelement-6 |  Signatur Signatur |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | CONF | 1 … 1 | F | S |  |  | hl7:assignedEntity
|
| | 1 … 1 | M | Personendaten des rechtlichen Unterzeichners.
Für den Namen ist verpflichtend Granularitätsstufe 2 ("strukturierte Angabe des Namens") anzuwenden!
Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.49 Laboratory Results Validator (DYNAMIC)
Validator (Authenticator) |  | hl7:authenticator
|
| | 0 … * | | (Weitere) validierende Person (=Mitunterzeichner), die das Dokument inhaltlich (medizinisch und technisch) freigibt. Es können mehrere Validatoren angegeben werden. Einer davon kann auch ident mit dem "rechtlichen Unterzeichner" (/ClinicalDocument/legalAuthenticator) sein. | |  |  | @typeCode
|
| cs | 0 … 1 | F | AUTHEN |  |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Results Validator | | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.49 |  |  | hl7:templateId
|
| II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.2.16 Laboratory Results Validator | | | uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.1.5 | | Auswahl | 1 … 1 | | Der Zeitpunkt, an dem das Dokument unterzeichnet wurde.
Grundsätzlich sind die Vorgaben gemäß für "Zeit-Elemente" zu befolgen. Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='UNK']
| | TS.AT.TZ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | TS.AT.TZ | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | |  |  | hl7:signatureCode
|
| CS | 1 … 1 | M | | | | CONF | 1 … 1 | F | S |  |  | hl7:assignedEntity
|
| | 1 … 1 | M | Personendaten des weiteren Unterzeichners.
Beinhaltet 1.2.40.0.34.6.0.11.9.41 Assigned Entity with id, name, addr and telecom (DYNAMIC) | | | Auswahl | 1 … 1 | | Auftraggeber / Ordering Provider
Elemente in der Auswahl:- hl7:participant eingefügt vom Template 1.2.40.0.34.6.0.11.1.42 Participant Auftraggeber / Ordering Provider (DYNAMIC)
- hl7:participant[@typeCode='REF'][@nullFlavor]
| | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.42 Participant Auftraggeber / Ordering Provider (DYNAMIC) |  |  | hl7:participant
|
| | 0 … 1 | | | | | cs | 1 … 1 | F | REF | | cs | 0 … 1 | F | OP | | II | 1 … 1 | M | Participant Auftraggeber / Ordering Provider | | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.42 | | II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.2.17 Ordering Provider | | | uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.1.6 | | Auswahl | 1 … 1 | | Das Auftragsdatum ist das Datum/Zeit, an dem der Auftrag vom Auftraggeber abgesendet wird. Das Auftragsdatum wird als "time"-Element beim Auftraggeber ausgeführt und ist verpflichtend anzugeben. Bei einer manuellen Erfassung eines Auftrags im Labor kann dieses als @nullFlavor="NA" ausgeführt werden. Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='NA']
| | IVL_TS | 0 … 1 | R | | | | wo [not(@nullFlavor)] | | | IVL_TS | 0 … 1 | R | | | | wo [@nullFlavor='NA'] | | | | 1 … 1 | M | | | | cs | 1 … 1 | F | PROV | | | Healthcare provider - Gesundheitsdienstanbieter
| | II | 1 … 1 | M | ID des Auftraggebers | | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:addr[not(@nullFlavor)] welches enthält Template 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC)
- hl7:addr[@nullFlavor='UNK']
| | AD | 0 … 1 | R | Adresse des Auftraggebers
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | AD | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:telecom[not(@nullFlavor)]
- hl7:telecom[@nullFlavor='UNK']
| | TEL.AT | 0 … * | R | Beliebig viele Kontaktdaten des Auftraggebers
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | | Schematron assert | role | error | | | | test | not(hl7:telecom[not(@nullFlavor)]) or not(hl7:telecom[@nullFlavor='UNK']) | | | | Meldung | telecom[@nullFlavor="UNK"] darf NUR angegeben werden, wenn KEIN befülltes "telecom"-Element vorhanden ist. | | | Auswahl | 1 … 1 | | Name des Auftraggebers.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
- hl7:associatedPerson[@nullFlavor]
| | | 0 … 1 | R | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | | | | | wo [@nullFlavor] | | | cs | 1 … 1 | F | UNK | | | 0 … 1 | R |
Organisation, der der Auftraggeber angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für "Organisations-Element" zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | |  |  | hl7:participant
|
| | 0 … 1 | | Auftraggeber nicht bekannt | | | wo [@typeCode='REF'] [@nullFlavor] | | | cs | 1 … 1 | F | REF | | cs | 1 … 1 | F | UNK | | | Beispiel | Auftraggeber nicht bekannt <participant typeCode="REF" nullFlavor="UNK">
<associatedEntity classCode="PROV"/></participant> | | | 1 … 1 | M | | | | cs | 1 … 1 | F | PROV |  | hl7:participant
|
| | | NP | Die Verwendung des ELGA participant-Elements, das den einweisenden/zuweisenden/überweisenden Arzt repräsentiert mit templateId 1.2.40.0.34.6.0.11.1.21 ist im Laborbefund NICHT ERLAUBT. | | | wo [@typeCode='REF'] [hl7:templateId/@root='1.2.40.0.34.6.0.11.1.21'] | | | Eingefügt | 0 … 1 | R | von 1.2.40.0.34.6.0.11.1.20 Participant Fachlicher Ansprechpartner (DYNAMIC)
Fachlicher Ansprechpartner
Es ist EMPFOHLEN, den fachlichen Ansprechpartner (Callback contact) im Labor- und Mikrobiologiebefund anzugeben.
|  | hl7:participant
|
| | 0 … 1 | R | Fachlicher Ansprechpartner
| | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.20']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | CALLBCK | | | Callback contact
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.20 |  |  | hl7:functionCode
|
| CE (extensible) | 0 … 1 | |
Optionale Angabe eines Funktionscodes des fachlichen Ansprechpartners, z.B: „Diensthabender Oberarzt“, „Verantwortlicher Arzt für Dokumentation“,„Stationsschwester“.
Eigene Codes und Bezeichnungen können verwendet werden.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 1 … 1 | R | |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | PROV | | |
Healthcare provider - Gesundheitsdiensteanbieter
| | CE | 0 … 1 | |
Optionale Angabe der Fachrichtung des fachlichen Ansprechpartners („Sonderfach“ gem. Ausbildungsordnung), z.B: „Facharzt/Fachärztin für Gynäkologie“.
Wenn ein fachlicher Ansprechpartner mehreren ärztlichen Sonderfächern zugeordnet ist, kann das anzugebende Sonderfach gewählt werden. Additivfächer werden nicht angegeben.
| | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.6 ELGA_AuthorSpeciality (DYNAMIC) |
| | AD | 0 … 1 | |
Adresse des Beteiligten.
Grundsätzlich sind die Vorgaben für "Adress-Elemente" zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 1 … * | M | Beliebig viele Kontaktdaten des Beteiligten. | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Es MUSS mindestens eine Telefonnummer angegeben werden. Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein. | | | 0 … 1 | R |
Name der Person
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | | 0 … 1 | R |
Organisation, der der Beteiligte angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für "Organisations-Element" zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | | Schematron assert | role | error | | | | test | not(hl7:participant[@typeCode='CALLBCK'][@nullFlavor]) | | | | Meldung | @nullFlavor ist für den fachlichen Ansprechpartner (participant[@typeCode='CALLBCK']) NICHT ERLAUBT. Sollten keine Informationen vorliegen, soll das Element entfallen. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.23 Participant Hausarzt (DYNAMIC) |  | hl7:participant
|
| | 0 … 1 | | Beteiligter (Hausarzt). | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.23']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND | | | In indirektem Bezug.
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.23 |  |  | hl7:functionCode
|
| CE | 1 … * | M |
Funktionscode des Beteiligten
| | | cs | 1 … 1 | F | PCP | | oid | 1 … 1 | F | 2.16.840.1.113883.5.88 | | st | 1 … 1 | F | HL7:ParticipationFunction |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | PROV | | | Healthcare provider - Gesundheitsdiensteanbieter.
| | Auswahl | 0 … * | |
Identifikation des Beteiligten (Person) aus dem GDA-Index.
Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | | Constraint |
Zugelassene nullFlavor:
- NI … Organisation hat keine ID
- UNK … Organisation hat eine ID, diese ist jedoch unbekannt
| | II | 0 … * | | | | | wo [not(@nullFlavor)] | | | II | 0 … 1 | | | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | AD | 0 … 1 | | Adresse des Hausarztes
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | | Beliebig viele Kontaktdaten des Hausarztes.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R | | | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Name des Hausarztes.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | R |
Arztpraxis oder Ordination.
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.27 Participant Auskunftsberechtigte Person (Notfallkontakt) (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Notfallkontakt / Auskunftsberechtigte Person)
| | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.27']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND | | | In indirektem Bezug.
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.27 |  |  | hl7:time
|
| IVL_TS | 0 … 1 | |
Zeitraum, in dem der angegebene Kontakt den Notfall-Kontakt darstellt.
Wird nur angegeben, wenn der Kontakt bereits absehbar nur in einem eingeschränkten Zeitraum zur Verfügung steht.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | ECON | | | Emergency contact - Notfall-Kontakt
| | CE | 0 … 1 | | Verwandtschaftsverhältnis des Beteiligten zum Patienten, z.B. DAU („daughter“), wenn die Beteiligte die Tochter des Patienten ist. | | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | Zulässige Werte gemäß Value-Set „ELGA_PersonalRelationship“
| | st | 0 … 1 | | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.111 | | st | 1 … 1 | F | HL7:RoleCode | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.17 ELGA_PersonalRelationship (DYNAMIC) |
| | AD | 0 … 1 | | Adresse des Beteiligten
Grundsätzlich sind die Vorgaben gemäß „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Auswahl | 0 … * | | Beliebig viele Kontaktdaten des Beteiligten. Elemente in der Auswahl:- hl7:telecom[not(@nullFlavor)]
- hl7:telecom[@nullFlavor='UNK']
| | | Constraint | Es SOLL mindestens eine Telefonnummer angegeben werden. | | TEL.AT | 0 … * | R | | | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | | | | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … 1 | | Die Kontaktadresse ist unbekannt. nullFlavor "UNK" | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … 1 | | Name des Beteiligten.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | R |
Organisation, der der Beteiligte angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.25 Participant Angehoerige (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Angehöriger)
| | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.25']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND | | | In indirektem Bezug.
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.25 |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | PRS | | | Personal relationship - In persönlicher Beziehung
| | CE | 1 … 1 | M | Verwandtschaftsverhältnis des Beteiligten zum Patienten. Beispiel: DAU („daughter“), wenn die Beteiligte die Tochter des Patienten ist oder NBOR für Nachbar. | | | cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.17 ELGA_PersonalRelationship (DYNAMIC) |
| | st | 0 … 1 | | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.111 | | st | 1 … 1 | F | HL7:RoleCode | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.17 ELGA_PersonalRelationship (DYNAMIC) |
| | AD | 0 … 1 | | Adresse des Beteiligten
Grundsätzlich sind die Vorgaben gemäß „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | | Beliebig viele Kontaktdaten des Beteiligten.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R | | | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Name des Beteiligten.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | R | Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.26 Participant Versicherung (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Versicherter/Versicherung). | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.26']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | HLD |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.26 |  |  | hl7:time
|
| IVL_TS | 0 … 1 | |
Gültigkeitszeitraum der Versicherungspolizze.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | POLHOLD | | | Policy holder - Halter einer Versicherungspolizze
| | Auswahl | 1 … 1 | |
Sozialversicherungsnummer des Patienten (SELF) oder der Person, bei der der Patient mitversichert ist (FAMDEP)
Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | | Constraint |
Zugelassene nullFlavor:
- NI … Patient hat keine Sozialversicherungsnummer (z.B. Ausländer, …)
- UNK … Patient hat eine Sozialversicherungsnummer, diese ist jedoch unbekannt
| | II | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | II | 0 … 1 | | | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | CE | 1 … 1 | M |
Versicherungsverhältnis codiert
Beispiele:
- SELF, wenn der Patient selbst der Versicherte ist.
- FAMDEP, wenn der Patient bei einem Familienmitglied mitversichert ist.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.111 | | st | 1 … 1 | F | HL7:RoleCode | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.9 ELGA_InsuredAssocEntity (DYNAMIC) |
| | AD | 0 … 1 | | Adresse des Beteiligten.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | | Beliebig viele Kontaktdaten des Beteiligten.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | | 0 … 1 | C | Name des Beteiligten.
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | | Constraint | Wenn das Versicherungsverhältnis "familienversichert" ("FAMDEP“) ist, MUSS eine associatedPerson angegeben sein, M [1..1], sonst kann sie komplett entfallen, O [0..1] | | | 1 … 1 | M |
Versicherungsgesellschaft.
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | | Schematron assert | role | error | | | | test | not(hl7:code[@code='FAMDEP']) or hl7:associatedPerson | | | | Meldung | Wenn das Versicherungsverhältnis "familienversichert" ist, dann muss eine associatedPerson angegeben sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.29 Participant Betreuungsorganisation (DYNAMIC) |  | hl7:participant
|
| | 0 … 1 | | Beteiligter (Betreuende Organisation) | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.29']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.29 |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | CAREGIVER | | | Betreuer
| | | 1 … 1 | M | Betreuende Organisation | | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.28 Participant Weitere Behandler (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Weitere Behandler) | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.28']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | CON |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.28 |  |  | hl7:functionCode
|
| CE (extensible) | 0 … 1 | | Funktionscode des Behandlers z.B: „Facharzt für Neurologie“
Eigene Codes und Bezeichnungen dürfen verwendet werden. | | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code sollte gewählt werden aus dem Value Set 1.2.40.0.34.10.6 ELGA_AuthorSpeciality (DYNAMIC) |
|  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | PROV | | | Gesundheitsdiensteanbieter.
| | AD | 0 … 1 | |
Adresse des Beteiligten.
Grundsätzlich sind die Vorgaben gemäß „Adress-Elemente“ zu befolgen
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | |
Beliebig viele Kontaktdaten des Beteiligten.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.)
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …)
Bsp: WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
Bei Angabe mehrerer Telefonnummern ist jeweils das Attribut @use anzugeben.
| | | 1 … 1 | M |
Beteiligte Person
Grundsätzlich sind die Vorgaben für „Personen-Element“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | | 0 … 1 | R |
Organisation, der der Beteiligte angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 1 … * | M | von 1.2.40.0.34.6.0.11.1.9 In Fulfillment Of (DYNAMIC) | | | Constraint | Da die Referenz auf einen Auftrag im Labor eine wesentliche Information darstellt, ist dieses Element VERPFLICHTEND anzugeben. |  | hl7:inFulfillmentOf
|
| | 1 … * | M | Komponente zur Dokumentation des Auftrags.
| | | |  | | at-cda-bbr-dataelement-42 |  Auftrag Auftrag |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 1 … 1 | F | FLFS |  |  | hl7:order
|
| | 1 … 1 | M | Auftrag.
| | | cs | 1 … 1 | F | ACT | | cs | 1 … 1 | F | RQO | | II | 1 … 1 | M | Auftragsnummer, Anforderungsnummer.
Grundsätzlich sind die Vorgaben gemäß Kapitel „Identifikations-Elemente“ zu befolgen.
| | | |  | | at-cda-bbr-dataelement-43 |  ID ID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | 1 … * | M | von 1.2.40.0.34.6.0.11.1.48 Documentation Of Service Event - Labor und Mikrobiologie (DYNAMIC) |  | hl7:documentationOf
|
| | 1 … * | M | Komponente für die Gesundheitsdienstleistung.
| |  |  | @typeCode
|
| cs | 0 … 1 | F | DOC |  |  | hl7:serviceEvent
|
| | 1 … 1 | M | Die serviceEvents in den ELGA Labor- und Mikrobiologiebefunden MÜSSEN die "section/code"-Elemente als auch die "section/templateId"-Elemente wiedergeben. Diese Informationen werden über eine Mapping-Vorschrift in die XDS-Metadaten übernommen und ermöglichen einem ELGA-Teilnehmer zu erkennen, welche Sections beinhaltet sind und in welchem Codierungsgrad diese vorliegen. Daher muss für jede Section, welche medizinische Information enthält (Ausnahmen sind "Brieftext" und "Abschließende Bemerkungen"), ein documentationOf/serviceEvent codiert werden.
| | | cs | 0 … 1 | F | ACT | | cs | 0 … 1 | F | EVN | | II | 0 … 1 | C | | | | st | 0 | NP | NP/nicht anwesend | | uid | 1 … 1 | R | In das serviceEvent/id[@root] MUSS die section/templateId[@root] geschrieben werden. Im Fall von mehreren "section/templateId"-Elementen MUSS jenes gewählt werden, dessen @id-Attribut in dem OID-Bereich 1.2.40.0.34.6.0.11.2.X zu finden ist. | | | Constraint | Grundsätzlich MUSS serviceEvent/id angegeben werden. Die serviceEvent/id IST NICHT ERLAUBT für das zusätzliche serviceEvent mit dem Code "18725-2 - Microbiology studies (set)" für Mikrobiologiebefunde bzw. Laborbefunde, die mikrobiologische Ergebnisse beinhalten. | | | Schematron assert | role | error | | | | test | hl7:code[@code='18725-2'][@codeSystem='2.16.840.1.113883.6.1'] or hl7:id | | | | Meldung | serviceEvent/id MUSS angegeben werden | | | | Schematron assert | role | error | | | | test | not(hl7:code[@code='18725-2'][@codeSystem='2.16.840.1.113883.6.1'] and hl7:id) | | | | Meldung | serviceEvent/id DARF NICHT angegeben werden | | | CE | 1 … 1 | M | Code der Gesundheitsdienstleistung.
↔ Hinweis zum XDS-Mapping:
Dieses Element wird in das XDS-Attribut "eventCodeList" gemappt.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.22 ELGA_ServiceEventsLabor (DYNAMIC) |
| | IVL_TS | 1 … 1 | M | Angabe des zeitlichen Erbringungsintervalls effectiveTime mit einer Start- "low" und Endzeit "high" (verpflichtend).
Startzeitpunkt: Datum und Zeitpunkt, an dem das analysierende Labor die Anforderung vom Zuweiser in der Labor EDV erfasst hat. Falls nicht vorhanden, sind Datum und Uhrzeit des Starts des Auftrags in der Labor EDV anzugeben.
Endzeit: Datum und Zeitpunkt des Abschlusses des Auftrags, welche in der Regel mit der medizinischen Freigabe des Auftrags ident ist.
↔ Hinweis zum XDS-Mapping:
Dieses Element wird in die XDS-Attribute "serviceStartTime" und "serviceStopTime" gemappt.
Für die automatisierte Datenübernahme aus dem CDA-Dokument in die XDS-Dokumentmetadaten ist stets ein Zeitintervall anzugeben.
ACHTUNG: Die Zeitangaben der jeweils ersten Gesundheitsdienstleistung (erstes "documentationOf/serviceEvent"-Element) werden in die Dokument-Metadaten übernommen!
Die Bedeutung der Dokument-Metadaten-Elemente lautet daher wie folgt:
- serviceStartTime: Beginn des ersten documentationOf/serviceEvent-Elements
- serviceStopTime: Ende des ersten documentationOf/serviceEvent-Elements
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | | | | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. | | Eingefügt | 0 … 1 | C | von 1.2.40.0.34.6.0.11.1.14 Document Replacement - Related Document (DYNAMIC) | | | Constraint | Wird ein Befund aktualisiert, weil z.B. zuvor noch Analyseergebnisse ausständig waren, MUSS dieses Element angegeben werden. |  | hl7:relatedDocument
|
| | 0 … 1 | C | | | | |  | | at-cda-bbr-dataelement-15 |  Bezug zu vorgehenden Dokumenten Bezug zu vorgehenden Dokumenten |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 1 … 1 | R |
Art des Bezugs zum Vordokument.
| | | Constraint |
Erlaubte @typeCodes:
RPLC - replaces: Das Dokument ersetzt ein existierendes Dokument. Der Status des zu ersetzenden Dokumentes wird auf "deprecated" gesetzt, das ursprüngliche Dokument bleibt aber noch im System als historische Referenz verfügbar.
APND - append: Zusammenhängen von Dokumenten. Dies ist in ELGA bereits über das Einbetten von Dokumenten realisiert.
XFRM - transformed: Das Dokument ist Ergebnis eines Transformationsprozesses, d.h. ist aus einem anderen Originaldokument hervorgegangen.
Hinweis: Die parallele Ablage von CDA-Dokumenten, welche vom Dokumentersteller bereits mit einem Stylesheet zu einem PDF Dokument gerendert wurden, kann mit der XFRM – Transaktion vorgenommen werden. Es ist nicht auszuschließen, dass die Transformation in lokalen Affinity Domains Anwendung findet. Für ELGA ist die Transformation jedoch kein Anwendungsfall.
|  |  | hl7:parentDocument
|
| | 1 … 1 | M | Vorhergehendes Dokument.
| | | cs | 0 … 1 | F | DOCCLIN | | cs | 0 … 1 | F | EVN | | II | 1 … 1 | M | Dokumenten-Id des vorgehenden Dokuments.
Grundsätzlich sind die Vorgaben für „Identifikations-Elemente“ zu befolgen.
| | | | Schematron assert | role | error | | | | test | not(hl7:relatedDocument) or hl7:relatedDocument[@typeCode='RPLC'] | | | | Meldung | Wird /ClinicalDocument/relatedDocument angegeben, MUSS relatedDocument[@typeCode='RPLC'] sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.50 Component Of - Encompassing Encounter with id (DYNAMIC) |  | hl7:componentOf
|
| | 0 … 1 | | Komponente für den Patientenkontakt.
| | | |  | | at-cda-bbr-dataelement-33 |  Patientenkontakt Patientenkontakt |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | hl7:encompassingEncounter
|
| | 1 … 1 | M | Patientenkontakt.
| | | cs | 0 … 1 | F | ENC | | cs | 0 … 1 | F | EVN | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='UNK']
| | II | 0 … 1 | | Identifikationselement zur Aufnahme der Aufenthaltszahl | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-34 |  ID ID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | st | 1 … 1 | R | Aufenthaltszahl, z.B.: Az123456
| | uid | 1 … 1 | R | OID der Liste der Aufenthaltszahlen der Organisation
| | st | 0 … 1 | | Name der Stelle, welche die ID zugewiesen hat, z.B.: "Amadeus Spital". | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | CE | 1 … 1 | M | Codierung des Patientenkontakts.
| | | |  | | at-cda-bbr-dataelement-39 |  Art des Aufenthalts Art des Aufenthalts |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | F | HL7:ActCode | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.5 ELGA_ActEncounterCode (DYNAMIC) |
| | IVL_TS | 1 … 1 | M |
Zeitraum des Patientenkontakts.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | | | |  | | at-cda-bbr-dataelement-37 |  Beginn des Patientenkontaktes Beginn des Patientenkontaktes |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Der Zeitraum des Patientenkontaktes MUSS die Vorgaben der speziellen Implementierungsleitfäden einhalten. Dabei gilt allgemein:
- Der Zeitraum besteht aus dem Zeitpunkt der administrativen Aufnahme in die Behandlung und dem Zeitpunkt der administrativen Entlassung aus der Behandlung.
- Der Entlassungszeitpunkt kann „unbekannt“ sein, wenn die administrative Entlassung noch nicht erfolgt ist. (nullFlavor UNK beim effectiveTime.high)
- Hinweis: Als Zeitpunkt der Aufnahme/Entlassung SOLL der Zeitpunkt der administrativen Aufnahme/Entlassung angegeben werden. Wenn der Zeitpunkt der administrativen Aufnahme/Entlassung nicht vorhanden ist, darf auch der Zeitpunkt der medizinischen Aufnahme/Entlassung angegeben werden.
| | | 0 … 1 | R |
Komponente für die verantwortliche Person.
| | | |  | | at-cda-bbr-dataelement-40 |  Verantwortliche Person Verantwortliche Person |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | 1 … 1 | M |
Entität der verantwortlichen Person.
Grundsätzlich sind die Vorgaben für „AssignedEntity-Element (Person + Organisation)“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.8 Encounter Location (DYNAMIC)
Die Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand, MUSS verpflichtend angegeben werden (z.B.: die entlassende Krankenanstalt mit Abteilung).
| | | 1 … 1 | M | | | | cs | 0 … 1 | F | LOC | | | 1 … 1 | M | | | | cs | 0 … 1 | F | SDLOC | | CE | 1 … 1 | M |
Der Code zur Klassifizierung des GDA repräsentiert die Art der Einrichtung, in der die Tätigkeit stattfand, die zur Erzeugung des Dokuments führte. Zum Beispiel sollten Dokumente, die während eines ambulanten Falls in einem Krankenhaus entstehen, mit dem healthcareFacilityTypeCode für „Krankenhaus“ gekennzeichnet werden.
Zulässige Werte gemäß Value-Set „ELGA_HealthcareFacilityTypeCode“ Für ELGA SOLL der Code dem Eintrag "GDA Rollenname" oder, wenn der GDA Rollenname nicht verfügbar ist, der "Aggregierten Rolle" im GDA-I entsprechen.
↔ Hinweis zum XDS-Mapping: Dieses Element wird ins XDS-Attribut XDSDocumentEntry.healthcareFacilityTypeCode gemappt.
Zu berücksichtigen sind jeweils die Attribute @code, @codeSystem und @displayName.
| | | st | 1 … 1 | R | |  |  |  |  |  | hl7:serviceProviderOrganization
|
| | 1 … 1 | M |
Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand.
Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | |  | hl7:component
|
| | 1 … 1 | M | | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| bl | 0 … 1 | F | true |  |  | hl7:structuredBody
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | DOCBODY | | cs | 0 … 1 | F | EVN | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.69 Brieftext (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.6 Überweisungsgrund - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.114 Überweisungsgrund - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.6 Überweisungsgrund - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.114 Überweisungsgrund - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.109 Anamnese - Labor und Mikrobiologie - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.111 Anamnese - Labor und Mikrobiologie - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.109 Anamnese - Labor und Mikrobiologie - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.111 Anamnese - Labor und Mikrobiologie - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.15 Angeforderte Untersuchungen - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.112 Angeforderte Untersuchungen - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.15 Angeforderte Untersuchungen - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.112 Angeforderte Untersuchungen - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | C | Beinhaltet 1.2.40.0.34.6.0.11.2.93 Probeninformation (Specimen Section) (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | Constraint | Die Dokumentation des Untersuchungsmaterials kann auf zwei Arten erfolgen: - Enthält ein Befund nur einen Befundbereich ("Laboratory Specialty Section"), so kann die Codierung innerhalb der einen Section erfolgen.
ODER - Bei Verwendung von mehreren Befundbereichen in einem Befund kann es zu Überschneidungen der
Untersuchungsmaterialien kommen (ein spezielles Untersuchungsmaterial kann in zwei Befundbereichen analysiert werden). Die CDA Level 3 Codierung eines Untersuchungsmaterials darf jedoch nur einmal im gesamten Befund erfolgen. Daher sind die Informationen zu den Untersuchungsmaterialien in einer eigenen, führenden "Probeninformation (Specimen Section)" zu
codieren.
| | | 1 … * | M | Beinhaltet 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.103 Befundbewertung (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | ELGA Labor- und Mikrobiologiebefunde können alle Kriterien erfüllen, die für Befundberichte von der Akkreditierung für medizinische Laboratorien (ISO 15189:2012) gefordert sind. Ein Anhängen des PDF-Befundes ist NICHT erforderlich!
Für die vollständige Erfüllung aller Akkreditierungsanforderungen an Befundberichte ist das erstellende Labor verantwortlich; die Akkreditierung wird von der Abt. Akkreditierung Austria im Bundesministerium für Digitalisierung und Wirtschaftsstandort durchgeführt.
Mit Rücksicht auf eine einfache Verwendbarkeit der Befunde durch die Benutzer (z.B. niedergelassene Ärzte), die häufig mit einer großen Anzahl von Laborbefunden eines Patienten konfrontiert sind, ist eine Duplizierung der Daten durch Anhängen einer PDF-Ansicht daher nicht gestattet.
Beinhaltet 1.2.40.0.34.6.0.11.2.71 Beilagen (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.70 Abschließende Bemerkung (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.33 Stylesheet Test eBefund (DYNAMIC) | | | Schematron assert | role | error | | | | test | matches(//processing-instruction('xml-stylesheet'), '[^\w]ELGA_Stylesheet_v1.0.xsl[^\w]') | | | | Meldung | (xml-processing-instr): Es muss ein xml-stylesheet-Prologattribut anwesend sein mit dem Wert für @href=ELGA_Stylesheet_v1.0.xsl | |
|
14.4.2.2 Mikrobiologiebefund
| Id | 1.2.40.0.34.6.0.11.0.14 | Gültigkeit ab | 2021‑03‑29 15:24:59 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 3.0.0+20211214 |
|---|
| Name | atlab_document_Mikrobiologiebefund | Bezeichnung | Mikrobiologiebefund |
|---|
| Beschreibung | Der Mikrobiologiebefund erlaubt es, die anfallenden Analysen entsprechend dem klassischen Untersuchungsverlauf der Mikrobiologie zu dokumentieren: - Beschreibung des entnommenen Materials (z.B. Mittelstrahlharn) inklusive makroskopischer Beurteilung
- Mikroskopische Analyse des Materials (z.B. Erythrozyten, Leukozyten, grampositive
Bakterien)
- Kultureller Erregernachweis (inkl. Antibiogramm mit Interpretation und/oder minimaler Hemmkonzentration)
- Molekularer Erregernachweis
- Infektionsserologie
|
|
| Kontext | Pfadname / |
|---|
| Klassifikation | CDA Document Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 47 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.1.10 | Inklusion |  Document Realm (1.0.1+20230717) Document Realm (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.30 | Inklusion |  Document TypeId (1.0.0+20210219) Document TypeId (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.1 | Inklusion |  Document Id (1.0.0+20210219) Document Id (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.45 | Inklusion |  Document StatusCode (1.0.1+20210624) Document StatusCode (1.0.1+20210624) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.46 | Inklusion |  Document TerminologyDate (1.0.0+20210219) Document TerminologyDate (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.44 | Inklusion |  Document PracticeSettingCode (1.1.0+20210303) Document PracticeSettingCode (1.1.0+20210303) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.11 | Inklusion |  Document Effective Time (1.0.1+20230717) Document Effective Time (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.12 | Inklusion |  Document Confidentiality Code (1.0.2+20230717) Document Confidentiality Code (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.13 | Inklusion |  Document Language (1.0.0+20210219) Document Language (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.15 | Inklusion |  Document Set Id and Version Number (1.0.0+20210219) Document Set Id and Version Number (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.3 | Inklusion |  Record Target (1.2.1) Record Target (1.2.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.2 | Inklusion |  Author (1.0.3+20230717) Author (1.0.3+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.22 | Inklusion |  Data Enterer (1.0.1+20230717) Data Enterer (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.4 | Inklusion |  Custodian (1.0.1+20211213) Custodian (1.0.1+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.24 | Inklusion |  Information Recipient (1.0.0+20210219) Information Recipient (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.5 | Inklusion |  Legal Authenticator (1.0.0+20210219) Legal Authenticator (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.49 | Inklusion |  Laboratory Results Validator (1.0.0+20211213) Laboratory Results Validator (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.42 | Inklusion |  Participant Auftraggeber / Ordering Provider (1.1.0+20211213) Participant Auftraggeber / Ordering Provider (1.1.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.20 | Inklusion |  Participant Fachlicher Ansprechpartner (1.0.2+20210803) Participant Fachlicher Ansprechpartner (1.0.2+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.23 | Inklusion |  Participant Hausarzt (1.0.1+20210803) Participant Hausarzt (1.0.1+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.27 | Inklusion |  Participant Auskunftsberechtigte Person (Notfallkontakt) (1.0.2+20210803) Participant Auskunftsberechtigte Person (Notfallkontakt) (1.0.2+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.25 | Inklusion |  Participant Angehoerige (1.0.1+20210803) Participant Angehoerige (1.0.1+20210803) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.26 | Inklusion |  Participant Versicherung (1.0.0+20210219) Participant Versicherung (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.29 | Inklusion |  Participant Betreuungsorganisation (1.0.0+20210219) Participant Betreuungsorganisation (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.28 | Inklusion |  Participant Weitere Behandler (1.0.0+20210219) Participant Weitere Behandler (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.9 | Inklusion |  In Fulfillment Of (1.0.1+20210628) In Fulfillment Of (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.48 | Inklusion |  Documentation Of Service Event - Labor und Mikrobiologie (1.0.0+20211213) Documentation Of Service Event - Labor und Mikrobiologie (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.14 | Inklusion |  Document Replacement - Related Document (1.0.1+20210628) Document Replacement - Related Document (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.50 | Inklusion |  Component Of - Encompassing Encounter with id (1.0.1+20230717) Component Of - Encompassing Encounter with id (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.69 | Containment |  Brieftext (1.0.1+20210628) Brieftext (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.6 | Containment |  Überweisungsgrund - codiert (1.0.0+20211213) Überweisungsgrund - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.114 | Containment |  Überweisungsgrund - uncodiert (1.0.0+20211213) Überweisungsgrund - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.109 | Containment |  Anamnese - Labor und Mikrobiologie - codiert (1.0.0+20211213) Anamnese - Labor und Mikrobiologie - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.111 | Containment |  Anamnese - Labor und Mikrobiologie - uncodiert (1.0.0+20211213) Anamnese - Labor und Mikrobiologie - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.15 | Containment |  Angeforderte Untersuchungen - codiert (1.0.0+20211213) Angeforderte Untersuchungen - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.112 | Containment |  Angeforderte Untersuchungen - uncodiert (1.0.0+20211213) Angeforderte Untersuchungen - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.93 | Containment |  Probeninformation (Specimen Section) (1.0.0+20211213) Probeninformation (Specimen Section) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.105 | Containment |  Laboratory Specialty Section (Mikroskopie) - codiert (1.0.0+20211213) Laboratory Specialty Section (Mikroskopie) - codiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.113 | Containment |  Laboratory Specialty Section (Mikroskopie) - uncodiert (1.0.0+20211213) Laboratory Specialty Section (Mikroskopie) - uncodiert (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.106 | Containment |  Laboratory Specialty Section (Kultureller Erregernachweis) (1.0.0+20211213) Laboratory Specialty Section (Kultureller Erregernachweis) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.107 | Containment |  Laboratory Specialty Section (Molekularer Erregernachweis) (1.0.0+20211213) Laboratory Specialty Section (Molekularer Erregernachweis) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.108 | Containment |  Laboratory Specialty Section (Infektionsserologie) (1.0.0+20211213) Laboratory Specialty Section (Infektionsserologie) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.110 | Containment |  Laboratory Specialty Section (Weitere Analysen) (1.0.0+20211213) Laboratory Specialty Section (Weitere Analysen) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.103 | Containment |  Befundbewertung (1.0.0+20211213) Befundbewertung (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.71 | Containment |  Beilagen (1.0.2+20230717) Beilagen (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.70 | Containment |  Abschließende Bemerkung (1.0.1+20210628) Abschließende Bemerkung (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.33 | Inklusion |  Stylesheet Test eBefund (1.0.1+20210628) Stylesheet Test eBefund (1.0.1+20210628) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.6.0.11.0.11 Laborbefund (2020‑08‑25 14:35:13) ref at-lab- |
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCCLIN |  | @moodCode
|
| cs | 0 … 1 | F | EVN | | | Constraint | Entsprechend den Vorgaben des IHE Frameworks für Labor sind für Personen und Organisationen die Angabe eines Namens ("name"-Element), einer Adresse ("addr"-Element) und einer Telekom-Verbindung ("telecom"-Element) verpflichtend. Diese können jedoch mit einem nullFlavor versehen werden. | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.10 Document Realm (DYNAMIC) |  | hl7:realmCode
|
| CS | 1 … 1 | M | Hoheitsbereich des Dokuments.
Fester Wert: @code = AT (aus Value Set „ELGA_RealmCode“) | |  |  | @code
|
| | 1 … 1 | F | AT | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.30 Document TypeId (DYNAMIC) |  | hl7:typeId
|
| II | 1 … 1 | M | Dokumentformat CDA R2
| |  |  | @root
|
| uid | 1 … 1 | F | 2.16.840.1.113883.1.3 |  |  | @extension
|
| st | 1 … 1 | F | POCD_HD000040 |  | hl7:templateId
|
| II | 1 … 1 | M | Fixe OID für alle Dokumente, die in der Governance-Gruppe "eHealth Austria" abgestimmt werden und von einem zentralen Art-Decor-Repository abgeleitet werden (AT-CDA-BBR). | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.0.1 |  | hl7:templateId
|
| II | 1 … 1 | M | OID des Implementierungsleitfadens "Labor- und Mikrobiologiebefund" (Dokument-OID). Dient als informative Referenz. | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.7.4.9.3 |  | hl7:templateId
|
| II | 1 … 1 | M | OID des Art-Decor-Templates für das Dokument "Mikrobiologiebefund" (Document Level Template für Schematron) | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.0.14 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.2.3 templateId
Im Grunde folgt dieser Leitfaden den Vorgaben der IHE. Die Codierung der "Laboratory Specialty Section" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3 | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.1 Document Id (DYNAMIC) |  | hl7:id
|
| II | 1 … 1 | M | Dokumenten-Id des CDA-Dokuments.
Es MUSS eine gültige und innerhalb des ID-Pools eindeutige Dokumenten-ID angegeben werden.
Grundsätzlich sind die Vorgaben gemäß „Identifikations-Elemente“ zu befolgen.
| |  |  | @root
|
| uid | 1 … 1 | R | |  | hl7:code
|
| CE | 1 … 1 | M | Für den Mikrobiologiebefund ist als Dokumententyp (/ClinicalDocument/code) "18725-2 - Microbiology studies (set)" und als Dokumentenklasse (/ClinicalDocument/code/translation) "11502-2 - Laboratory report" anzugeben.
↔ Hinweis zum XDS-Mapping:
- Das code-Element wird in das XDS-Metadaten-Attribut XDSDocumentEntry.typeCode übernommen.
- Das translation-Element wird in das XDS-Metadaten-Attribut XDSDocumentEntry.classCode übernommen.
| |  |  | @codeSystemName
|
| st | 0 … 1 | F | LOINC |  |  | @code
|
| CONF | 1 … 1 | F | 18725-2 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.1 (LOINC) |  |  | @displayName
|
| 1 … 1 | F | Microbiology studies (set) |  |  | hl7:translation
|
| CD | 1 … 1 | M | Fixe Dokumentenklasse "11502-2 - Laboratory report" | | | st | 0 … 1 | F | LOINC | | CONF | 1 … 1 | F | 11502-2 | | 1 … 1 | F | 2.16.840.1.113883.6.1 (LOINC) | | 1 … 1 | F | Laboratory report |  | hl7:title
|
| ST | 1 … 1 | M | Der Titel des CDA Dokuments für den lesenden Empfänger. MUSS die Art des Dokuments (Dokumenttyp) widerspiegeln.
Zum Beispiel "Mikrobiologiebefund".
| | | Eingefügt | 0 … 1 | C | von 1.2.40.0.34.6.0.11.1.45 Document StatusCode (DYNAMIC) | | | Constraint | Labor- und Mikrobiologiebefunde sind grundsätzlich abgeschlossene bzw. "fertige" Dokumente - in diesen Fällen erübrigt sich die Angabe eines Status.
Befunde, in denen die Ergebnisse bestimmter Analysen noch ausständig sind ("Wert folgt"), MÜSSEN den Dokumentenstatus "active" erhalten und das Ergebnis der ausständigen Analyse MUSS den SNOMED CT Code "255599008 - Incomplete (qualifier value)" erhalten. |  | sdtc:statusCode
|
| CS | 0 … 1 | C |
Status eines Dokuments.
e-Befunde sind grundsätzlich abgeschlossene bzw. "fertige" ("completed") Dokumente, daher entfällt die Angabe eines Status. In folgenden Ausnahmen SOLL der Status eines Dokuments wie folgt angegeben werden:
- “active”: z.B. wenn bekannt ist, dass Updates folgen werden: Etwa für "vorläufige ärztliche Entlassungsbriefe" oder Laborbefunde, für die noch Ergebnisse einzelner Analysen ausständig sind
- “nullified”: z.B. für Dokumente, die gemäß Anwendungsfall "Storno von ELGA-Dokumenten" storniert werden, wobei zusätzlich ein letztes Dokument mit Storniert-Status in der Versionskette registriert wird.
↔ Hinweis zum XDS-Mapping: Der Status wird nicht in die XDS-Metadaten übernommen!
| | | | Constraint |
Zulässige Werte für sdtc:statusCode/@code sind "active" und "nullified"
| | | CONF | | @code muss "nullified" sein | | oder | | @code muss "active" sein |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.46 Document TerminologyDate (DYNAMIC) |  | hl7at:terminologyDate
|
| TS.DATE.FULL | 1 … 1 | M | Das Terminologie-Datum des Dokumentes
Das Datum, an dem die lokal zur Implementierung verwendeten Value Sets mit dem österreichischen Terminologieserver abgeglichen wurden, wird hier angegeben.
| | | | Constraint | Das Datum der letzten Terminologie-Aktualisierung MUSS entsprechend klassischer HL7 V3 Notation im Format "YYYYMMDD" angegeben werden.
Beispiel: 20200527
|  | hl7at:formatCode
|
| CD | 1 … 1 | M | ↔ Hinweis zum XDS-Mapping:
@code wird in das XDS-Attribut XDSDocumentEntry.formatCode übernommen. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | ELGA_FormatCode |  |  | @code
|
| CONF | 1 … 1 | F | urn:hl7-at:lab:3.0.0+20211214 |  |  | @codeSystem
|
| 1 … 1 | F | 1.2.40.0.34.5.37 |  |  | @displayName
|
| 1 … 1 | F | HL7 Austria Labor- und Mikrobiologiebefund 3.0.0+20211214 | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.44 Document PracticeSettingCode (DYNAMIC) |  | hl7at:practiceSettingCode
|
| CD | 1 … 1 | M | Die fachliche Zuordnung des Dokumentes | |  |  | @displayName
|
| | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.75 ELGA_PracticeSetting (DYNAMIC) |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.11 Document Effective Time (DYNAMIC)
Angabe des medizinisch zutreffendsten Datums - in der Regel das Abnahmedatum/-zeit des Untersuchungsmaterials. Sollte dieses nicht vorliegen, kann auf andere möglichst passende Zeitpunkte zurückgegriffen werden: Auftragszeitpunkt, Laboreingangszeitpunkt, Vidierungszeitpunkt, etc. |  | hl7:effectiveTime
|
| TS.AT.TZ | 1 … 1 | M |
Relevantes Datum des Dokuments.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
| | | |  | | at-cda-bbr-dataelement-11 |  Erstellungsdatum Erstellungsdatum |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.12 Document Confidentiality Code (DYNAMIC) |  | hl7:confidentialityCode
|
| CE | 1 … 1 | M | Vertraulichkeitscode des Dokuments aus Value Set „ELGA_Confidentiality“. | | | |  | | at-cda-bbr-dataelement-13 |  Vertraulichkeitscode Vertraulichkeitscode |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @codeSystemName
|
| st | 1 … 1 | F | HL7:Confidentiality | | | Constraint | Für ELGA-Dokumente ist ausschließlich "N" erlaubt!
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.13 Document Language (DYNAMIC) |  | hl7:languageCode
|
| CS.LANG | 1 … 1 | M | Sprachcode des Dokuments.
| | | |  | | at-cda-bbr-dataelement-14 |  Sprachcode Sprachcode |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @code
|
| cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.10 ELGA_LanguageCode (DYNAMIC) |
| | | Constraint | Für ELGA ist in @code für CDA und Ableitungen in die XDSDocumentEntry-Metadaten derzeit ausschließlich der Wert "de-AT" zulässig.
Für eHealth und zukünftige Versionen der ELGA Leitfäden können weitere Sprachcodes erlaubt werden. | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.15 Document Set Id and Version Number (DYNAMIC) |  | hl7:setId
|
| II | 1 … 1 | M |
Eindeutige Id des Dokumentensets. Diese bleibt über alle Versionen der Dokumente gleich (initialer Wert bleibt erhalten). Die setId SOLL unterschiedlich zur clinicalDocument.id sein. ↔ Hinweis zum XDS-Mapping: Dieses Element wird ins XDS-Attribut referenceIdList
("urn:elga:iti:xds:2014:ownDocument_setId") gemappt. Hinweis: Bestimmte Systeme, die bei der Übernahme der setId in die XDS-Metadaten mit dem V2-Datentyp CX arbeiten, könnten ein Problem mit @extension-Attributen haben, die länger als 15 Zeichen sind. | |  | hl7:versionNumber
|
| INT.NONNEG | 1 … 1 | M | Versionsnummer des Dokuments, wird bei neuen Dokumenten mit 1 festgelegt.
Die versionNumber ist eine natürliche Zahl für die fortlaufende Versionszählung. Mit einer neuen Version wird diese Zahl hochgezählt, während die setId gleich bleibt.
| |  |  | @value
|
| int | 1 … 1 | R | Versionsnummer als positive ganze Zahl.
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.3 Record Target (DYNAMIC) |  | hl7:recordTarget
|
| | 1 … 1 | M | Komponente für die Patientendaten. | | | |  | | at-cda-bbr-dataelement-64 |  Patient Patient |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | RCT |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:patientRole
|
| | 1 … 1 | M | Patientendaten.
| | | cs | 0 … 1 | F | PAT | | II | 2 … * | R | Patientenidentifikatoren | | | |  | | at-cda-bbr-dataelement-193 |  EKVK EKVK |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-65 |  LokaleID LokaleID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-66 |  SVNr SVNr |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-67 |  bPK-GH bPK-GH |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Hinweis: Die Reihenfolge der id-Elemente MUSS unbedingt eingehalten werden!
* id[1] Identifikation des Patienten im lokalen System (1..1 M)
↔ Hinweis zum XDS-Mapping: Das Element id[1] wird ins XDS-Attribut sourcePatientId gemappt.
* id[2] Sozialversicherungsnummer des Patienten (1..1 R):
- @root: OID der Liste aller österreichischen Sozialversicherungen, fester Wert: 1.2.40.0.10.1.4.3.1 (1..1 M)
- @extension: Vollständige Sozialversicherungsnummer des Patienten (10 Stellen) (1..1 M)
- @assigningAuthorityName: Fester Wert: Österreichische Sozialversicherung (0..1 O)
Zugelassene nullFlavor:
- NI … Patient hat keine Sozialversicherungsnummer (z.B. Ausländer)
- UNK … Patient hat eine Sozialversicherungsnummer, diese ist jedoch unbekannt
* id[@root="1.2.40.0.10.2.1.1.149"] Bereichsspezifisches Personenkennzeichen (0..1 O):
- @root : OID der österreichischen bPK, fester Wert: 1.2.40.0.10.2.1.1.149 (1..1 M)
- @extension : bPK des Patienten: concat(Bereichskürzel, ":", bPK) (Base64,28 Zeichen). Typischerweise bPK-GH (Gesundheit). Kann im Zusammenhang mit E-ID auch andere Bereichskürzel tragen.
Anmerkung : Das bPK dient ausschließlich technisch der Zuordnung der elektronischen Identität und darf daher weder angezeigt werden noch am Ausdruck erscheinen noch in allfälligen Downloads enthalten sein (1..1 M)
- @assigningAuthorityName : Fester Wert: Österreichische Stammzahlenregisterbehörde (0..1 O)
* id[@root="1.2.40.0.34.4.21"] Europäische Krankenversicherungskarte kurz (0..1 O):
- @root: OID der EKVK, fester Wert: 1.2.40.0.34.4.21 (1..1 M) - @extension: Datenfeld 3 der EKVK - @assigningAuthorityName : Fester Wert: Nationaler Krankenversicherungsträger (0..1 O) * id[@root="1.2.40.0.10.1.4.3.8"] Europäische Krankenversicherungskarte lang (0..1 O):
- @root: OID der EKVK, fester Wert: 1.2.40.0.34.4.21 (1..1 M) - @extension: Datenfelder der EKVK nach folgender Bildungsvorschrift: concat(Feld 6,"^",Feld 7,"^",Feld 8,"^",Feld 9) wobei Feld 6 "Persönliche Kennnummer" angegeben sein MUSS (1..1 M). Die übrigen Datenfelder sind optional (0..1 O). In Feld 9 MUSS die Datumsangabe im Format YYYMMDD erfolgen.
- @assigningAuthorityName : Fester Wert: Nationaler Krankenversicherungsträger (0..1 O)
Grundsätzlich sind die Vorgaben gemäß „Identifikations-Elemente“ zu befolgen. | | | Beispiel | EKVK Beispiel-Max <id root="1.2.40.0.34.4.21" extension="123456789^1100-OEGK^800400010016^20251231"/> | | | Beispiel | EKVK Beispiel-Min <id root="1.2.40.0.34.4.21" extension="123456789"/> | | | 0 … 2 | R | Adresse des Patienten.
Es MUSS eine mögliche Adresse unterstützt werden. Spezielle Leitfäden (z.B. Entlassungsbrief Pflege) können es erforderlich machen, dass mehr als eine Adresse unterstützt werden muss.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | |  | | at-cda-bbr-dataelement-68 |  Adresse Adresse |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Werden mehrere gleichartige address-Elemente strukturiert (z.B. Home, Pflege), MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … * | R | Kontakt-Element. Grundsätzlich sind die Vorgaben gemäß „Kontaktdaten-Element“ zu befolgen.
| | | |  | | at-cda-bbr-dataelement-72 |  Kontaktdaten Kontaktdaten |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | url | 1 … 1 | R |
Formatkonvention siehe „telecom-Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß Value-Set „ELGA_URLScheme“
| | cs | 0 … 1 | | Bedeutung des angegebenen Kontakts (z.B Heim, Arbeitsplatz), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | | 1 … 1 | M | Name des Patienten.
Für den Namen ist verpflichtend Granularitätsstufe 2 („strukturierte Angabe des Namens‘‘) anzuwenden!
Grundsätzlich sind die Vorgaben gemäß „Namen-Elemente von Personen PN“ zu befolgen. | | | |  | | at-cda-bbr-dataelement-70 |  Name Name |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | cs | 0 … 1 | F | PSN | | cs | 0 … 1 | F | INSTANCE | | PN | 1 … 1 | M | Namen-Element (Person) | | | cs | 0 … 1 | |
Die genaue Bedeutung des angegebenen Namens, z.B. Angabe eines Künstlernamens mit „A" für „Artist“.
Zulässige Werte gemäß Value Set „ELGA_EntityNameUse“.
Wird kein @use Attribut angegeben, gilt der Name als rechtlicher Name („L“).
| | ENXP | 0 … * | |
Beliebig viele Präfixe zum Namen, z.B. Akademische Titel
Achtung: Die Angabe der Anrede („Frau“, „Herr“), ist im CDA nicht vorgesehen!
| | | cs | 0 … 1 | |
Bedeutung eines prefix-Elements, z.B. Angabe eines akademischen mit "AC" für „Academic“.
Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier".
| | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | ENXP | 1 … * | M | Mindestens ein Hauptname (Nachname). | | | cs | 0 … 1 | | Bedeutung eines family-Elements, z.B. Angabe eines Geburtsnamen mit „BR" für „Birth“. Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier“. | | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | ENXP | 1 … * | M | Mindestens ein Vorname | | | cs | 0 … 1 | |
Die genaue Bedeutung eines given-Elements, beispielsweise dass das angegebene Element einen Geburtsnamen bezeichnet, z.B. BR („Birth“).
Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier“
| | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | ENXP | 0 … * | | Beliebig viele Suffixe zum Namen | | | cs | 0 … 1 | | Die genaue Bedeutung eines suffix-Elements, beispielsweise dass das angegebene Suffix einen akademischen Titel darstellt, z.B.: AC („Academic“).
Zulässige Werte gemäß Value Set „ELGA_EntityNamePartQualifier“. | | | CONF | | Der Wert von @qualifier muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.8 ELGA_EntityNamePartQualifier (DYNAMIC) |
| | Auswahl | 1 … 1 | | Das "administrative Geschlecht" ist das soziale oder gesellschaftliche Geschlecht ("Gender"). Das administrative Geschlecht ist daher grundsätzlich getrennt von den biologischen Merkmalen der Person zu sehen. Grundsätzlich soll das administrative Geschlecht dem im
Zentralen Melderegister (ZMR) eingetragenen Geschlecht entsprechen. Über ein Translation-Element können weitere Angaben zum Geschlecht gemacht werden, wenn diese abweichend vom administrativen Geschlecht sind, z.B.: - Biologisches Geschlecht
- Geschlecht in der
Sozialversicherung
- Geschlecht für die Stations-/Bettenbelegung im Krankenhaus
Codierung des Geschlechts des Patienten aus ValueSet "ELGA_AdministrativeGender". - hl7:administrativeGenderCode[not(@nullFlavor)]
- hl7:administrativeGenderCode[@nullFlavor='UNK']
|  |  |  |  |  | hl7:administrativeGenderCode
|
| CE | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-74 |  Geschlecht Geschlecht |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | st | 1 … 1 | R | | | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.1 | | st | 0 … 1 | F | HL7:AdministrativeGender | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.4 ELGA_AdministrativeGender (DYNAMIC) |
| | CD | 0 … * | R | Über ein Translation-Element können weitere Angaben zum Geschlecht gemacht werden, wenn diese abweichend vom administrativen Geschlecht sind, z.B.: Biologisches Geschlecht, Geschlecht in der Sozialversicherung, Geschlecht für die Stations-/Bettenbelegung im Krankenhaus | | | st | 1 … 1 | R | | | | Beispiel | Beispiel für eine SNOMED CT Angabe <translation code="772004004" codeSystem="2.16.840.1.113883.6.96" displayName="Non-binary gender"/> |  |  |  |  |  | hl7:administrativeGenderCode
|
| CE | 0 … 1 | | Mittels nullFlavor="UNK" wird "Unbekannt" abgebildet. Dies schließt die Ausprägung "Keine Angabe" mit ein. | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … 1 | | Geburtsdatum des Patienten.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
- hl7:birthTime
- hl7:birthTime[@nullFlavor='UNK']
| | TS.AT.VAR | 0 … 1 | | | | | |  | | at-cda-bbr-dataelement-75 |  Geburtsdatum Geburtsdatum |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | TS.AT.VAR | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | BL | 0 … 1 | R | Kennzeichen, dass die Person verstorben ist. Kann alternativ zum Todesdatum angegeben werden, v.a. wenn der Todeszeitpunkt nicht bekannt ist. | | | |  | | at-cda-bbr-dataelement-192 |  Verstorben-Kennzeichen Verstorben-Kennzeichen |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | TS.AT.TZ | 0 … 1 | R | Todesdatum der Person. | | | |  | | at-cda-bbr-dataelement-191 |  Todesdatum Todesdatum |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | CE | 0 … 1 | R | Codierung des Familienstands des Patienten.
Zulässige Werte gemäß Value-Set „ELGA_MaritalStatus“ | | | |  | | at-cda-bbr-dataelement-98 |  Familienstand Familienstand |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.2 | | st | 1 … 1 | F | HL7:MaritalStatus | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.11 ELGA_MaritalStatus (DYNAMIC) |
|  |  |  |  | hl7:religiousAffiliationCode
|
| CE | 0 … 1 | R | Codierung des Religionsbekenntnisses des Patienten.
Zulässige Werte gemäß Value-Set „ELGA_ReligiousAffiliation“ | | | |  | | at-cda-bbr-dataelement-99 |  Religionsbekenntnis Religionsbekenntnis |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.2.16.1.4.1 | | st | 1 … 1 | F | HL7.AT:ReligionAustria | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.18 ELGA_ReligiousAffiliation (DYNAMIC) |
| | | | NP | Rasse des Patienten.
Darf nicht verwendet werden! | | | | | NP | Ethnische Zugehörigkeit des Patienten.
Darf nicht verwendet werden! | | | | 0 … * | R | Gesetzlicher Vertreter:
- Vorsorgebevollmächtigte/r (Bevollmächtigte/r durch Vorsorgevollmacht)
- Gewählte/r ErwachsenenvertreterIn
- Gesetzliche/r ErwachsenenvertreterIn
- Gerichtliche/r ErwachsenenvertreterIn (Sachwalter)
Der gesetzliche Vetreter kann entweder eine Person (guardianPerson) oder eine Organisation (guardianOrganization) sein.
Beim Patienten können optional ein oder mehrere gesetzliche Vetreter angegeben werden. Wenn ein gesetzliche Vetreter bekannt ist, SOLL diese Information auch angegeben werden.
| | | |  | | at-cda-bbr-dataelement-88 |  Gesetzlicher Vertreter Gesetzlicher Vertreter |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 0 … 1 | F | GUARD | | | 0 … 1 | R | Die Adresse des gesetzlichen Vertreters oder der Organisation.
Grundsätzlich sind die Vorgaben für „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | TEL.AT | 0 … * | R | Beliebig viele Kontaktdaten des gesetzlichen Vertreters als Person oder Organisation.
Grundsätzlich sind die Vorgaben gemäß „Kontaktdaten-Element“ zu befolgen.
| | | st | 1 … 1 | R |
Formatkonvention siehe „telecom-Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß Value-Set „ELGA_URLScheme“
| | set_cs | 0 … 1 | | Bedeutung des angegebenen Kontakts (z.B. Heim, Arbeitsplatz) Bsp: WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Angabe des gesetzlichen Vertreters als Person (guardianPerson in Granularitätsstufe 1 oder 2) ODER als Organisation (guardianOrganization)
Elemente in der Auswahl:- hl7:guardianPerson welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:guardianPerson welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
- hl7:guardianOrganization welches enthält Template 1.2.40.0.34.6.0.11.9.27 Organization Name Compilation (DYNAMIC)
| | | 0 … 1 | | Name des gesetzlichen Vertreters: Angabe in Granularitätsstufe 1
Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | | 0 … 1 | | Name des gesetzlichen Vertreters: Angabe in Granularitätsstufe 2
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | |  |  |  |  |  |  | hl7:guardianOrganization
|
| | 0 … 1 | R | Name des gesetzlichen Vertreters (Organisation)
Beinhaltet 1.2.40.0.34.6.0.11.9.27 Organization Name Compilation (DYNAMIC) | | | | 0 … 1 | R | Geburtsort des Patienten. | | | |  | | at-cda-bbr-dataelement-76 |  Geburtsort Geburtsort |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 0 … 1 | F | BIRTHPL | | | 1 … 1 | M | | | | cs | 0 … 1 | F | PLC | | cs | 0 … 1 | F | INSTANCE | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:addr welches enthält Template 1.2.40.0.34.6.0.11.9.10 Address Compilation Minimal (DYNAMIC)
- hl7:addr welches enthält Template 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC)
| | AD | 0 … 1 | | Die Adresse des Geburtsorts. Minimalangabe. Alle Elemente optional.
Beinhaltet 1.2.40.0.34.6.0.11.9.10 Address Compilation Minimal (DYNAMIC) | | | AD | 0 … 1 | | Die Adresse des Geburtsorts, struktuiert.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | |  |  |  |  | hl7:languageCommunication
|
| | 0 … * | R | Informationen bezüglich der Sprachfähigkeiten und Ausdrucksform des Patienten.
| | | |  | | at-cda-bbr-dataelement-100 |  Sprachfähigkeit Sprachfähigkeit |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | CS | 1 … 1 | M | Sprache, die vom Patienten zu einem bestimmten Grad beherrscht wird (geschrieben oder gesprochen).
In der Klasse languageCommunication können Informationen bezüglich der Sprachfähigkeiten und Ausdrucksform (z.B. gesprochen oder geschrieben) des Patienten angegeben werden.
Dieser Leitfaden schränkt die möglichen Werte für die Sprache auf Werte aus dem Value Set ELGA_HumanLanguage ein.
Die Gebärdensprache ist als eigene Sprache inkl. Ländercode anzugeben, mit der Ergänzung des Länder-/Regional-Codes (z.B. sgn-at), die Ausdrucksweise (MoodCode) wird in diesem Fall nicht angegeben (denn expressed / received signed wären redundant). | | | |  | | at-cda-bbr-dataelement-101 |  Sprache Sprache |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | Zulässige Werte gemäß Value-Set „ELGA_HumanLanguage“ aus Code-System „HL7:HumanLanguage 2.16.840.1.113883.6.121“
Gemäß IETF / RFC 3066 enthält es ein bestimmtes Subset von Codes aus ISO 639-1 und ISO 639-2 (also zwei- und dreistellige Sprachcodes). Gemäß RFC 3066 ist es zulässig, eine Angabe der landestypischen Ausprägung der Sprache nach einem Bindestrich anzufügen. Das Land wird dabei nach ISO 3166-1 Alpha 2 angegeben. Dies MUSS bei der Auswertung des languageCodes berücksichtigt und toleriert werden.
| | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.173 ELGA_HumanLanguage (DYNAMIC) |
| | CE | 0 … 1 | C | Ausdrucksform der Sprache.
Zulässige Werte gemäß Value-Set „ELGA_LanguageAbilityMode“
| | | cs | 1 … 1 | R | | | st | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.60 | | st | 0 … 1 | F | HL7:LanguageAbilityMode | | | Constraint | Bei Strukturierung einer Gebärdensprache ist dieses Element NICHT ERLAUBT, NP [0..0] und MUSS daher komplett entfallen | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.175 ELGA_LanguageAbilityMode (DYNAMIC) |
|  |  |  |  |  | hl7:proficiencyLevelCode
|
| CE | 0 … 1 | R | Grad der Sprachkenntnis in der Sprache.
Zulässige Werte gemäß Value-Set „ELGA_ProficiencyLevelCode“
| | | |  | | at-cda-bbr-dataelement-102 |  Grad der Sprachkenntnis Grad der Sprachkenntnis |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | st | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.61 | | st | 0 … 1 | F | HL7:LanguageAbilityProficiency | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.174 ELGA_ProficiencyLevelCode (DYNAMIC) |
| | BL | 0 … 1 | R | Kennzeichnung, ob die Sprache in der angegebenen Ausdrucksform vom Patienten bevorzugt wird. | | | |  | | at-cda-bbr-dataelement-103 |  Sprachpräferenz Sprachpräferenz |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Schematron assert | role | error | | | | test | not(hl7:id[1]/@nullFlavor) | | | | Meldung | Die Verwendung von id/@nullFlavor ist an dieser Stelle NICHT ERLAUBT. | | | | Schematron assert | role | error | | | | test | not(hl7:id[2]/@nullFlavor) or (hl7:id[2][@nullFlavor='UNK'] or hl7:id[2][@nullFlavor='NI']) | | | | Meldung | Zugelassene nullFlavor sind "NI" und "UNK" | | | Eingefügt | 1 … * | M | von 1.2.40.0.34.6.0.11.1.2 Author (DYNAMIC) |  | hl7:author
|
| | 1 … * | M | Verfasser des Dokuments.
| |  |  | @typeCode
|
| cs | 0 … 1 | F | AUT |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:functionCode
|
| CE (extensible) | 0 … 1 | R |
Funktionscode des Verfassers des Dokuments, z.B: „Diensthabender Oberarzt“, „Verantwortlicher Arzt für Dokumentation“,„Stationsschwester“.
Eigene Codes und Bezeichnungen können verwendet werden.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | Auswahl | 1 … 1 | |
Der Zeitpunkt, zu dem das Dokument verfasst bzw. inhaltlich fertiggestellt wurde.
Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='UNK']
| | TS.AT.TZ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | TS.AT.TZ | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK |  |  | hl7:assignedAuthor
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ASSIGNED | | Auswahl | 1 … * | |
Identifikation des Verfassers des Dokuments im lokalen System des/der datenerstellenden Gerätes/Software.
ODER Identifikation des/der datenerstellenden Gerätes/Software.
Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | | Constraint | Zugelassene nullFlavor:
- NI ….... Person hat keine ID / Gerät/Software hat keine ID
- UNK … Person hat eine ID, diese ist jedoch unbekannt / Gerät/Software hat eine ID, diese ist jedoch unbekannt
| | II | 0 … * | |
Identifikation des Verfassers des Dokuments im lokalen System des/der datenerstellenden Gerätes/Software.
ODER Identifikation des/der datenerstellenden Gerätes/Software.
| | | wo [not(@nullFlavor)] | | | II | 0 … 1 | | | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | CE | 0 … 1 | R |
Angabe der Fachrichtung des Verfassers des Dokuments („Sonderfach“ gem. Ausbildungsordnung), z.B: „Facharzt/Fachärztin für Gynäkologie“.
Wenn ein Autor mehreren ärztlichen Sonderfächern zugeordnet ist, kann das anzugebende Sonderfach gewählt werden. Additivfächer werden nicht angegeben.
| | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.6 ELGA_AuthorSpeciality (DYNAMIC) |
| | TEL.AT | 0 … * | |
Kontaktdaten des Verfassers des Dokuments.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:assignedPerson welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
- hl7:assignedAuthoringDevice welches enthält Template 1.2.40.0.34.6.0.11.9.18 Device Compilation (DYNAMIC)
| | | 0 … 1 | |
Personendaten des Verfassers des Dokuments.
Grundsätzlich sind die Vorgaben für „Personen-Element“ zu befolgen, name-Element ist hier Mandatory.
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | |  |  |  |  | hl7:assignedAuthoringDevice
|
| | 0 … 1 | | Datenerstellende/s Software/Gerät
Beinhaltet 1.2.40.0.34.6.0.11.9.18 Device Compilation (DYNAMIC) | |  |  |  | hl7:representedOrganization
|
| | 1 … 1 | M | Organisation, in deren Auftrag der Verfasser des Dokuments die Dokumentation verfasst hat.
↔ Hinweis zum XDS-Mapping: Da manche offiziellen Bezeichnungen von GDA sehr lang werden können, SOLL das name Element einer möglichst eindeutigen Kurzbezeichnung der Organisation entsprechen (im GDA-I im Tag description enthalten). Bei größeren Organisationen SOLL zusätzlich die Abteilung angegeben werden, damit die Zuordnung für den Leser einfacher wird.
Beispiel: Statt "Allgemeines Krankenhaus der Stadt Wien-Medizinischer Universitätscampus" --> "Wien AKH" bzw. "Wien AKH - Augenambulanz"
Beinhaltet 1.2.40.0.34.6.0.11.9.5 Organization Compilation with id, name (DYNAMIC) | | | | Constraint |
- id MUSS der OID der Organisation aus dem GDA-Index entsprechen.
- name SOLL der Kurzbezeichnung im GDA-I entsprechen (sofern vorhanden)
- Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden., z.B.: „Amadeus Spital, Chirurgische Abteilung“
-
Ausnahme: Wenn als Author ein/e Software/Gerät fungiert und keine OID aus dem GDA-I angegeben werden kann, MÜSSEN die Angaben der Organisation des Geräte-/Software-Betreibers oder Herstellers entsprechen.
| | | Schematron assert | role | error | | | | test | count(hl7:author/hl7:assignedAuthor/hl7:assignedPerson)>0 | | | | Meldung | Es MUSS immer zumindest eine Person als Autor angeführt sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.22 Data Enterer (DYNAMIC) |  | hl7:dataEnterer
|
| | 0 … 1 | |
z.B. Schreibkraft, Medizinische Dokumentationsassistenz
| | | |  | | at-cda-bbr-dataelement-16 |  Schreibkraft Schreibkraft |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | ENT |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:time
|
| TS.AT.TZ | 0 … 1 | R |
Der Zeitpunkt zu dem die Daten dokumentiert wurden.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-17 |  Zeitpunkt des Schreibens Zeitpunkt des Schreibens |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | hl7:assignedEntity
|
| | 1 … 1 | M | Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.4 Custodian (DYNAMIC) |  | hl7:custodian
|
| | 1 … 1 | M | Verwahrer des Dokuments. | | | |  | | at-cda-bbr-dataelement-24 |  Verwahrer Verwahrer |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | CST |  |  | hl7:assignedCustodian
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ASSIGNED |  |  |  | hl7:representedCustodianOrganization
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 1 … * | M | Identifikation des Verwahrers des Dokuments. Wenn dieser im GDA-I angeführt ist, ist die entsprechende OID zu verwenden.
Grundsätzlich sind die Vorgaben für „Identifikations-Elemente“ zu befolgen. | | | ON | 1 … 1 | M | Name des Verwahrers des Dokuments (Organisation). Grundsätzlich sind die Vorgaben für „Namen-Elemente von Organisationen ON“ zu befolgen. | | | TEL.AT | 0 … * | | Kontaktdaten des Verwahrers des originalen Dokuments (Organisation). Grundsätzlich sind die Vorgaben für „Kontaktdaten-Elemente“ zu befolgen. | | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R | | | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 1 … 1 | M | Adresse des Verwahrers des Dokuments (Organisation). Grundsätzlich sind die Vorgaben für „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.24 Information Recipient (DYNAMIC) |  | hl7:informationRecipient
|
| | 0 … * | | Beabsichtiger Empfänger des Dokuments.
| | | |  | | at-cda-bbr-dataelement-26 |  Empfänger Empfänger |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | | Typ des Informationsempfängers, z.B: PRCP „Primärer Empfänger“.
Werden mehrere Empfänger angegeben, MUSS der primäre Empfänger über den typeCode definiert werden.
Hinweis: Das ist relevant, wenn Funktionen aus dem gerichteten Befundversand oder für den Briefdruck auf das Dokument angewendet werden. | | | CONF | | Der Wert von @typeCode muss gewählt werden aus dem Value Set 1.2.40.0.34.10.29 ELGA_InformationRecipientType (DYNAMIC) |
| | |  | | at-cda-bbr-dataelement-27 |  Empfänger Typ Empfänger Typ |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | hl7:intendedRecipient
|
| | 1 … 1 | M | | | | cs | 0 … 1 | | | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | II | 0 … * | | Identifikation des beabsichtigten Empfängers (Person).
Empfohlene Information für einen Empfänger ist die ID aus dem GDA-Index.
Grundsätzlich sind die Vorgaben für „Identifikations-Elemente“ zu befolgen. | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-28 |  ID des Empfängers ID des Empfängers |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | II | 0 … 1 | | NI … Person hat keine ID | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | UNK ... Person hat eine ID, diese ist jedoch unbekannt | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … 1 | | Personendaten des beabsichtigten Empfängers.
Empfehlung: Der Name des Empfängers und die Organisation, der er angehört, sollen in möglichst hoher Granularität angegeben werden. Aufgrund der gängigen Praxis kann als minimale Information für den Empfänger der unstrukturierte Name angegeben werden.
Grundsätzlich sind die Vorgaben gemäß Kapitel „Personen-Element“ zu befolgen. Elemente in der Auswahl:- hl7:informationRecipient[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:informationRecipient[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
|  |  |  |  | hl7:informationRecipient
|
| | … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | |  | | at-cda-bbr-dataelement-29 |  Name Name |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  |  |  | hl7:informationRecipient
|
| | … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | |  |  |  | hl7:receivedOrganization
|
| | 0 … 1 | R | Organisation, der der beabsichtigte Empfänger angehört, z.B.: „Ordination des empfangenden Arztes“.
Grundsätzlich sind die Vorgaben gemäß Kapitel „Organisations-Element“ zu befolgen. | | | |  | | at-cda-bbr-dataelement-30 |  Organisation Organisation |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.5 Legal Authenticator (DYNAMIC)
"Medizinischer Validator" oder der laborverantwortliche Arzt |  | hl7:legalAuthenticator
|
| | 1 … 1 | M | Hauptunterzeichner, Rechtlicher Unterzeichner
| | | |  | | at-cda-bbr-dataelement-1 |  Rechtlicher Unterzeichner Rechtlicher Unterzeichner |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | @typeCode
|
| cs | 0 … 1 | F | LA | | Auswahl | 1 … 1 | | Der Zeitpunkt, an dem das Dokument unterzeichnet wurde.
Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='UNK']
| | TS.AT.TZ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-5 |  Zeitpunkt der Unterzeichnung Zeitpunkt der Unterzeichnung |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | TS.AT.TZ | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK |  |  | hl7:signatureCode
|
| CS | 1 … 1 | M | Signaturcode gibt an, dass das Originaldokument unterzeichnet wurde.
| | | |  | | at-cda-bbr-dataelement-6 |  Signatur Signatur |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | CONF | 1 … 1 | F | S |  |  | hl7:assignedEntity
|
| | 1 … 1 | M | Personendaten des rechtlichen Unterzeichners.
Für den Namen ist verpflichtend Granularitätsstufe 2 ("strukturierte Angabe des Namens") anzuwenden!
Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.49 Laboratory Results Validator (DYNAMIC)
Validator (Authenticator) |  | hl7:authenticator
|
| | 0 … * | | (Weitere) validierende Person (=Mitunterzeichner), die das Dokument inhaltlich (medizinisch und technisch) freigibt. Es können mehrere Validatoren angegeben werden. Einer davon kann auch ident mit dem "rechtlichen Unterzeichner" (/ClinicalDocument/legalAuthenticator) sein. | |  |  | @typeCode
|
| cs | 0 … 1 | F | AUTHEN |  |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Results Validator | | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.49 |  |  | hl7:templateId
|
| II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.2.16 Laboratory Results Validator | | | uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.1.5 | | Auswahl | 1 … 1 | | Der Zeitpunkt, an dem das Dokument unterzeichnet wurde.
Grundsätzlich sind die Vorgaben gemäß für "Zeit-Elemente" zu befolgen. Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='UNK']
| | TS.AT.TZ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | TS.AT.TZ | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | |  |  | hl7:signatureCode
|
| CS | 1 … 1 | M | | | | CONF | 1 … 1 | F | S |  |  | hl7:assignedEntity
|
| | 1 … 1 | M | Personendaten des weiteren Unterzeichners.
Beinhaltet 1.2.40.0.34.6.0.11.9.41 Assigned Entity with id, name, addr and telecom (DYNAMIC) | | | Auswahl | 1 … 1 | | Auftraggeber / Ordering Provider
Elemente in der Auswahl:- hl7:participant eingefügt vom Template 1.2.40.0.34.6.0.11.1.42 Participant Auftraggeber / Ordering Provider (DYNAMIC)
- hl7:participant[@typeCode='REF'][@nullFlavor]
| | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.42 Participant Auftraggeber / Ordering Provider (DYNAMIC) |  |  | hl7:participant
|
| | 0 … 1 | | | | | cs | 1 … 1 | F | REF | | cs | 0 … 1 | F | OP | | II | 1 … 1 | M | Participant Auftraggeber / Ordering Provider | | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.42 | | II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.2.17 Ordering Provider | | | uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.1.6 | | Auswahl | 1 … 1 | | Das Auftragsdatum ist das Datum/Zeit, an dem der Auftrag vom Auftraggeber abgesendet wird. Das Auftragsdatum wird als "time"-Element beim Auftraggeber ausgeführt und ist verpflichtend anzugeben. Bei einer manuellen Erfassung eines Auftrags im Labor kann dieses als @nullFlavor="NA" ausgeführt werden. Elemente in der Auswahl:- hl7:time[not(@nullFlavor)]
- hl7:time[@nullFlavor='NA']
| | IVL_TS | 0 … 1 | R | | | | wo [not(@nullFlavor)] | | | IVL_TS | 0 … 1 | R | | | | wo [@nullFlavor='NA'] | | | | 1 … 1 | M | | | | cs | 1 … 1 | F | PROV | | | Healthcare provider - Gesundheitsdienstanbieter
| | II | 1 … 1 | M | ID des Auftraggebers | | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:addr[not(@nullFlavor)] welches enthält Template 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC)
- hl7:addr[@nullFlavor='UNK']
| | AD | 0 … 1 | R | Adresse des Auftraggebers
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | AD | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:telecom[not(@nullFlavor)]
- hl7:telecom[@nullFlavor='UNK']
| | TEL.AT | 0 … * | R | Beliebig viele Kontaktdaten des Auftraggebers
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | | Schematron assert | role | error | | | | test | not(hl7:telecom[not(@nullFlavor)]) or not(hl7:telecom[@nullFlavor='UNK']) | | | | Meldung | telecom[@nullFlavor="UNK"] darf NUR angegeben werden, wenn KEIN befülltes "telecom"-Element vorhanden ist. | | | Auswahl | 1 … 1 | | Name des Auftraggebers.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
- hl7:associatedPerson[@nullFlavor]
| | | 0 … 1 | R | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | | | | | wo [@nullFlavor] | | | cs | 1 … 1 | F | UNK | | | 0 … 1 | R |
Organisation, der der Auftraggeber angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für "Organisations-Element" zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | |  |  | hl7:participant
|
| | 0 … 1 | | Auftraggeber nicht bekannt | | | wo [@typeCode='REF'] [@nullFlavor] | | | cs | 1 … 1 | F | REF | | cs | 1 … 1 | F | UNK | | | Beispiel | Auftraggeber nicht bekannt <participant typeCode="REF" nullFlavor="UNK">
<associatedEntity classCode="PROV"/></participant> | | | 1 … 1 | M | | | | cs | 1 … 1 | F | PROV |  | hl7:participant
|
| | | NP | Die Verwendung des ELGA participant-Elements, das den einweisenden/zuweisenden/überweisenden Arzt repräsentiert mit templateId 1.2.40.0.34.6.0.11.1.21 ist im Mikrobiologiebefund NICHT ERLAUBT. | | | wo [@typeCode='REF'] [hl7:templateId/@root='1.2.40.0.34.6.0.11.1.21'] | | | Eingefügt | 0 … 1 | R | von 1.2.40.0.34.6.0.11.1.20 Participant Fachlicher Ansprechpartner (DYNAMIC)
Fachlicher Ansprechpartner
Es ist EMPFOHLEN, den fachlichen Ansprechpartner (Callback contact) im Labor- und Mikrobiologiebefund anzugeben.
|  | hl7:participant
|
| | 0 … 1 | R | Fachlicher Ansprechpartner
| | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.20']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | CALLBCK | | | Callback contact
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.20 |  |  | hl7:functionCode
|
| CE (extensible) | 0 … 1 | |
Optionale Angabe eines Funktionscodes des fachlichen Ansprechpartners, z.B: „Diensthabender Oberarzt“, „Verantwortlicher Arzt für Dokumentation“,„Stationsschwester“.
Eigene Codes und Bezeichnungen können verwendet werden.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 1 … 1 | R | |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | PROV | | |
Healthcare provider - Gesundheitsdiensteanbieter
| | CE | 0 … 1 | |
Optionale Angabe der Fachrichtung des fachlichen Ansprechpartners („Sonderfach“ gem. Ausbildungsordnung), z.B: „Facharzt/Fachärztin für Gynäkologie“.
Wenn ein fachlicher Ansprechpartner mehreren ärztlichen Sonderfächern zugeordnet ist, kann das anzugebende Sonderfach gewählt werden. Additivfächer werden nicht angegeben.
| | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.6 ELGA_AuthorSpeciality (DYNAMIC) |
| | AD | 0 … 1 | |
Adresse des Beteiligten.
Grundsätzlich sind die Vorgaben für "Adress-Elemente" zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 1 … * | M | Beliebig viele Kontaktdaten des Beteiligten. | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Es MUSS mindestens eine Telefonnummer angegeben werden. Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein. | | | 0 … 1 | R |
Name der Person
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | | 0 … 1 | R |
Organisation, der der Beteiligte angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für "Organisations-Element" zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | | Schematron assert | role | error | | | | test | not(hl7:participant[@typeCode='CALLBCK'][@nullFlavor]) | | | | Meldung | @nullFlavor ist für den fachlichen Ansprechpartner (participant[@typeCode='CALLBCK']) NICHT ERLAUBT. Sollten keine Informationen vorliegen, soll das Element entfallen. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.23 Participant Hausarzt (DYNAMIC) |  | hl7:participant
|
| | 0 … 1 | | Beteiligter (Hausarzt). | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.23']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND | | | In indirektem Bezug.
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.23 |  |  | hl7:functionCode
|
| CE | 1 … * | M |
Funktionscode des Beteiligten
| | | cs | 1 … 1 | F | PCP | | oid | 1 … 1 | F | 2.16.840.1.113883.5.88 | | st | 1 … 1 | F | HL7:ParticipationFunction |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | PROV | | | Healthcare provider - Gesundheitsdiensteanbieter.
| | Auswahl | 0 … * | |
Identifikation des Beteiligten (Person) aus dem GDA-Index.
Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | | Constraint |
Zugelassene nullFlavor:
- NI … Organisation hat keine ID
- UNK … Organisation hat eine ID, diese ist jedoch unbekannt
| | II | 0 … * | | | | | wo [not(@nullFlavor)] | | | II | 0 … 1 | | | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | AD | 0 … 1 | | Adresse des Hausarztes
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | | Beliebig viele Kontaktdaten des Hausarztes.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R | | | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Name des Hausarztes.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | R |
Arztpraxis oder Ordination.
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.27 Participant Auskunftsberechtigte Person (Notfallkontakt) (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Notfallkontakt / Auskunftsberechtigte Person)
| | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.27']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND | | | In indirektem Bezug.
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.27 |  |  | hl7:time
|
| IVL_TS | 0 … 1 | |
Zeitraum, in dem der angegebene Kontakt den Notfall-Kontakt darstellt.
Wird nur angegeben, wenn der Kontakt bereits absehbar nur in einem eingeschränkten Zeitraum zur Verfügung steht.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | ECON | | | Emergency contact - Notfall-Kontakt
| | CE | 0 … 1 | | Verwandtschaftsverhältnis des Beteiligten zum Patienten, z.B. DAU („daughter“), wenn die Beteiligte die Tochter des Patienten ist. | | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | Zulässige Werte gemäß Value-Set „ELGA_PersonalRelationship“
| | st | 0 … 1 | | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.111 | | st | 1 … 1 | F | HL7:RoleCode | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.17 ELGA_PersonalRelationship (DYNAMIC) |
| | AD | 0 … 1 | | Adresse des Beteiligten
Grundsätzlich sind die Vorgaben gemäß „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Auswahl | 0 … * | | Beliebig viele Kontaktdaten des Beteiligten. Elemente in der Auswahl:- hl7:telecom[not(@nullFlavor)]
- hl7:telecom[@nullFlavor='UNK']
| | | Constraint | Es SOLL mindestens eine Telefonnummer angegeben werden. | | TEL.AT | 0 … * | R | | | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | | | | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … 1 | | Die Kontaktadresse ist unbekannt. nullFlavor "UNK" | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | Auswahl | 1 … 1 | | Name des Beteiligten.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | R |
Organisation, der der Beteiligte angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.25 Participant Angehoerige (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Angehöriger)
| | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.25']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND | | | In indirektem Bezug.
|  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.25 |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | PRS | | | Personal relationship - In persönlicher Beziehung
| | CE | 1 … 1 | M | Verwandtschaftsverhältnis des Beteiligten zum Patienten. Beispiel: DAU („daughter“), wenn die Beteiligte die Tochter des Patienten ist oder NBOR für Nachbar. | | | cs | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.17 ELGA_PersonalRelationship (DYNAMIC) |
| | st | 0 … 1 | | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.111 | | st | 1 … 1 | F | HL7:RoleCode | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.17 ELGA_PersonalRelationship (DYNAMIC) |
| | AD | 0 … 1 | | Adresse des Beteiligten
Grundsätzlich sind die Vorgaben gemäß „Adress-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | | Beliebig viele Kontaktdaten des Beteiligten.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R | | | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | Auswahl | 1 … 1 | | Name des Beteiligten.
Elemente in der Auswahl:- hl7:associatedPerson[hl7:name[count(child::*)=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC)
- hl7:associatedPerson[hl7:name[count(child::*)!=0]] welches enthält Template 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.12 Person Name Compilation G1 M (DYNAMIC) | | | wo [hl7:name [count(child::*)=0]] | | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | wo [hl7:name [count(child::*)!=0]] | | | | 0 … 1 | R | Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.26 Participant Versicherung (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Versicherter/Versicherung). | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.26']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | HLD |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.26 |  |  | hl7:time
|
| IVL_TS | 0 … 1 | |
Gültigkeitszeitraum der Versicherungspolizze.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | POLHOLD | | | Policy holder - Halter einer Versicherungspolizze
| | Auswahl | 1 … 1 | |
Sozialversicherungsnummer des Patienten (SELF) oder der Person, bei der der Patient mitversichert ist (FAMDEP)
Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='NI']
- hl7:id[@nullFlavor='UNK']
| | | Constraint |
Zugelassene nullFlavor:
- NI … Patient hat keine Sozialversicherungsnummer (z.B. Ausländer, …)
- UNK … Patient hat eine Sozialversicherungsnummer, diese ist jedoch unbekannt
| | II | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | II | 0 … 1 | | | | | wo [@nullFlavor='NI'] | | | cs | 1 … 1 | F | NI | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | cs | 1 … 1 | F | UNK | | CE | 1 … 1 | M |
Versicherungsverhältnis codiert
Beispiele:
- SELF, wenn der Patient selbst der Versicherte ist.
- FAMDEP, wenn der Patient bei einem Familienmitglied mitversichert ist.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | F | 2.16.840.1.113883.5.111 | | st | 1 … 1 | F | HL7:RoleCode | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.9 ELGA_InsuredAssocEntity (DYNAMIC) |
| | AD | 0 … 1 | | Adresse des Beteiligten.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | | Beliebig viele Kontaktdaten des Beteiligten.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | | 0 … 1 | C | Name des Beteiligten.
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | | Constraint | Wenn das Versicherungsverhältnis "familienversichert" ("FAMDEP“) ist, MUSS eine associatedPerson angegeben sein, M [1..1], sonst kann sie komplett entfallen, O [0..1] | | | 1 … 1 | M |
Versicherungsgesellschaft.
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | | Schematron assert | role | error | | | | test | not(hl7:code[@code='FAMDEP']) or hl7:associatedPerson | | | | Meldung | Wenn das Versicherungsverhältnis "familienversichert" ist, dann muss eine associatedPerson angegeben sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.29 Participant Betreuungsorganisation (DYNAMIC) |  | hl7:participant
|
| | 0 … 1 | | Beteiligter (Betreuende Organisation) | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.29']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | IND |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.29 |  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | CAREGIVER | | | Betreuer
| | | 1 … 1 | M | Betreuende Organisation | | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 0 … * | | von 1.2.40.0.34.6.0.11.1.28 Participant Weitere Behandler (DYNAMIC) |  | hl7:participant
|
| | 0 … * | | Beteiligter (Weitere Behandler) | | | wo [hl7:templateId [@root='1.2.40.0.34.6.0.11.1.28']] | |  |  | @typeCode
|
| cs | 1 … 1 | F | CON |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP |  |  | hl7:templateId
|
| II | 1 … 1 | M | Template ID zur Identifikation dieser Art von Beteiligten
| | | uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.1.28 |  |  | hl7:functionCode
|
| CE (extensible) | 0 … 1 | | Funktionscode des Behandlers z.B: „Facharzt für Neurologie“
Eigene Codes und Bezeichnungen dürfen verwendet werden. | | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code sollte gewählt werden aus dem Value Set 1.2.40.0.34.10.6 ELGA_AuthorSpeciality (DYNAMIC) |
|  |  | hl7:associatedEntity
|
| | 1 … 1 | M | Beschreibung der Entität.
| | | cs | 1 … 1 | F | PROV | | | Gesundheitsdiensteanbieter.
| | AD | 0 … 1 | |
Adresse des Beteiligten.
Grundsätzlich sind die Vorgaben gemäß „Adress-Elemente“ zu befolgen
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | TEL.AT | 0 … * | |
Beliebig viele Kontaktdaten des Beteiligten.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.)
Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“
Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …)
Bsp: WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
Bei Angabe mehrerer Telefonnummern ist jeweils das Attribut @use anzugeben.
| | | 1 … 1 | M |
Beteiligte Person
Grundsätzlich sind die Vorgaben für „Personen-Element“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.11 Person Name Compilation G2 M (DYNAMIC) | | | | 0 … 1 | R |
Organisation, der der Beteiligte angehört (mit Adresse und Kontaktdaten der Organisation).
Grundsätzlich sind die Vorgaben für „Organisations-Element“ zu befolgen.
| | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | | cs | 0 … 1 | F | ORG | | cs | 0 … 1 | F | INSTANCE | | II | 0 … * | | Beliebig viele IDs der Organisation. z.B.: ID aus dem GDA-Index, DVR-Nummer, ATU-Nummer, etc. | | | wo [not(@nullFlavor)] | | | ON | 1 … 1 | M | Name der Organisation. Bei Organisationen, die im GDA-Index angegeben sind, soll deren Kurzbezeichnung verwendet werden.
Zu dem Namen größerer Organisationen SOLL auch die Abteilung angegeben werden.
| | | TEL.AT | 0 … * | |
Kontaktdaten der Organisation.
Grundsätzlich sind die Vorgaben für „Kontaktdaten-Element“ zu befolgen.
| | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Die Kontaktadresse (Telefonnummer, Email, etc.), z.B. tel:+43.1.1234567Formatkonvention siehe „telecom – Format Konventionen für Telekom-Daten“ Zulässige Werteliste für telecom Präfixe gemäß „ELGA_URLScheme“
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set „ELGA_TelecomAddressUse“
| | | Constraint | Werden mehrere gleichartige telecom-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | AD | 0 … 1 | | Adresse der Organisation.
Beinhaltet 1.2.40.0.34.6.0.11.9.25 Address Compilation (DYNAMIC) | | | wo [not(@nullFlavor)] | | | Eingefügt | 1 … * | M | von 1.2.40.0.34.6.0.11.1.9 In Fulfillment Of (DYNAMIC) | | | Constraint | Da die Referenz auf einen Auftrag im Labor eine wesentliche Information darstellt, ist dieses Element VERPFLICHTEND anzugeben. |  | hl7:inFulfillmentOf
|
| | 1 … * | M | Komponente zur Dokumentation des Auftrags.
| | | |  | | at-cda-bbr-dataelement-42 |  Auftrag Auftrag |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 1 … 1 | F | FLFS |  |  | hl7:order
|
| | 1 … 1 | M | Auftrag.
| | | cs | 1 … 1 | F | ACT | | cs | 1 … 1 | F | RQO | | II | 1 … 1 | M | Auftragsnummer, Anforderungsnummer.
Grundsätzlich sind die Vorgaben gemäß Kapitel „Identifikations-Elemente“ zu befolgen.
| | | |  | | at-cda-bbr-dataelement-43 |  ID ID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | Eingefügt | 1 … * | M | von 1.2.40.0.34.6.0.11.1.48 Documentation Of Service Event - Labor und Mikrobiologie (DYNAMIC) |  | hl7:documentationOf
|
| | 1 … * | M | Komponente für die Gesundheitsdienstleistung.
| |  |  | @typeCode
|
| cs | 0 … 1 | F | DOC |  |  | hl7:serviceEvent
|
| | 1 … 1 | M | Die serviceEvents in den ELGA Labor- und Mikrobiologiebefunden MÜSSEN die "section/code"-Elemente als auch die "section/templateId"-Elemente wiedergeben. Diese Informationen werden über eine Mapping-Vorschrift in die XDS-Metadaten übernommen und ermöglichen einem ELGA-Teilnehmer zu erkennen, welche Sections beinhaltet sind und in welchem Codierungsgrad diese vorliegen. Daher muss für jede Section, welche medizinische Information enthält (Ausnahmen sind "Brieftext" und "Abschließende Bemerkungen"), ein documentationOf/serviceEvent codiert werden.
| | | cs | 0 … 1 | F | ACT | | cs | 0 … 1 | F | EVN | | II | 0 … 1 | C | | | | st | 0 | NP | NP/nicht anwesend | | uid | 1 … 1 | R | In das serviceEvent/id[@root] MUSS die section/templateId[@root] geschrieben werden. Im Fall von mehreren "section/templateId"-Elementen MUSS jenes gewählt werden, dessen @id-Attribut in dem OID-Bereich 1.2.40.0.34.6.0.11.2.X zu finden ist. | | | Constraint | Grundsätzlich MUSS serviceEvent/id angegeben werden. Die serviceEvent/id IST NICHT ERLAUBT für das zusätzliche serviceEvent mit dem Code "18725-2 - Microbiology studies (set)" für Mikrobiologiebefunde bzw. Laborbefunde, die mikrobiologische Ergebnisse beinhalten. | | | Schematron assert | role | error | | | | test | hl7:code[@code='18725-2'][@codeSystem='2.16.840.1.113883.6.1'] or hl7:id | | | | Meldung | serviceEvent/id MUSS angegeben werden | | | | Schematron assert | role | error | | | | test | not(hl7:code[@code='18725-2'][@codeSystem='2.16.840.1.113883.6.1'] and hl7:id) | | | | Meldung | serviceEvent/id DARF NICHT angegeben werden | | | CE | 1 … 1 | M | Code der Gesundheitsdienstleistung.
↔ Hinweis zum XDS-Mapping:
Dieses Element wird in das XDS-Attribut "eventCodeList" gemappt.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.22 ELGA_ServiceEventsLabor (DYNAMIC) |
| | IVL_TS | 1 … 1 | M | Angabe des zeitlichen Erbringungsintervalls effectiveTime mit einer Start- "low" und Endzeit "high" (verpflichtend).
Startzeitpunkt: Datum und Zeitpunkt, an dem das analysierende Labor die Anforderung vom Zuweiser in der Labor EDV erfasst hat. Falls nicht vorhanden, sind Datum und Uhrzeit des Starts des Auftrags in der Labor EDV anzugeben.
Endzeit: Datum und Zeitpunkt des Abschlusses des Auftrags, welche in der Regel mit der medizinischen Freigabe des Auftrags ident ist.
↔ Hinweis zum XDS-Mapping:
Dieses Element wird in die XDS-Attribute "serviceStartTime" und "serviceStopTime" gemappt.
Für die automatisierte Datenübernahme aus dem CDA-Dokument in die XDS-Dokumentmetadaten ist stets ein Zeitintervall anzugeben.
ACHTUNG: Die Zeitangaben der jeweils ersten Gesundheitsdienstleistung (erstes "documentationOf/serviceEvent"-Element) werden in die Dokument-Metadaten übernommen!
Die Bedeutung der Dokument-Metadaten-Elemente lautet daher wie folgt:
- serviceStartTime: Beginn des ersten documentationOf/serviceEvent-Elements
- serviceStopTime: Ende des ersten documentationOf/serviceEvent-Elements
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | | | | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. | | Eingefügt | 0 … 1 | C | von 1.2.40.0.34.6.0.11.1.14 Document Replacement - Related Document (DYNAMIC) | | | Constraint | Wird ein Befund aktualisiert, weil z.B. zuvor noch Analyseergebnisse ausständig waren, MUSS dieses Element angegeben werden. |  | hl7:relatedDocument
|
| | 0 … 1 | C | | | | |  | | at-cda-bbr-dataelement-15 |  Bezug zu vorgehenden Dokumenten Bezug zu vorgehenden Dokumenten |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 1 … 1 | R |
Art des Bezugs zum Vordokument.
| | | Constraint |
Erlaubte @typeCodes:
RPLC - replaces: Das Dokument ersetzt ein existierendes Dokument. Der Status des zu ersetzenden Dokumentes wird auf "deprecated" gesetzt, das ursprüngliche Dokument bleibt aber noch im System als historische Referenz verfügbar.
APND - append: Zusammenhängen von Dokumenten. Dies ist in ELGA bereits über das Einbetten von Dokumenten realisiert.
XFRM - transformed: Das Dokument ist Ergebnis eines Transformationsprozesses, d.h. ist aus einem anderen Originaldokument hervorgegangen.
Hinweis: Die parallele Ablage von CDA-Dokumenten, welche vom Dokumentersteller bereits mit einem Stylesheet zu einem PDF Dokument gerendert wurden, kann mit der XFRM – Transaktion vorgenommen werden. Es ist nicht auszuschließen, dass die Transformation in lokalen Affinity Domains Anwendung findet. Für ELGA ist die Transformation jedoch kein Anwendungsfall.
|  |  | hl7:parentDocument
|
| | 1 … 1 | M | Vorhergehendes Dokument.
| | | cs | 0 … 1 | F | DOCCLIN | | cs | 0 … 1 | F | EVN | | II | 1 … 1 | M | Dokumenten-Id des vorgehenden Dokuments.
Grundsätzlich sind die Vorgaben für „Identifikations-Elemente“ zu befolgen.
| | | | Schematron assert | role | error | | | | test | not(hl7:relatedDocument) or hl7:relatedDocument[@typeCode='RPLC'] | | | | Meldung | Wird /ClinicalDocument/relatedDocument angegeben, MUSS relatedDocument[@typeCode='RPLC'] sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.1.50 Component Of - Encompassing Encounter with id (DYNAMIC) |  | hl7:componentOf
|
| | 0 … 1 | | Komponente für den Patientenkontakt.
| | | |  | | at-cda-bbr-dataelement-33 |  Patientenkontakt Patientenkontakt |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | hl7:encompassingEncounter
|
| | 1 … 1 | M | Patientenkontakt.
| | | cs | 0 … 1 | F | ENC | | cs | 0 … 1 | F | EVN | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='UNK']
| | II | 0 … 1 | | Identifikationselement zur Aufnahme der Aufenthaltszahl | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-34 |  ID ID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | st | 1 … 1 | R | Aufenthaltszahl, z.B.: Az123456
| | uid | 1 … 1 | R | OID der Liste der Aufenthaltszahlen der Organisation
| | st | 0 … 1 | | Name der Stelle, welche die ID zugewiesen hat, z.B.: "Amadeus Spital". | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | CE | 1 … 1 | M | Codierung des Patientenkontakts.
| | | |  | | at-cda-bbr-dataelement-39 |  Art des Aufenthalts Art des Aufenthalts |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | F | HL7:ActCode | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.5 ELGA_ActEncounterCode (DYNAMIC) |
| | IVL_TS | 1 … 1 | M |
Zeitraum des Patientenkontakts.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | | | |  | | at-cda-bbr-dataelement-37 |  Beginn des Patientenkontaktes Beginn des Patientenkontaktes |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Der Zeitraum des Patientenkontaktes MUSS die Vorgaben der speziellen Implementierungsleitfäden einhalten. Dabei gilt allgemein:
- Der Zeitraum besteht aus dem Zeitpunkt der administrativen Aufnahme in die Behandlung und dem Zeitpunkt der administrativen Entlassung aus der Behandlung.
- Der Entlassungszeitpunkt kann „unbekannt“ sein, wenn die administrative Entlassung noch nicht erfolgt ist. (nullFlavor UNK beim effectiveTime.high)
- Hinweis: Als Zeitpunkt der Aufnahme/Entlassung SOLL der Zeitpunkt der administrativen Aufnahme/Entlassung angegeben werden. Wenn der Zeitpunkt der administrativen Aufnahme/Entlassung nicht vorhanden ist, darf auch der Zeitpunkt der medizinischen Aufnahme/Entlassung angegeben werden.
| | | 0 … 1 | R |
Komponente für die verantwortliche Person.
| | | |  | | at-cda-bbr-dataelement-40 |  Verantwortliche Person Verantwortliche Person |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | 1 … 1 | M |
Entität der verantwortlichen Person.
Grundsätzlich sind die Vorgaben für „AssignedEntity-Element (Person + Organisation)“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.8 Encounter Location (DYNAMIC)
Die Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand, MUSS verpflichtend angegeben werden (z.B.: die entlassende Krankenanstalt mit Abteilung).
| | | 1 … 1 | M | | | | cs | 0 … 1 | F | LOC | | | 1 … 1 | M | | | | cs | 0 … 1 | F | SDLOC | | CE | 1 … 1 | M |
Der Code zur Klassifizierung des GDA repräsentiert die Art der Einrichtung, in der die Tätigkeit stattfand, die zur Erzeugung des Dokuments führte. Zum Beispiel sollten Dokumente, die während eines ambulanten Falls in einem Krankenhaus entstehen, mit dem healthcareFacilityTypeCode für „Krankenhaus“ gekennzeichnet werden.
Zulässige Werte gemäß Value-Set „ELGA_HealthcareFacilityTypeCode“ Für ELGA SOLL der Code dem Eintrag "GDA Rollenname" oder, wenn der GDA Rollenname nicht verfügbar ist, der "Aggregierten Rolle" im GDA-I entsprechen.
↔ Hinweis zum XDS-Mapping: Dieses Element wird ins XDS-Attribut XDSDocumentEntry.healthcareFacilityTypeCode gemappt.
Zu berücksichtigen sind jeweils die Attribute @code, @codeSystem und @displayName.
| | | st | 1 … 1 | R | |  |  |  |  |  | hl7:serviceProviderOrganization
|
| | 1 … 1 | M |
Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand.
Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | |  | hl7:component
|
| | 1 … 1 | M | | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| bl | 0 … 1 | F | true |  |  | hl7:structuredBody
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | DOCBODY | | cs | 0 … 1 | F | EVN | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.69 Brieftext (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.6 Überweisungsgrund - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.114 Überweisungsgrund - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.6 Überweisungsgrund - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.114 Überweisungsgrund - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.109 Anamnese - Labor und Mikrobiologie - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.111 Anamnese - Labor und Mikrobiologie - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.109 Anamnese - Labor und Mikrobiologie - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.111 Anamnese - Labor und Mikrobiologie - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.15 Angeforderte Untersuchungen - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.112 Angeforderte Untersuchungen - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.15 Angeforderte Untersuchungen - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.112 Angeforderte Untersuchungen - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | C | Beinhaltet 1.2.40.0.34.6.0.11.2.93 Probeninformation (Specimen Section) (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | Constraint | Die Dokumentation des Untersuchungsmaterials kann auf zwei Arten erfolgen: - Enthält ein Befund nur einen Befundbereich ("Laboratory Specialty Section"), so kann die Codierung innerhalb der einen Section erfolgen.
ODER - Bei Verwendung von mehreren Befundbereichen in einem Befund kann es zu Überschneidungen der
Untersuchungsmaterialien kommen (ein spezielles Untersuchungsmaterial kann in zwei Befundbereichen analysiert werden). Die CDA Level 3 Codierung eines Untersuchungsmaterials darf jedoch nur einmal im gesamten Befund erfolgen. Daher sind die Informationen zu den Untersuchungsmaterialien in einer eigenen, führenden "Probeninformation (Specimen Section)" zu
codieren.
WICHTIG: Derzeit werden Mikrobiologiebefunde mit mehreren Untersuchungsmaterialien NICHT unterstützt. Das bedeutet, dass in einem Mikrobiologiebefund nur genau ein Untersuchungsmaterial dokumentiert werden darf. Falls mehrere Untersuchungsmaterialien zu einem Auftrag eingesendet wurden, müssen diese auf mehrere Befunde aufgeteilt werden! Die Information kann in der Section "Befundbewertung" festgehalten werden. | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.105 Laboratory Specialty Section (Mikroskopie) - codiert (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.2.113 Laboratory Specialty Section (Mikroskopie) - uncodiert (DYNAMIC)
| | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.105 Laboratory Specialty Section (Mikroskopie) - codiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.113 Laboratory Specialty Section (Mikroskopie) - uncodiert (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.106 Laboratory Specialty Section (Kultureller Erregernachweis) (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.107 Laboratory Specialty Section (Molekularer Erregernachweis) (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.108 Laboratory Specialty Section (Infektionsserologie) (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | Schematron assert | role | error | | | | test | count(hl7:component[hl7:section[hl7:templateId[@root = '1.2.40.0.34.6.0.11.2.105']]] | hl7:component[hl7:section[hl7:templateId[@root = '1.2.40.0.34.6.0.11.2.113']]] | hl7:component[hl7:section[hl7:templateId[@root = '1.2.40.0.34.6.0.11.2.106']]] | hl7:component[hl7:section[hl7:templateId[@root = '1.2.40.0.34.6.0.11.2.107']]] | hl7:component[hl7:section[hl7:templateId[@root = '1.2.40.0.34.6.0.11.2.108']]]) >= 1 | | | | Meldung | Zumindest eine der Laboratory Specialty Sections (Mikroskopie, Kultureller Erregernachweis, Molekularer Erregernachweis, Infektionsserologie) muss im Mikrobiologiebefund vorkommen. | | | | 0 … 1 | | Dokumentation weiterer Analysen, die nicht durch die anderen Laboratory Specialty Sections (Mikroskopie, Kultureller Erregernachweis, Molekularer Erregernachweis, Infektionsserologie) abgebildet werden können.
Beinhaltet 1.2.40.0.34.6.0.11.2.110 Laboratory Specialty Section (Weitere Analysen) (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.103 Befundbewertung (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | ELGA Labor- und Mikrobiologiebefunde können alle Kriterien erfüllen, die für Befundberichte von der Akkreditierung für medizinische Laboratorien (ISO 15189:2012) gefordert sind. Ein Anhängen des PDF-Befundes ist NICHT erforderlich!
Für die vollständige Erfüllung aller Akkreditierungsanforderungen an Befundberichte ist das erstellende Labor verantwortlich; die Akkreditierung wird von der Abt. Akkreditierung Austria im Bundesministerium für Digitalisierung und Wirtschaftsstandort durchgeführt.
Mit Rücksicht auf eine einfache Verwendbarkeit der Befunde durch die Benutzer (z.B. niedergelassene Ärzte), die häufig mit einer großen Anzahl von Laborbefunden eines Patienten konfrontiert sind, ist eine Duplizierung der Daten durch Anhängen einer PDF-Ansicht daher nicht gestattet.
Beinhaltet 1.2.40.0.34.6.0.11.2.71 Beilagen (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.2.70 Abschließende Bemerkung (DYNAMIC) | | | cs | 0 … 1 | F | COMP | | bl | 0 … 1 | F | true | | | Schematron assert | role | error | | | | test | count(//hl7:procedure[hl7:templateId/@root='1.2.40.0.34.6.0.11.3.161'])=1 | | | | Meldung | Derzeit werden Mikrobiologiebefunde mit mehreren Untersuchungsmaterialien NICHT unterstützt. | | | Eingefügt | | | von 1.2.40.0.34.6.0.11.9.33 Stylesheet Test eBefund (DYNAMIC) | | | Schematron assert | role | error | | | | test | matches(//processing-instruction('xml-stylesheet'), '[^\w]ELGA_Stylesheet_v1.0.xsl[^\w]') | | | | Meldung | (xml-processing-instr): Es muss ein xml-stylesheet-Prologattribut anwesend sein mit dem Wert für @href=ELGA_Stylesheet_v1.0.xsl | |
|
Wichtiger Hinweis: Header-Elemente welche spezifisch für den Labor- und Mikrobiologiebefund angepasst wurden oder für die es spezielle Einschränkungen in Form von Asserts gibt, sind grundsätzlich der Spezifikation im Kapitel
Document Level Templates zu entnehmen.
Diese angepassten bzw. betroffenen Elemente umfassen:
- /ClinicalDocument/templateId
- /ClinicalDocument/code
- /ClinicalDocument/title
- /ClinicalDocument/sdtc:statusCode
- /ClinicalDocument/formatCode
- /ClinicalDocument/author
Ebenso wurden folgende Header-Elemente speziell für den Labor- und Mikrobiologiebefund erstellt und werden in diesem Kapitel noch einmal detailliert dargestellt:
14.4.3.1 Laboratory Results Validator
| Id | 1.2.40.0.34.6.0.11.1.49 | Gültigkeit ab | 2021‑01‑19 14:09:15 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_header_LaboratoryResultsValidator | Bezeichnung | Laboratory Results Validator |
|---|
| Beschreibung | (Weitere) validierende Person (=Mitunterzeichner), die das Dokument inhaltlich (medizinisch und technisch) freigibt. Es können mehrere Validatoren angegeben werden. Einer davon kann auch ident mit dem "rechtlichen Unterzeichner" (/ClinicalDocument/legalAuthenticator) sein. |
|
| Klassifikation | CDA Header Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.41 | Containment |  Assigned Entity with id, name, addr and telecom (1.0.1+20211213) Assigned Entity with id, name, addr and telecom (1.0.1+20211213) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.6.0.11.1.6 Authenticator (2019‑03‑04 13:11:54) ref at-cda-bbr- |
|---|
| Beispiel | | Strukturbeispiel | <authenticator typeCode="AUTHEN">
<!-- Laboratory Results Validator -->
<templateId root="1.3.6.1.4.1.19376.1.3.3.1.5"/> <!-- IHE PalM TF3 Rev.10, 6.3.2.16 Laboratory Results Validator -->
<templateId root="1.3.6.1.4.1.19376.1.3.3.1.5"/> <!-- Zeitpunkt der Unterzeichnung -->
<time value="20161201121500+0100"/> <!-- Signaturcode -->
<signatureCode code="S"/> <!-- Personen- und Organisationsdaten des weiteren Unterzeichners des Dokuments -->
<assignedEntity>
<!-- Identifikation des weiteren Unterzeichners des Dokuments -->
<id root="1.2.40.0.34.99.4613.3.3" extension="3333" assigningAuthorityName="Amadeus Spital"/> <!-- Adresse des weiteren Unterzeichners -->
<addr>
<streetName>Währinger Gürtel</streetName> <houseNumber>18-20</houseNumber> <postalCode>1090</postalCode> <city>Wien</city> <state>Wien</state> <country>AUT</country> </addr> <!-- Kontaktdaten des weiteren Unterzeichners des Dokuments -->
<telecom use="WP" value="tel:+43.1.3453446.3333"/> <!-- Personendaten des weiteren Unterzeichners des Dokuments -->
<assignedPerson>
<!-- Name des weiteren Unterzeichners des Dokuments -->
<name>
<prefix>Univ.-Prof.Dr.</prefix> <family>Sigrid</family> <given>Kollmann</given> </name> </assignedPerson> <!-- Organisation, in deren Auftrag der weitere Unterzeichner des Dokuments die Dokumentation unterzeichnet hat. -->
<representedOrganization>
<!-- ID der Organisation aus dem GDA Index -->
<id root="1.2.40.0.34.99.4613" assigningAuthorityName="GDA Index"/> <!-- Name der Organisation -->
<name>Amadeus Spital - Labor</name> <!-- Kontaktdaten der Organisation -->
<telecom value="tel:+43.1.3453446.0"/> <telecom value="fax:+43.1.3453446.4674"/> <telecom value="mailto:info@amadeusspital.at"/> <telecom value="http://www.amadeusspital.at"/> <!-- Adresse der Organisation -->
<addr>
<streetName>Währinger Gürtel</streetName> <houseNumber>18-20</houseNumber> <postalCode>1090</postalCode> <city>Wien</city> <state>Wien</state> <country>AUT</country> </addr> </representedOrganization> </assignedEntity></authenticator> |
|
|---|
|
14.4.3.2 Participant Auftraggeber / Ordering Provider
| Id | 1.2.40.0.34.6.0.11.1.42 ref at-cda-bbr- | Gültigkeit ab | 2021‑04‑28 08:55:00Andere Versionen mit dieser Id:  atcdabbr_header_ParticipantAuftraggeber_OrderingProvider vom 2021‑02‑19 11:12:16 atcdabbr_header_ParticipantAuftraggeber_OrderingProvider vom 2021‑02‑19 11:12:16 atcdabbr_header_ParticipantAuftraggeber_OrderingProvider vom 2020‑02‑10 08:46:46 atcdabbr_header_ParticipantAuftraggeber_OrderingProvider vom 2020‑02‑10 08:46:46
|
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.1.0+20211213 |
|---|
| Name | atcdabbr_header_ParticipantAuftraggeber_OrderingProvider | Bezeichnung | Participant Auftraggeber / Ordering Provider |
|---|
| Beschreibung | Der Auftraggeber (IHE "Ordering Provider") ist die Organisation oder der Arzt, welche/welcher den Auftrag erstellt hat. |
|---|
| Klassifikation | CDA Header Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 3 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.25 | Containment |  Address Compilation (1.0.1+20230717) Address Compilation (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.11 | Containment |  Person Name Compilation G2 M (1.0.1+20230717) Person Name Compilation G2 M (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.9 | Containment |  Organization Compilation with name (1.0.0+20210219) Organization Compilation with name (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Version: Template 1.2.40.0.34.6.0.11.1.42 Participant Auftraggeber / Ordering Provider (2021‑02‑19 11:12:16) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.1.42 Participant Auftraggeber / Ordering Provider (2020‑02‑10 08:46:46) ref at-cda-bbr-
Version: Template 1.2.40.0.34.11.1.1.1 HeaderParticipant Ansprechpartner (2014‑03‑25) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <participant typeCode="REF">
<templateId root="1.2.40.0.34.6.0.11.1.42"/> <templateId root="1.3.6.1.4.1.19376.1.3.3.1.6"/> <time value="20161201071500+0100"/> <associatedEntity classCode="PROV">
<id root="1.2.40.0.34.99.1" assigningAuthorityName="GDA Index"/> <addr>
<streetAddressLine>Taborstraße 16</streetAddressLine> <city>Wien</city> <postalCode>1020</postalCode> <country>AUT</country> </addr> <telecom use="WP" value="tel:01.47110815.123"/> <associatedPerson>
<name>
<prefix qualifier="AC">Dr.</prefix> <family>Frank</family> <given>Dieter</given> </name> </associatedPerson> <scopingOrganization>
<id root="1.2.40.0.34.99.1" assigningAuthorityName="GDA Index"/> <name>Musterklinikum Unterstadt</name> <telecom use="WP" value="tel:01.47110815"/> </scopingOrganization> </associatedEntity></participant> |
|
|---|
|
14.4.3.3 Documentation Of Service Event - Labor und Mikrobiologie
| Id | 1.2.40.0.34.6.0.11.1.48 | Gültigkeit ab | 2020‑08‑26 15:29:24 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_header_DocumentationOfServiceEventLaborUndMikrobiologie | Bezeichnung | Documentation Of Service Event - Labor und Mikrobiologie |
|---|
| Beschreibung | Dokumentation der Gesundheitsdienstleistung. Mit der Assoziation documentationOf/serviceEvent wird die eigentliche Gesundheitsdienstleistung repräsentiert, die in dem Dokument dokumentiert wird (z.B. Hämatologie, Kultureller Erregernachweis, etc.). WICHTIG: Im Zuge der Einstellung eines Labor- oder Mikrobiologiebefundes wird durch die XDS-Metadaten dargestellt, welche Sections im gegenständlichen Labor- oder Mikrobiologiebefund enthalten sind und in welcher Ausprägung (maschinenlesbar oder nicht). Daher MUSS der jeweilige section/code als serviceEvent/code als auch die section/templateId (OID Bereich: 1.2.40.0.34.6.0.11.2.X) der Section als serviceEvent/id[@root] angegeben werden. Dies MUSS - mit Ausnahme von den Sections "Brieftext" und "Abschließende Bemerkungen" - für alle Sections erfolgen. Das bedeutet, dass für jede Section ein eigenes documentationOf/serviceEvent angelegt werden muss. Zusätzlich gilt für Mikrobiologiebefunde und für Laborbefunde, die mikrobiologische Ergebnisse beinhalten, dass zusätzlich ein "documentationOf/serviceEvent" mit dem Code "18725-2 - Microbiology studies (set)" codiert werden MUSS. ↔ Hinweis zum XDS-Mapping:Da diese Informationen in die XDS-Metadaten übernommen werden, ergeben sich folgende Implikationen: - Es MÜSSEN für alle Sections entsprechende "documentationOf/serviceEvent"-Elemente angegeben werden.
- Die Zeitangaben des ersten "documentationOf/serviceEvent"-Elements werden in die Dokument-Metadaten übernommen.
- Die serviceEvents sind die einzigen medizinischen Informationen zum Dokument im XDS-Dokumentenregister. Sie können daher als Such-/Filterkriterium verwendet werden und scheinen ggf. in den Ergebnissen der Suchabfragen auf.
|
|
| Klassifikation | CDA Header Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.15 | Containment |  Time Interval Information minimal (1.0.1+20210628) Time Interval Information minimal (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.24 | Containment |  Performer - Laboratory (1.0.0+20211213) Performer - Laboratory (1.0.0+20211213) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.6.0.11.1.17 Documentation Of Service Event (2019‑03‑14 15:08:34) ref at-cda-bbr- |
|---|
| Beispiel | | Strukturbeispiel Hämatologie | <documentationOf typeCode="DOC">
<serviceEvent classCode="ACT" moodCode="EVN">
<id root="1.2.40.0.34.6.0.11.2.102"/> <code code="300" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Hämatologie"/> <effectiveTime>
<low value="20190611102209+0200"/> <high value="20190611132209+0200"/> </effectiveTime> <performer typeCode="PRF">
<!-- template 1.2.40.0.34.6.0.11.9.24 'Performer - Laboratory' -->
</performer> </serviceEvent></documentationOf> |
|
|---|
| Beispiel | | Strukturbeispiel Microbial culture finding (finding) | <documentationOf typeCode="DOC">
<serviceEvent classCode="ACT" moodCode="EVN">
<id root="1.2.40.0.34.6.0.11.2.106"/> <code code="446394004" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microbial culture finding (finding)"/> <effectiveTime>
<low value="20190611102209+0200"/> <high value="20190611132209+0200"/> </effectiveTime> <performer typeCode="PRF">
<!-- template 1.2.40.0.34.6.0.11.9.24 'Performer - Laboratory' -->
</performer> </serviceEvent></documentationOf> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | Komponente für die Gesundheitsdienstleistung.
| |  | @typeCode
|
| cs | 0 … 1 | F | DOC |  | hl7:serviceEvent
|
| | 1 … 1 | M | Die serviceEvents in den ELGA Labor- und Mikrobiologiebefunden MÜSSEN die "section/code"-Elemente als auch die "section/templateId"-Elemente wiedergeben. Diese Informationen werden über eine Mapping-Vorschrift in die XDS-Metadaten übernommen und ermöglichen einem ELGA-Teilnehmer zu erkennen, welche Sections beinhaltet sind und in welchem Codierungsgrad diese vorliegen. Daher muss für jede Section, welche medizinische Information enthält (Ausnahmen sind "Brieftext" und "Abschließende Bemerkungen"), ein documentationOf/serviceEvent codiert werden.
| |  |  | @classCode
|
| cs | 0 … 1 | F | ACT |  |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  |  | hl7:id
|
| II | 0 … 1 | C | | | | st | 0 | NP | NP/nicht anwesend | | uid | 1 … 1 | R | In das serviceEvent/id[@root] MUSS die section/templateId[@root] geschrieben werden. Im Fall von mehreren "section/templateId"-Elementen MUSS jenes gewählt werden, dessen @id-Attribut in dem OID-Bereich 1.2.40.0.34.6.0.11.2.X zu finden ist. | | | Constraint | Grundsätzlich MUSS serviceEvent/id angegeben werden. Die serviceEvent/id IST NICHT ERLAUBT für das zusätzliche serviceEvent mit dem Code "18725-2 - Microbiology studies (set)" für Mikrobiologiebefunde bzw. Laborbefunde, die mikrobiologische Ergebnisse beinhalten. | | | Schematron assert | role | error | | | | test | hl7:code[@code='18725-2'][@codeSystem='2.16.840.1.113883.6.1'] or hl7:id | | | | Meldung | serviceEvent/id MUSS angegeben werden | | | | Schematron assert | role | error | | | | test | not(hl7:code[@code='18725-2'][@codeSystem='2.16.840.1.113883.6.1'] and hl7:id) | | | | Meldung | serviceEvent/id DARF NICHT angegeben werden | |  |  | hl7:code
|
| CE | 1 … 1 | M | Code der Gesundheitsdienstleistung.
↔ Hinweis zum XDS-Mapping:
Dieses Element wird in das XDS-Attribut "eventCodeList" gemappt.
| | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.22 ELGA_ServiceEventsLabor (DYNAMIC) |
|  |  | hl7:effectiveTime
|
| IVL_TS | 1 … 1 | M | Angabe des zeitlichen Erbringungsintervalls effectiveTime mit einer Start- "low" und Endzeit "high" (verpflichtend).
Startzeitpunkt: Datum und Zeitpunkt, an dem das analysierende Labor die Anforderung vom Zuweiser in der Labor EDV erfasst hat. Falls nicht vorhanden, sind Datum und Uhrzeit des Starts des Auftrags in der Labor EDV anzugeben.
Endzeit: Datum und Zeitpunkt des Abschlusses des Auftrags, welche in der Regel mit der medizinischen Freigabe des Auftrags ident ist.
↔ Hinweis zum XDS-Mapping:
Dieses Element wird in die XDS-Attribute "serviceStartTime" und "serviceStopTime" gemappt.
Für die automatisierte Datenübernahme aus dem CDA-Dokument in die XDS-Dokumentmetadaten ist stets ein Zeitintervall anzugeben.
ACHTUNG: Die Zeitangaben der jeweils ersten Gesundheitsdienstleistung (erstes "documentationOf/serviceEvent"-Element) werden in die Dokument-Metadaten übernommen!
Die Bedeutung der Dokument-Metadaten-Elemente lautet daher wie folgt:
- serviceStartTime: Beginn des ersten documentationOf/serviceEvent-Elements
- serviceStopTime: Ende des ersten documentationOf/serviceEvent-Elements
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | |  |  | hl7:performer
|
| | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. |
|
14.4.3.4 Component Of - Encompassing Encounter with id
| Id | 1.2.40.0.34.6.0.11.1.50 ref at-cda-bbr- | Gültigkeit ab | 2023‑02‑28 10:37:28Andere Versionen mit dieser Id:  atcdabbr_header_ComponentOfEncompassingEncounterWithId vom 2021‑03‑22 15:48:13 atcdabbr_header_ComponentOfEncompassingEncounterWithId vom 2021‑03‑22 15:48:13
|
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.1+20230717 |
|---|
| Name | atcdabbr_header_ComponentOfEncompassingEncounterWithId | Bezeichnung | Component Of - Encompassing Encounter with id |
|---|
| Beschreibung |
Component Of - Encompassing Encounter gibt an, in welchem Rahmen der dokumentierte Patientenkontakt stattgefunden hat. Dokumente werden nicht notwendigerweise immer während eines Patientenkontakts erstellt, sondern ggf. auch zu einem späteren Zeitpunkt, wenn beispielsweise ein Arzt wegen eines pathologischen Laborwertes den Patienten vergeblich versucht zu erreichen und dennoch seine Verlaufsdokumentation fortführt.
Wenn die Dokumentation ein Entlass- oder Verlegungsdokument ist, muss die Information in dieser Klasse mitgegeben werden, inklusive der Dauer des Aufenthalts (hier: nicht nur stationäre Aufenthalte, sondern auch der Patientenkontakt in der Praxis eines niedergelassenen GDA beispielsweise) und der Einrichtung, wo der Patientenaufenthalt stattfand.
Verweis auf speziellen Implementierungsleitfaden:
Ob der Patientenkontakt angegeben werden muss, und welche Bedeutung dieses Element hat ergibt sich aus dem jeweiligen speziellen Implementierungsleitfaden.
|
|
| Klassifikation | CDA Header Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Assoziiert mit | | Assoziiert mit 5 Konzepte | | Id | Name | Datensatz |
|---|
| at-cda-bbr-dataelement-33 |  Patientenkontakt Patientenkontakt |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-34 |  ID ID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-37 |  Beginn des Patientenkontaktes Beginn des Patientenkontaktes |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-39 |  Art des Aufenthalts Art des Aufenthalts |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden | | at-cda-bbr-dataelement-40 |  Verantwortliche Person Verantwortliche Person |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|
|
|---|
| Benutzt | | Benutzt 3 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.15 | Containment |  Time Interval Information minimal (1.0.1+20210628) Time Interval Information minimal (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.22 | Containment |  Assigned Entity (1.0.2+20230717) Assigned Entity (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.1.8 | Inklusion |  Encounter Location (1.0.0+20210219) Encounter Location (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.1.50 Component Of - Encompassing Encounter with id (2021‑03‑22 15:48:13) ref at-cda-bbr-
Spezialisierung: Template 1.2.40.0.34.6.0.11.1.7 Component Of - Encompassing Encounter (2021‑02‑19 10:32:49) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.1.7 Component Of - Encompassing Encounter (2020‑09‑29 10:39:03) ref at-cda-bbr-
Version: Template 1.2.40.0.34.11.20013 HeaderEncompassingEncounter (2011‑12‑19) ref elgabbr- |
|---|
| Beispiel | | Strukturbeispiel mit stationärem Patientenkontakt | <componentOf typeCode="COMP">
<encompassingEncounter classCode="ENC" moodCode="EVN">
<!-- Aufenthaltszahl -->
<id root="1.2.40.0.34.99.111.1.4" extension="Az123456" assigningAuthorityName="Amadeus Spital"/> <!-- Codierung des Patientenkontakts, hier für stationär -->
<code code="IMP" displayName="Inpatient encounter" codeSystem="2.16.840.1.113883.5.4" codeSystemName="HL7:ActCode"/> <!-- Zeitraum des Patientenkontakts, mit administrativer Aufnahme am 24.12.2018 um 8:20:15 und administrativer Entlassung am 25.12.2018 um 11:30:00 -->
<effectiveTime>
<low value="20181224082015+0100"/> <high value="20181225113000+0100"/> </effectiveTime> <!-- Verantwortliche Person für den Patientenkontakt -->
<responsibleParty>
<assignedEntity>
<!-- Identifikation der Verantwortlichen Person für den Patientenkontakt-->
<!-- template 1.2.40.0.34.6.0.11.9.22 'Assigned Entity' -->
</assignedEntity> </responsibleParty> <!-- Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand -->
<location>
<healthCareFacility>
<code code="300" displayName="Allgemeine Krankenanstalt" codeSystem="1.2.40.0.34.5.2"/> <serviceProviderOrganization>
<!-- template 1.2.40.0.34.6.0.11.9.9 'Organization Compilation with name' -->
</serviceProviderOrganization> </healthCareFacility> </location> </encompassingEncounter></componentOf> |
|
|---|
| Beispiel | | Strukturbeispiel mit stationärem Patientenkontakt und unbekannter Entlassung | <componentOf typeCode="COMP">
<encompassingEncounter classCode="ENC" moodCode="EVN">
<!-- Aufenthaltszahl -->
<id root="1.2.40.0.34.99.111.1.4" extension="Az123456" assigningAuthorityName="Amadeus Spital"/> <!-- Codierung des Patientenkontakts, hier für stationär -->
<code code="IMP" displayName="Inpatient encounter" codeSystem="2.16.840.1.113883.5.4" codeSystemName="HL7:ActCode"/> <!-- Zeitraum des Patientenkontakts, mit administrativer Aufnahme am 24.12.2018 um 8:20:15 und noch nicht stattgefundener administrativer oder medizinischer Entlassung -->
<effectiveTime>
<low value="20181224082015+0100"/> <high nullFlavor="UNK"/> </effectiveTime> <!-- Verantwortliche Person für den Patientenkontakt -->
<responsibleParty>
<assignedEntity>
<!-- Identifikation der Verantwortlichen Person für den Patientenkontakt-->
<!-- template 1.2.40.0.34.6.0.11.9.22 'Assigned Entity' -->
</assignedEntity> </responsibleParty> <!-- Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand -->
<location>
<healthCareFacility>
<code code="300" displayName="Allgemeine Krankenanstalt" codeSystem="1.2.40.0.34.5.2"/> <serviceProviderOrganization>
<!-- template 1.2.40.0.34.6.0.11.9.9 'Organization Compilation with name' -->
</serviceProviderOrganization> </healthCareFacility> </location> </encompassingEncounter></componentOf> |
|
|---|
| Beispiel | | Strukturbeispiel mit ambulantem Patientenkontakt | <componentOf typeCode="COMP">
<encompassingEncounter classCode="ENC" moodCode="EVN">
<!-- Aufenthaltszahl -->
<id root="1.2.40.0.34.99.111.1.4" extension="Az123456" assigningAuthorityName="Amadeus Spital"/> <!-- Codierung des Patientenkontakts, hier für ambulant -->
<code code="AMB" displayName="ambulatory" codeSystem="2.16.840.1.113883.5.4" codeSystemName="HL7:ActCode"/> <!-- Zeitraum des Patientenkontakts, mit administrativer Aufnahme am 24.12.2018 um 8:20:15 und administrativer Entlassung am 24.12.2018 um 11:30:00 -->
<effectiveTime>
<low value="20181224082015+0100"/> <high value="20181224113000+0100"/> </effectiveTime> <!-- Verantwortliche Person für den Patientenkontakt -->
<responsibleParty>
<assignedEntity>
<!-- Identifikation der Verantwortlichen Person für den Patientenkontakt-->
<!-- template 1.2.40.0.34.6.0.11.9.22 'Assigned Entity' -->
</assignedEntity> </responsibleParty> <!-- Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand -->
<location>
<healthCareFacility>
<code code="304" displayName="Selbstständiges Ambulatorium" codeSystem="1.2.40.0.34.5.2"/> <serviceProviderOrganization>
<!-- template 1.2.40.0.34.6.0.11.9.9 'Organization Compilation with name' -->
</serviceProviderOrganization> </healthCareFacility> </location> </encompassingEncounter></componentOf> |
|
|---|
| Beispiel | | Strukturbeispiel mit ambulantem Patientenkontakt und unbekannter Entlassung | <componentOf typeCode="COMP">
<encompassingEncounter classCode="ENC" moodCode="EVN">
<!-- Aufenthaltszahl -->
<id root="1.2.40.0.34.99.111.1.4" extension="Az123456" assigningAuthorityName="Amadeus Spital"/> <!-- Codierung des Patientenkontakts, hier für ambulant -->
<code code="AMB" displayName="ambulatory" codeSystem="2.16.840.1.113883.5.4" codeSystemName="HL7:ActCode"/> <!-- Zeitraum des Patientenkontakts, mit administrativer Aufnahme am 24.12.2018 um 8:20:15 und nicht stattgefundener administrativer oder medizinischer Entlassung -->
<effectiveTime>
<low value="20181224082015+0100"/> <high nullFlavor="UNK"/> </effectiveTime> <!-- Verantwortliche Person für den Patientenkontakt -->
<responsibleParty>
<assignedEntity>
<!-- Identifikation der Verantwortlichen Person für den Patientenkontakt-->
<!-- template 1.2.40.0.34.6.0.11.9.22 'Assigned Entity' -->
</assignedEntity> </responsibleParty> <!-- Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand -->
<location>
<healthCareFacility>
<code code="304" displayName="Selbstständiges Ambulatorium" codeSystem="1.2.40.0.34.5.2"/> <serviceProviderOrganization>
<!-- template 1.2.40.0.34.6.0.11.9.9 'Organization Compilation with name' -->
</serviceProviderOrganization> </healthCareFacility> </location> </encompassingEncounter></componentOf> |
|
|---|
| Beispiel | | Strukturbeispiel mit virtuellem Patientenkontakt | <componentOf typeCode="COMP">
<encompassingEncounter classCode="ENC" moodCode="EVN">
<!-- Aufenthaltszahl -->
<id root="1.2.40.0.34.99.111.1.4" extension="Az123456" assigningAuthorityName="Amadeus Spital"/> <!-- Codierung des Patientenkontakts, hier für einen virtuellen Kontakt wie beim Telemonitoring -->
<code code="VR" displayName="virtual" codeSystem="2.16.840.1.113883.5.4" codeSystemName="HL7:ActCode"/> <!-- Zeitraum des Patientenkontakts, mit administrativer Aufnahme am 24.12.2018 um 8:20:15 und administrativer Entlassung am 31.1.2019 um 11:30:00 -->
<effectiveTime>
<low value="20181224082015+0100"/> <high value="20190131113000+0100"/> </effectiveTime> <!-- Verantwortliche Person für den Patientenkontakt -->
<responsibleParty>
<assignedEntity>
<!-- Identifikation der Verantwortlichen Person für den Patientenkontakt-->
<!-- template 1.2.40.0.34.6.0.11.9.22 'Assigned Entity' -->
</assignedEntity> </responsibleParty> <!-- Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand -->
<location>
<healthCareFacility>
<code code="300" displayName="Allgemeine Krankenanstalt" codeSystem="1.2.40.0.34.5.2"/> <serviceProviderOrganization>
<!-- template 1.2.40.0.34.6.0.11.9.9 'Organization Compilation with name' -->
</serviceProviderOrganization> </healthCareFacility> </location> </encompassingEncounter></componentOf> |
|
|---|
| Beispiel | | Strukturbeispiel mit virtuellem Patientenkontakt und unbekannter Entlassung | <componentOf typeCode="COMP">
<encompassingEncounter classCode="ENC" moodCode="EVN">
<!-- Aufenthaltszahl -->
<id root="1.2.40.0.34.99.111.1.4" extension="Az123456" assigningAuthorityName="Amadeus Spital"/> <!-- Codierung des Patientenkontakts, hier für einen virtuellen Kontakt wie beim Telemonitoring -->
<code code="VR" displayName="virtual" codeSystem="2.16.840.1.113883.5.4" codeSystemName="HL7:ActCode"/> <!-- Zeitraum des Patientenkontakts, mit administrativer Aufnahme am 24.12.2018 um 8:20:15 und nicht stattgefundener administrativer oder medizinischer Entlassung -->
<effectiveTime>
<low value="20181224082015+0100"/> <high nullFlavor="UNK"/> </effectiveTime> <!-- Verantwortliche Person für den Patientenkontakt -->
<responsibleParty>
<assignedEntity>
<!-- Identifikation der Verantwortlichen Person für den Patientenkontakt-->
<!-- template 1.2.40.0.34.6.0.11.9.22 'Assigned Entity' -->
</assignedEntity> </responsibleParty> <!-- Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand -->
<location>
<healthCareFacility>
<code code="300" displayName="Allgemeine Krankenanstalt" codeSystem="1.2.40.0.34.5.2"/> <serviceProviderOrganization>
<!-- template 1.2.40.0.34.6.0.11.9.9 'Organization Compilation with name' -->
</serviceProviderOrganization> </healthCareFacility> </location> </encompassingEncounter></componentOf> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | Komponente für den Patientenkontakt.
| | | |  | | at-cda-bbr-dataelement-33 |  Patientenkontakt Patientenkontakt |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
|  | @typeCode
|
| cs | 0 … 1 | F | COMP |  | hl7:encompassingEncounter
|
| | 1 … 1 | M | Patientenkontakt.
| |  |  | @classCode
|
| cs | 0 … 1 | F | ENC |  |  | @moodCode
|
| cs | 0 … 1 | F | EVN | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:id[not(@nullFlavor)]
- hl7:id[@nullFlavor='UNK']
| | II | 0 … 1 | | Identifikationselement zur Aufnahme der Aufenthaltszahl | | | wo [not(@nullFlavor)] | | | |  | | at-cda-bbr-dataelement-34 |  ID ID |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | st | 1 … 1 | R | Aufenthaltszahl, z.B.: Az123456
| | uid | 1 … 1 | R | OID der Liste der Aufenthaltszahlen der Organisation
| | st | 0 … 1 | | Name der Stelle, welche die ID zugewiesen hat, z.B.: "Amadeus Spital". | | II | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | |  |  | hl7:code
|
| CE | 1 … 1 | M | Codierung des Patientenkontakts.
| | | |  | | at-cda-bbr-dataelement-39 |  Art des Aufenthalts Art des Aufenthalts |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | F | HL7:ActCode | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.5 ELGA_ActEncounterCode (DYNAMIC) |
|  |  | hl7:effectiveTime
|
| IVL_TS | 1 … 1 | M |
Zeitraum des Patientenkontakts.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.15 Time Interval Information minimal (DYNAMIC) | | | |  | | at-cda-bbr-dataelement-37 |  Beginn des Patientenkontaktes Beginn des Patientenkontaktes |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | Constraint | Der Zeitraum des Patientenkontaktes MUSS die Vorgaben der speziellen Implementierungsleitfäden einhalten. Dabei gilt allgemein:
- Der Zeitraum besteht aus dem Zeitpunkt der administrativen Aufnahme in die Behandlung und dem Zeitpunkt der administrativen Entlassung aus der Behandlung.
- Der Entlassungszeitpunkt kann „unbekannt“ sein, wenn die administrative Entlassung noch nicht erfolgt ist. (nullFlavor UNK beim effectiveTime.high)
- Hinweis: Als Zeitpunkt der Aufnahme/Entlassung SOLL der Zeitpunkt der administrativen Aufnahme/Entlassung angegeben werden. Wenn der Zeitpunkt der administrativen Aufnahme/Entlassung nicht vorhanden ist, darf auch der Zeitpunkt der medizinischen Aufnahme/Entlassung angegeben werden.
|  |  | hl7:responsibleParty
|
| | 0 … 1 | R |
Komponente für die verantwortliche Person.
| | | |  | | at-cda-bbr-dataelement-40 |  Verantwortliche Person Verantwortliche Person |  Dataset A Allgemeiner Leitfaden Dataset A Allgemeiner Leitfaden |
| | | 1 … 1 | M |
Entität der verantwortlichen Person.
Grundsätzlich sind die Vorgaben für „AssignedEntity-Element (Person + Organisation)“ zu befolgen.
Beinhaltet 1.2.40.0.34.6.0.11.9.22 Assigned Entity (DYNAMIC) | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.1.8 Encounter Location (DYNAMIC)
Die Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand, MUSS verpflichtend angegeben werden (z.B.: die entlassende Krankenanstalt mit Abteilung).
|  |  | hl7:location
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | LOC | | | 1 … 1 | M | | | | cs | 0 … 1 | F | SDLOC | | CE | 1 … 1 | M |
Der Code zur Klassifizierung des GDA repräsentiert die Art der Einrichtung, in der die Tätigkeit stattfand, die zur Erzeugung des Dokuments führte. Zum Beispiel sollten Dokumente, die während eines ambulanten Falls in einem Krankenhaus entstehen, mit dem healthcareFacilityTypeCode für „Krankenhaus“ gekennzeichnet werden.
Zulässige Werte gemäß Value-Set „ELGA_HealthcareFacilityTypeCode“ Für ELGA SOLL der Code dem Eintrag "GDA Rollenname" oder, wenn der GDA Rollenname nicht verfügbar ist, der "Aggregierten Rolle" im GDA-I entsprechen.
↔ Hinweis zum XDS-Mapping: Dieses Element wird ins XDS-Attribut XDSDocumentEntry.healthcareFacilityTypeCode gemappt.
Zu berücksichtigen sind jeweils die Attribute @code, @codeSystem und @displayName.
| | | st | 1 … 1 | R | |  |  |  |  | hl7:serviceProviderOrganization
|
| | 1 … 1 | M |
Organisation, in deren Verantwortungsbereich der Patientenkontakt stattfand.
Beinhaltet 1.2.40.0.34.6.0.11.9.9 Organization Compilation with name (DYNAMIC) | |
|
14.4.4 Section Level Templates
14.4.4.1 Überweisungsgrund - codiert
14.4.4.2 Überweisungsgrund - uncodiert
| Id | 1.2.40.0.34.6.0.11.2.114 | Gültigkeit ab | 2021‑08‑11 10:39:17 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_UeberweisungsgrundUncodiert | Bezeichnung | Überweisungsgrund - uncodiert |
|---|
| Beschreibung | Der Grund für eine Gesundheitsdienstleistung. Enthält eine narrative Beschreibung des Grundes für den Auftrag (Beschreibung aus der Sicht des Gesundheitsdiensteanbieters) und/oder die eigene Beschreibung des Patienten (z.B. Hauptsymptome des Patienten). |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.114 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 4 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.36 | Containment |  Author Body (1.0.1+20230717) Author Body (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.3 | Containment |  Informant Body (1.0.1+20211213) Informant Body (1.0.1+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.19 | Containment |  Eingebettetes Objekt Entry (1.0.2+20230717) Eingebettetes Objekt Entry (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.6 Überweisungsgrund - codiert (2021‑01‑14 09:58:40) ref at-lab-
Adaptation: Template 1.2.40.0.34.6.0.11.2.47 Konsultations- oder Überweisungsgrund - kodiert (2019‑11‑05 10:43:44) ref at-cda-bbr- |
|---|
| Beispiel | | Beispiel | <section>
<templateId root="1.2.40.0.34.6.0.11.2.114"/> <code code="46239-0" codeSystem="2.16.840.1.113883.6.1" displayName="Chief complaint+Reason for visit" codeSystemName="LOINC"/> <title>Überweisungsgrund</title> <!-- CDA Level 2 -->
<text>
<!-- ... -->
<!-- Beschreibung des Grundes für den Auftrag -->
<!-- ... -->
</text></section> |
|
|---|
|
14.4.4.3 Anamnese - Labor und Mikrobiologie - codiert
14.4.4.4 Anamnese - Labor und Mikrobiologie - uncodiert
14.4.4.5 Angeforderte Untersuchungen - codiert
14.4.4.6 Angeforderte Untersuchungen - uncodiert
| Id | 1.2.40.0.34.6.0.11.2.112 | Gültigkeit ab | 2021‑08‑11 10:16:37 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_AngeforderteUntersuchungenUncodiert | Bezeichnung | Angeforderte Untersuchungen - uncodiert |
|---|
| Beschreibung | Die Section enthält die vom Auftraggeber angeforderten Untersuchungen bzw. das angeforderte Analysespektrum.
Angabe als Freitext, tabellarische Darstellung empfohlen.
Beispiele: "Kultur und Resistenz", "Pilzkultur", "Kultur auf spezielle Erreger", "MRSA-Screening" |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.112 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 3 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.36 | Containment |  Author Body (1.0.1+20230717) Author Body (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.3 | Containment |  Informant Body (1.0.1+20211213) Informant Body (1.0.1+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.15 Angeforderte Untersuchungen - codiert (2021‑01‑14 12:18:39) ref at-lab-
Adaptation: Template 1.2.40.0.34.6.0.11.2.13 Durchgeführte Maßnahmen - kodiert (2018‑12‑04 10:21:16) ref at-cda-bbr- |
|---|
| Beispiel | | Beispiel | <section>
<templateId root="1.2.40.0.34.6.0.11.2.112"/> <code code="400999005" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Procedure requested (situation)"/> <title>Angeforderte Untersuchungen</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Angeforderte Untersuchungen</th> </tr> </thead> <tbody>
<tr ID="req-1">
<td>MRSA-Screening</td> </tr> <!-- weitere angeforderte Untersuchungen -->
</tbody> </table> </text></section> |
|
|---|
|
14.4.4.7 Probeninformation (Specimen Section)
| Id | 1.2.40.0.34.6.0.11.2.93 | Gültigkeit ab | 2021‑01‑14 14:01:18 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_ProbeninformationSpecimenSection | Bezeichnung | Probeninformation (Specimen Section) |
|---|
| Beschreibung | Die Dokumentation des Untersuchungsmaterials kann auf zwei Arten erfolgen: - Enthält ein Befund nur einen Befundbereich ("Laboratory Specialty Section"), so kann die Codierung innerhalb der einen Section erfolgen.
ODER - Bei Verwendung von mehreren Befundbereichen in einem Befund kann es zu Überschneidungen der Untersuchungsmaterialien
kommen (ein spezielles Untersuchungsmaterial kann in zwei Befundbereichen analysiert werden). Die CDA Level 3 Codierung eines Untersuchungsmaterials darf jedoch nur einmal im gesamten Befund erfolgen. Daher sind die Informationen zu den Untersuchungsmaterialien in einer eigenen, führenden "Probeninformation (Specimen Section)" zu codieren.
WICHTIG: Derzeit werden Mikrobiologiebefunde mit mehreren Untersuchungsmaterialien NICHT unterstützt. Das bedeutet, dass in einem Mikrobiologiebefund nur genau ein Untersuchungsmaterial dokumentiert werden darf. Falls mehrere Untersuchungsmaterialien zu einem Auftrag eingesendet wurden, müssen diese auf mehrere Befunde aufgeteilt werden! Die Information kann in der Section "Befundbewertung" festgehalten werden. Der Inhalt dieser Section enthält sämtliche Informationen über das zu befundende Untersuchungsmaterial. Die Darstellung in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Bemerkung Labor" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich eine Bemerkung vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| Matrial-ID | [O] | Identifikation des Untersuchungsmaterials. | | Probenentnahme | [R] | Zeitpunkt oder Zeitintervall der Entnahme des Untersuchungsmaterials. Wert muss nicht angegeben werden bzw. darf "unbekannt" sein. Format: dd.MM.yyyy
hh24:mi | | Untersuchtes Material | [R] | Enthält Materialart [R] und Entnahmeort [O]. | | Probenentnahme durch | [O] | Für Entnahme des Untersuchungsmaterials zuständige Person und gegebenenfalls Organisation. | | Probeneingang | [R] | Zeitpunkt des Eingangs des Untersuchungsmaterials im Labor.
Format: dd.MM.yyyy hh24:mi | | Bemerkung Labor | [R] | Allfällige Bemerkungen zur Qualität des Untersuchungsmaterials. |
|
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.93 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.160 | Containment |  Probeninformation (Specimen Entry) (1.0.0+20211213) Probeninformation (Specimen Entry) (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.11.4.2.1 Spezimen-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Vollständiger 'section/text' | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.93"/> <code code="10" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Probeninformation"/> <title>Probeninformation</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th styleCode="xELGA_colw:15">Material-ID</th> <th styleCode="xELGA_colw:10">Probenentnahme</th> <th styleCode="xELGA_colw:14">Untersuchtes Material</th> <th styleCode="xELGA_colw:17">Probenentnahme durch</th> <th styleCode="xELGA_colw:10">Probeneingang</th> <th styleCode="xELGA_colw:25">Bemerkung Labor</th> </tr> </thead> <tbody>
<tr ID="SPEC-1-1">
<td>PL-081201-02</td> <td>01.12.2012 06:34</td> <td>Plasma, Linke Ellenbeuge</td> <td>Dr. Humpel, Amadeus Spital</td> <td>01.12.2012 08:15</td> <td ID="SpecimenComment01">leicht hämolytisch</td> </tr> <tr ID="SPEC-2-1">
<td>WD-081201-01</td> <td>01.12.2012 06:34</td> <td>Wunddrainage, rechter Oberarm</td> <td>Dr. Humpel, Amadeus Spital</td> <td>01.12.2012
08:15</td> <td/> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<!-- include 1.2.40.0.34.6.0.11.3.160 'Probeninformation (Specimen Entry)' -->
</entry></section> |
|
|---|
| Beispiel | | Minimaler 'section/text' | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.93"/> <code code="10" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Probeninformation"/> <title>Probeninformation</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Probenentnahme</th> <th>Untersuchtes Material</th> <th>Probeneingang</th> <th>Bemerkung Labor</th> </tr> </thead> <tbody>
<tr ID="SPEC-1-1">
<td>01.12.2012 06:34</td> <td>Plasma, Linke Ellenbeuge</td> <td>01.12.2012 08:15</td> <td ID="SpecimenComment01">leicht hämolytisch</td> </tr> <tr ID="SPEC-2-1">
<td>01.12.2012 06:34</td> <td>Wunddrainage,
rechter Oberarm</td> <td>01.12.2012 08:15</td> <td/> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<!-- include 1.2.40.0.34.6.0.11.3.160 'Probeninformation (Specimen Entry)' -->
</entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | Probeninformation (Specimen Section) | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Probeninformation (Specimen Section)
| |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.93 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section | |  | hl7:code
|
| CE | 1 … 1 | M | | |  |  | @codeSystemName
|
| st | 0 … 1 | F | ELGA_LaborparameterErgaenzung |  |  | @code
|
| CONF | 1 … 1 | F | 10 |  |  | @codeSystem
|
| 1 … 1 | F | 1.2.40.0.34.5.11 (ELGA_LaborparameterErgaenzung) |  |  | @displayName
|
| 1 … 1 | F | Probeninformation |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Probeninformation" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Menschenlesbare Information über das Material in tabellarischer Form. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.160 Probeninformation (Specimen Entry) (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.8 Laboratory Specialty Section
| Id | 1.2.40.0.34.6.0.11.2.102 | Gültigkeit ab | 2021‑01‑21 11:16:21 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySection | Bezeichnung | Laboratory Specialty Section |
|---|
| Beschreibung | Die Section "Laboratory Specialty Section" entspricht den Befundbereichen basierend auf dem Value Set "ELGA_Laborstruktur", wobei für Befundbereiche NUR Einträge des Level 1 verwendet werden dürfen. Bei der Verwendung der Befundbereiche ist die Reihenfolge gemäß Value Set verpflichtend einzuhalten. Achtung: Aus dem Value Set "ELGA_Laborstruktur" werden der Code "10 - Probeninformation" durch die Section "Probeninformation (Specimen Section)" und der Code "20 - Befundbewertung" durch die Section "Befundbewertung" abgedeckt und dürfen somit im Rahmen dieses Templates NICHT VERWENDET werden. Darstellung der Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. "section/text" darf keine medizinisch relevanten Inhalte enthalten, die nicht aus den CDA Level 3 codierten Daten abgeleitet werden können. Die CDA Level 3 codierten Informationen sind über das Template "Laboratory Report Data Processing Entry" abzubilden, welches die Grundlage für die Strukturierung (Abschnitte, Formatierung, etc.) von "section/text" bildet. Die Darstellung der Analyseergebnisse in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Einheit" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich eine Einheit vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| Analyse | [R] | Bezeichung der Analyse (MUSS dem "Begriff"/"displayName" aus dem Value Set
"ELGA_Laborparameter" entsprechen). | | Ergebnis | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der Analyse. | | Einheit | [R] | Einheit; falls zutreffend, ist der UCUM printName anzugeben. | | Referenzbereich | [O] | Der für die gegebenen Rahmenbedinungen (Patient,
Analyse, Untersuchungsmaterial, etc.) passende Referenzbereich ist anzugeben. | | Interpretation | [O] | Codierte Angabe der Interpretation des Ergebnisses (siehe unten). | | Externes Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. |
Anmerkungen zur Tabelle: - Spezialuntersuchungen, die nicht in das angegebene Schema passen (z.B Molekulare Diagnostik, Allergiediagnostik etc.), können bei Bedarf auch anders dargestellt werden. Ensprechende Beispieldokumente stehen zur Verfügung.
- Für die Spalte "Ergebnis" und "Referenzbereich" wird EMPFOHLEN, bei Dezimalzahlen einen Punkt als Dezimaltrennzeichen zu
verwenden – gleich wie im maschinenlesbaren Teil.
Interpretation der Ergebnisse In Laborbefunden ist es üblich, eine codierte Bewertung zu jedem Ergebnis anzugeben. Basierend auf dem Auszug aus dem Value Set "ELGA_ObservationInterpretation" stellt die folgende Tabelle dar, wie eine Darstellung in CDA Level 2, in Abhängigkeit von den CDA Level 3 codierten Interpretationen, aussehen kann. | Darstellung CDA Level 2 | Codierung CDA Level 3 | Beschreibung |
|---|
| Befundinterpretation für numerische Ergebnisse |
|---|
| ++ | HH | Oberhalb des Referenzbereiches und über einer oberen Warngrenze | | + | H | Oberhalb des
Referenzbereiches | | N | Normal (innerhalb des Referenzbereiches) | | - | L | Unterhalb des Referenzbereiches | | -- | LL | Unterhalb des Referenzbereiches und unter einer unteren Warngrenze | | Befundinterpretation für nicht numerische
Ergebnisse |
|---|
| N | Normal (innerhalb des Referenzbereiches) | | * | A | Abnormal | | ** | AA | Abnormal Warngrenze |
Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben (siehe Beispiel). |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.102 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.25 | Containment |  Laboratory Report Data Processing Entry (1.0.1) Laboratory Report Data Processing Entry (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.102"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="300" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Hämatologie"/> <title>Hämatologie</title> <!-- CDA Level 2 -->
<text>
<!-- Befundgruppe "Blutbild" -->
<paragraph styleCode="xELGA_h3">Blutbild</paragraph> <table>
<thead>
<tr>
<th>Analyse</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereiche</th> <th>Interpretation</th> </tr> </thead> <tbody>
<!-- auffälliges/pathologisches Ergebnis gekennzeichnet durch styleCode -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>+</td> </tr> <tr ID="OBS-1-2">
<td>Thrombozyten</td> <td>165</td> <td>10^9/L</td> <td ID="OBSREF-1-2">150-360</td> <td/> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<!-- template 1.2.40.0.34.6.0.11.3.25 'Laboratory Report Data Processing Entry' -->
</entry></section> |
|
|---|
| Beispiel | | Allergiediagnostik | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.102"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="1800" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Allergiediagnostik"/> <title>Allergiediagnostik</title> <!-- CDA Level 2 -->
<text>
<!-- Befundgruppe "Globalmarker" -->
<paragraph styleCode="xELGA_h3">Globalmarker</paragraph> <table>
<thead>
<tr>
<th styleCode="xELGA_colw:70">Analyse</th> <th>Ergebnis</th> <th>Interpretation</th> </tr> </thead> <tfoot>
<tr>
<td>
Die "erweiterten Analyseinformationen" wurden direkt unter die Analysenbezeichnungen geschrieben. <br/> (Hier kann sonst ein Kommentar zu den Globalmarkern
stehen.) </td> </tr> </tfoot> <tbody>
<tr ID="OBS-1-1">
<td>
sx1 Inhalatives Screening <br/> <content styleCode="italics">Lieschgras, Roggen, Birke, Beifuß, Dermatophagoides pteronyssinus, Katzenschuppen, Hundeschuppen, Cladosporium herbarum</content> </td> <td>negativ</td> <td/> </tr> <tr ID="OBS-1-2" styleCode="xELGA_red">
<td>
mx1 Schimmelpilzemix 1 <br/> <content styleCode="italics">Alternaria alternata, Aspergillus fumigatus, Cladosporium herbarum, Penicillium notatum</content> </td> <td>positiv</td> <td>A</td> </tr> </tbody> </table> <!-- Befundgruppe "Inhalationsallergene IgE" -->
<paragraph styleCode="xELGA_h3">Inhalationsallergene IgE</paragraph> <table>
<thead>
<tr>
<th styleCode="xELGA_colw:40">Analyse</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereich</th> <th>Interpretation</th> </tr> </thead> <tfoot>
<tr>
<td>Drei Optionen der Befunddarstellung der Allergiediagnostik: 1) quantitativ, 2) mit
RAST-Klasse und 3) kombiniert (quantitativ + RAST-Interpretation) </td> </tr> </tfoot> <tbody>
<tr ID="OBS-2-1">
<td>Penicillum notatum (Pinselschimmel)</td> <td><0.35</td> <td>kU/L</td> <td><0.35</td> <td/> </tr> <tr ID="OBS-2-2">
<td>Cladosporium herbarum IgE RAST</td> <td>0</td> <td>RAST</td> <td/> <td/> </tr> <tr ID="OBS-2-3" styleCode="xELGA_red">
<td>Alternaria
alternata (Alternaria tenuis) IgE</td> <td>5.18</td> <td>kU/L</td> <td><0.35</td> <td>RAST 3</td> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<!-- template 1.2.40.0.34.6.0.11.3.25 'Laboratory Report Data Processing Entry' -->
</entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.102 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | | | | Constraint | Aus dem Value Set dürfen NUR Einträge des Level 1 verwendet werden. Bei der Verwendung der Befundbereiche ist die Reihenfolge gemäß Value Set verpflichtend einzuhalten.
Achtung: Der Code "10 - Probeninformation" wird durch die Section "Probeninformation (Specimen Section)" und der Code "20 - Befundbewertung" wird durch die Section "Befundbewertung" abgedeckt und dürfen somit im Rahmen dieses Templates NICHT VERWENDET werden. | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.47 ELGA_Laborstruktur (DYNAMIC) |
|  | hl7:title
|
| ST | 1 … 1 | M | Der Titel MUSS dem displayName des "code"-Elements entsprechen. | |  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.9 Laboratory Specialty Section (Mikroskopie) - codiert
| Id | 1.2.40.0.34.6.0.11.2.105 | Gültigkeit ab | 2021‑04‑06 08:10:32 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySectionMikroskopieCodiert | Bezeichnung | Laboratory Specialty Section (Mikroskopie) - codiert |
|---|
| Beschreibung | Die Section "Laboratory Specialty Section (Mikroskopie)" entspricht jenem Befundbereich eines Mikrobiologiebefundes, in dem alle Mikroskopie-Ergebnisse dokumentiert werden. Jedes Ergebnis wird direkt in einer separaten "Laboratory Observation" codiert werden. Darstellung der Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. "section/text" darf keine medizinisch relevanten Inhalte enthalten, die nicht aus den CDA Level 3 codierten Daten abgeleitet werden können. Die CDA Level 3 codierten Informationen sind über das Template "Laboratory Report Data Processing Entry" abzubilden, welches die Grundlage für die Strukturierung (Abschnitte, Formatierung, etc.) von "section/text" bildet. Die Darstellung der Mikroskopieergebnisse in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Ergebnis" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich ein Ergebnis vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| Eigenschaft | [R] | Bezeichung der Analyse (MUSS dem "Begriff"/"displayName" aus dem Value Set
"ELGA_Laborparameter" entsprechen). | | Ergebnis | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der Analyse | | Einheit | [O] | Einheit; falls zutreffend, ist der UCUM printName anzugeben. | | Externes Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. |
Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben. |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.105 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.25 | Containment |  Laboratory Report Data Processing Entry (1.0.1) Laboratory Report Data Processing Entry (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (2021‑01‑21 11:16:21) ref at-lab-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.105"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="395538009" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microscopic specimen observation (finding)"/> <title>Mikroskopie</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Eigenschaft</th> <th>Ergebnis</th> <th>Einheit</th> </tr> </thead> <tbody>
<tr ID="OBS-1-1">
<td>Epithelzellen</td> <td ID="OBS-1-1-result">vereinzelt</td> <td/> </tr> <tr ID="OBS-1-2">
<td>grampositive Stäbchen</td> <td ID="OBS-1-2-result">spärlich</td> <td/> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <templateId root="1.3.6.1.4.1.19376.1.3.1"/> <act classCode="ACT" moodCode="EVN">
<code code="395538009" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microscopic specimen observation (finding)"/> <statusCode code="completed"/> <!-- Codierung der "Epithelzellen" -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="V00681-9" codeSystem="1.2.40.0.34.5.11" displayName="Epithelzellen in unspezifiziertem Material, lichtmikroskopisch"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="89292003" codeSystem="2.16.840.1.113883.6.96" displayName="Rare (qualifier value)">
<originalText>
<reference value="#OBS-1-1-result"/> </originalText> </value> </observation> </entryRelationship> <!-- Codierung der "grampositiven Stäbchen" -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="V00700-7" codeSystem="1.2.40.0.34.5.11" displayName="grampositive Stäbchen in unspezifiziertem Material, lichtmikroskopisch"/> <text>
<reference value="#OBS-1-2"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="57176003" codeSystem="2.16.840.1.113883.6.96" displayName="Few (qualifier value)">
<originalText>
<reference value="#OBS-1-2-result"/> </originalText> </value> </observation> </entryRelationship> </act> </entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section (Mikroskopie) - codiert | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.105 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 395538009 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Microscopic specimen observation (finding) |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Mikroskopie" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.10 Laboratory Specialty Section (Mikroskopie) - uncodiert
| Id | 1.2.40.0.34.6.0.11.2.113 | Gültigkeit ab | 2021‑08‑11 10:26:02 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySectionMikroskopieUncodiert | Bezeichnung | Laboratory Specialty Section (Mikroskopie) - uncodiert |
|---|
| Beschreibung | Die Section "Laboratory Specialty Section (Mikroskopie)" entspricht jenem Befundbereich eines Mikrobiologiebefundes, in dem alle Mikroskopie-Ergebnisse dokumentiert werden. Darstellung der Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. Die Darstellung der Mikroskopieergebnisse in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Optionalität ist wie folgt zu verstehen: z.B. ist das "Ergebnis" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich ein Ergebnis vorhanden ist. <TBODY>| Spaltenname | Optionalität | Beschreibung |
|---|
| Eigenschaft | [R] | Bezeichung der Analyse (MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Laborparameter"
entsprechen). | | Ergebnis | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der Analyse | | Einheit | [O] | Einheit; falls zutreffend, ist der UCUM printName anzugeben. | | Externes Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. | </TBODY> Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben. |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.113 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.105 Laboratory Specialty Section (Mikroskopie) - codiert (2021‑04‑06 08:10:32) ref at-lab-
Spezialisierung: Template 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (2021‑01‑21 11:16:21) ref at-lab-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.113"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="395538009" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microscopic specimen observation (finding)"/> <title>Mikroskopie</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Eigenschaft</th> <th>Ergebnis</th> <th>Einheit</th> </tr> </thead> <tbody>
<tr ID="OBS-1-1">
<td>Epithelzellen</td> <td ID="OBS-1-1-result">vereinzelt</td> <td/> </tr> <tr ID="OBS-1-2">
<td>grampositive Stäbchen</td> <td ID="OBS-1-2-result">spärlich</td> <td/> </tr> </tbody> </table> </text></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section (Mikroskopie) - uncodiert | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.113 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 395538009 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Microscopic specimen observation (finding) |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Mikroskopie" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.11 Laboratory Specialty Section (Kultureller Erregernachweis)
| Id | 1.2.40.0.34.6.0.11.2.106 | Gültigkeit ab | 2021‑04‑06 08:46:53 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySectionKulturellerErregernachweis | Bezeichnung | Laboratory Specialty Section (Kultureller Erregernachweis) |
|---|
| Beschreibung | Die "Laboratory Specialty Section (Kultureller Erregernachweis)" entspricht jenem Befundbereich eines Mikrobiologiebefundes, in dem alle kulturellen Erregernachweise inklusive möglicher Antibiogramme dokumentiert werden. Jeder ermittelte Erreger und dessen Antibiogramm wird in einem separaten "Laboratory Isolate Organzier" codiert. Darstellung der
Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. "section/text" darf keine medizinisch relevanten Inhalte enthalten, die nicht aus den CDA Level 3 codierten Daten abgeleitet werden können. Die CDA Level 3 codierten Informationen sind über das Template "Laboratory Report Data Processing Entry" abzubilden, welches die Grundlage für die Strukturierung (Abschnitte, Formatierung, etc.) von "section/text" bildet. Darstellung der kulturellen Ergebnisse Die Darstellung der kulturellen Erregernachweise in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Methode" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich eine Methode vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| [O] | Fortlaufende Nummerierung der Erreger, um die Referenzierung in der Antibiogrammtabelle zu erleichtern. | | Erreger | [R] | Bezeichung des Erregers (MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Mikroorganismen_VS"
entsprechen). | | Methode | [R] | Angabe der durchgeführten Methode, mit der der Erreger nachgewiesen wurde (MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Laborparameter" entsprechen). | | Ergebnis / Keimzahl | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der
Analyse. | | Externes Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. |
Darstellung der Antibiogramme Die Darstellung der Antibiogramme in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. - Die
erste Spalte "Wirkstoff" der Tabelle erhält für jeden getesteten Wirkstoff eine Zeile. Der dargestellte Name MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Antibiogramm_VS" entsprechen.
- Für jeden Erreger, für den ein Antibiogramm vorliegt, wird eine zusätzliche Spalte erstellt. Die Spaltenüberschrift enthält den Erregernamen. Sollte die Matrix bei zu
vielen Erregern zu unübersichtlich werden, wird empfohlen, die Erreger in der Tabelle des Erregernachweises zu nummerieren und in der Tabelle des Antibiogramms lediglich die Nummer des getesteten Keims als Spaltenüberschrift anzugeben.
- Am Schnittpunkt von Erreger (Spalte) und Wirkstoff (Zeile) wird jeweils die Resistenz (R, S, I) und/oder die minimale
Hemmkonzentration (MHK) festgehalten. Es ist auch möglich, dass weder Resistenz noch MHK für eine Erreger-Wirkstoff-Kombination vorhanden ist. In diesem Fall bleibt die Zelle der Tabelle leer.
Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben. |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.106 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.25 | Containment |  Laboratory Report Data Processing Entry (1.0.1) Laboratory Report Data Processing Entry (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (2021‑01‑21 11:16:21) ref at-lab-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.105"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="446394004" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microbial culture finding (finding)"/> <title>Kultureller Erregernachweis</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Erreger</th> <th>Methode</th> <th>Ergebnis / Keimzahl</th> </tr> </thead> <tbody>
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">Staphylococcus epidermidis</td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">nachgewiesen</td> </tr> <tr ID="OBS-MIBI-2">
<!-- Dokumentation eines weiteren Erregers -->
</tr> </tbody> </table> <paragraph styleCode="xELGA_h3">Antibiogramm</paragraph> <table>
<thead>
<tr>
<th>Wirkstoff</th> <th>Staphylococcus epidermidis</th> <th>
<!-- Dokumentation eines weiteren Erregers -->
</th> </tr> </thead> <tfoot>
<tr>
<td>S = sensibel bei Standarddosierung, I = sensibel bei erhöhter Exposition, R =
resistent, [] minimale Hemmkonzentration in mg/L</td> </tr> </tfoot> <tbody>
<tr>
<td ID="OBS-AB-1">Penicillin</td> <td ID="OBS-AB-1-1">R</td> <td ID="OBS-AB-1-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> <tr>
<td ID="OBS-AB-2">Oxacillin</td> <td ID="OBS-AB-2-1">S</td> <td ID="OBS-AB-2-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> <tr>
<td ID="OBS-AB-3">Erythromycin</td> <td ID="OBS-AB-3-1">S</td> <td ID="OBS-AB-3-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> <tr>
<td ID="OBS-AB-4">Clindamycin</td> <td ID="OBS-AB-4-1">S</td> <td ID="OBS-AB-4-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <act classCode="ACT" moodCode="EVN">
<code code="446394004" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microbial culture finding (finding)"/> <statusCode code="completed"/> <entryRelationship typeCode="COMP">
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="60875001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Staphylococcus epidermidis">
<originalText>
<reference value="#OBS-MIBI-1-1"/> </originalText> </code> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="11475-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Microorganism identified in Specimen by Culture">
<originalText>
<reference value="#OBS-MIBI-1-2"/> </originalText> </code> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" displayName="Detected (qualifier value)">
<originalText>
<reference value="#OBS-MIBI-1-3"/> </originalText> </value> </observation> </component> <!-- Antibiogramm -->
<component typeCode="COMP">
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="365705006" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Finding of antimicrobial susceptibility (finding)"/> <statusCode code="completed"/> <component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18964-7" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Penicillin [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" nullFlavor="UNK"/> <interpretationCode code="R" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Resistant"/> </observation> </component> <!-- ... -->
<!-- Codierung weiterer Antibiogrammergebnisse für "Staphylococcus epidermidis" -->
<!-- ... -->
</organizer> </component> </organizer> <!-- ... -->
<!-- Weitere "Laboratory Isolate Organizer" für weitere Erreger -->
<!-- ... -->
</entryRelationship> </act> </entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section (Kultureller Erregernachweis) | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.106 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 446394004 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Microbial culture finding (finding) |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Kultureller Erregernachweis" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.12 Laboratory Specialty Section (Molekularer Erregernachweis)
| Id | 1.2.40.0.34.6.0.11.2.107 | Gültigkeit ab | 2021‑04‑06 08:56:54 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySectionMolekularerErregernachweis | Bezeichnung | Laboratory Specialty Section (Molekularer Erregernachweis) |
|---|
| Beschreibung | Die "Laboratory Specialty Section (Molekularer Erregernachweis)" entspricht jenem Befundbereich eines Mikrobiologiebefundes, in dem alle molekularen Erregernachweise dokumentiert werden. Jeder ermittelte Erreger wird direkt in einer separaten "Laboratory Observation" codiert. Darstellung der Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. "section/text" darf keine medizinisch relevanten Inhalte enthalten, die nicht aus den CDA Level 3 codierten Daten abgeleitet werden können. Die CDA Level 3 codierten Informationen sind über das Template "Laboratory Report Data Processing Entry" abzubilden, welches die Grundlage für die Strukturierung (Abschnitte, Formatierung, etc.) von "section/text" bildet. Die Darstellung der molekularen Erregernachweise in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Ergebnis" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich ein Ergebnis vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| Analyse / Erreger / Methode | [R] | Bezeichung der Analyse /
Erreger / Methode (MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Laborparameter" entsprechen). | | Ergebnis | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der Analyse / Erreger / Methode. | | Einheit | [O] | Einheit; falls zutreffend, ist der UCUM printName anzugeben. | | Referenzbereich / Nachweisgrenze / Linearitätsbereich | [O] | Der für die gegebenen Rahmenbedinungen (Patient, Analyse, Untersuchungsmaterial, etc.) passende Referenzbereich. | | Interpretation | [O] | Codierte Angabe der Interpretation des Ergebnisses (siehe unten). | | Externes
Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. |
Anmerkungen zur Tabelle: - Für die Spalte "Ergebnis" und "Referenzbereich" wird EMPFOHLEN, bei Dezimalzahlen einen Punkt als Dezimaltrennzeichen zu verwenden – gleich wie im maschinenlesbaren
Teil.
Interpretation der Ergebnisse In Laborbefunden ist es üblich, eine codierte Bewertung zu jedem Ergebnis anzugeben. Basierend auf dem Auszug aus dem Value Set "ELGA_ObservationInterpretation" stellt die folgende Tabelle dar, wie eine Darstellung in CDA Level 2, in Abhängigkeit von den CDA Level 3 codierten Interpretationen, aussehen kann. | Darstellung CDA Level 2 | Codierung CDA Level 3 | Beschreibung |
|---|
| Befundinterpretation für numerische Ergebnisse |
|---|
| ++ | HH | Oberhalb des Referenzbereiches und über einer oberen Warngrenze | | + | H | Oberhalb des
Referenzbereiches | | N | Normal (innerhalb des Referenzbereiches) | | - | L | Unterhalb des Referenzbereiches | | -- | LL | Unterhalb des Referenzbereiches und unter einer unteren Warngrenze | | Befundinterpretation für nicht numerische
Ergebnisse |
|---|
| N | Normal (innerhalb des Referenzbereiches) | | * | A | Abnormal | | ** | AA | Abnormal Warngrenze |
Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben (siehe Beispiel). |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.107 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.25 | Containment |  Laboratory Report Data Processing Entry (1.0.1) Laboratory Report Data Processing Entry (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (2021‑01‑21 11:16:21) ref at-lab-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.107"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="108262000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Molecular biology method (procedure)"/> <title>Molekularer Erregernachweis</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Analyse / Erreger / Methode</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereich / Nachweisgrenze / Linearitätsbereich</th> <th>Interpretation</th> </tr> </thead> <tbody>
<!-- auffälliges/pathologisches Ergebnis gekennzeichnet durch styleCode -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Norovirus-RNA</td> <td>nachgewiesen</td> <td/> <td ID="OBSREF-1-1"/> <td/> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <act classCode="ACT" moodCode="EVN">
<code code="108262000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Molecular biology method (procedure)"/> <statusCode code="completed"/> <!-- Codierung von "Norovirus-RNA" als "Laboratory Observation" -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="56748-7" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Norovirus RNA [Presence] in Unspecified specimen by Probe and target amplification method"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" displayName="Detected (qualifier value)"/> </observation> </entryRelationship> </act> </entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section (Molekularer Erregernachweis) | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.107 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 108262000 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Molecular biology method (procedure) |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Molekularer Erregernachweis" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.13 Laboratory Specialty Section (Infektionsserologie)
| Id | 1.2.40.0.34.6.0.11.2.108 | Gültigkeit ab | 2021‑04‑06 09:46:38 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySectionInfektionsserologie | Bezeichnung | Laboratory Specialty Section (Infektionsserologie) |
|---|
| Beschreibung | Die "Laboratory Specialty Section (Infektionsserologie)" entspricht jenem Befundbereich eines Mikrobiologiebefundes, in dem alle Ergebnisse der Infektionsserologie dokumentiert werden. Jedes ermittelte Ergebnis wird direkt in einer separaten "Laboratory Observation" codiert. Darstellung der Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. "section/text" darf keine medizinisch relevanten Inhalte enthalten, die nicht aus den CDA Level 3 codierten Daten abgeleitet werden können. Die CDA Level 3 codierten Informationen sind über das Template "Laboratory Report Data Processing Entry" abzubilden, welches die Grundlage für die Strukturierung (Abschnitte, Formatierung, etc.) von "section/text" bildet. Die Darstellung der Infektionsserologie in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Ergebnis" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich ein Ergebnis vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| Analyse / Erreger / Methode | [R] | Bezeichung der Analyse / Erreger /
Methode (MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Laborparameter" entsprechen). | | Ergebnis | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der Analyse / Erreger / Methode. | | Einheit | [O] | Einheit; falls zutreffend, ist der UCUM printName anzugeben. | | Referenzbereich / Nachweisgrenze / Linearitätsbereich | [O] | Der für die gegebenen Rahmenbedinungen (Patient, Analyse, Untersuchungsmaterial, etc.) passende Referenzbereich. | | Interpretation | [O] | Codierte Angabe der Interpretation des Ergebnisses (siehe unten). | | Externes
Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. |
Anmerkungen zur Tabelle: - Für die Spalte "Ergebnis" und "Referenzbereich" wird EMPFOHLEN, bei Dezimalzahlen einen Punkt als Dezimaltrennzeichen zu verwenden – gleich wie im maschinenlesbaren
Teil.
Interpretation der Ergebnisse In Laborbefunden ist es üblich, eine codierte Bewertung zu jedem Ergebnis anzugeben. Basierend auf dem Auszug aus dem Value Set "ELGA_ObservationInterpretation" stellt die folgende Tabelle dar, wie eine Darstellung in CDA Level 2, in Abhängigkeit von den CDA Level 3 codierten Interpretationen, aussehen kann. | Darstellung CDA Level 2 | Codierung CDA Level 3 | Beschreibung |
|---|
| Befundinterpretation für numerische Ergebnisse |
|---|
| ++ | HH | Oberhalb des Referenzbereiches und über einer oberen Warngrenze | | + | H | Oberhalb des
Referenzbereiches | | N | Normal (innerhalb des Referenzbereiches) | | - | L | Unterhalb des Referenzbereiches | | -- | LL | Unterhalb des Referenzbereiches und unter einer unteren Warngrenze | | Befundinterpretation für nicht numerische
Ergebnisse |
|---|
| N | Normal (innerhalb des Referenzbereiches) | | * | A | Abnormal | | ** | AA | Abnormal Warngrenze |
Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben. |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.108 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.25 | Containment |  Laboratory Report Data Processing Entry (1.0.1) Laboratory Report Data Processing Entry (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (2021‑01‑21 11:16:21) ref at-lab-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.108"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="722143004" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Infectious disease diagnostic study note (record artifact)"/> <title>Infektionsserologie</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Analyse / Erreger / Methode</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereich / Nachweisgrenze / Linearitätsbereich</th> <th>Interpretation</th> </tr> </thead> <tbody>
<tr ID="OBS-1-1">
<td>Toxoplasma gondii IgG</td> <td>nicht nachgewiesen</td> <td/> <td ID="OBSREF-1-1"/> <td/> </tr> <tr ID="OBS-1-2">
<td>anti-HBs</td> <td>0.16</td> <td>mIU/ml</td> <td ID="OBSREF-1-2"><10.00</td> <td/> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <act classCode="ACT" moodCode="EVN">
<code code="722143004" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Infectious disease diagnostic study note (record artifact)"/> <statusCode code="completed"/> <!-- Codierung von "Toxoplasma gondii IgG" als "Laboratory Observation" -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="22580-5" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Toxoplasma G. IgG AK ql."/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260415000" codeSystem="2.16.840.1.113883.6.96" displayName="Not detected (qualifier value)"/> </observation> </entryRelationship> <!-- Codierung von "anti-HBs" als "Laboratory Observation" -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="16935-9" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="HBV s-AK qn."/> <text>
<reference value="#OBS-2-1"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" value="0.16" unit="[mIU/ml]"/> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Normal"/> <referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-2-1"/> </text> <value xsi:type="IVL_PQ">
<low nullFlavor="NINF"/> <high value="10.0"/> </value> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Normal"/> </observationRange> </referenceRange> </observation> </entryRelationship> </act> </entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section (Infektionsserologie) | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.108 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 722143004 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Infectious disease diagnostic study note (record artifact) |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Infektionsserologie" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.14 Laboratory Specialty Section (Weitere Analysen)
| Id | 1.2.40.0.34.6.0.11.2.110 | Gültigkeit ab | 2021‑01‑22 11:11:58 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_section_LaboratorySpecialtySectionWeitereAnalysen | Bezeichnung | Laboratory Specialty Section (Weitere Analysen) |
|---|
| Beschreibung | Die "Laboratory Specialty Section (Weitere Analysen)" dient als zusätzlicher Befundbereich eines Mikrobiologiebefundes, in dem Analysen dokumentiert werden können, die keiner der anderen Laboratory Specialty Section (Mikroskopie, Kultureller Erregernachweis, Molekularer Erregernachweis, Infektionsserologie) zugeordnet werden können. Jede Analyse wird direkt in einer separaten "Laboratory Observation" codiert. Darstellung der Ergebnisse "section/text" enthält den narrativen Text, der der CDA Level 2 Darstellung der medizinischen Inhalte entspricht. "section/text" darf keine medizinisch relevanten Inhalte enthalten, die nicht aus den CDA Level 3 codierten Daten abgeleitet werden können. Die CDA Level 3 codierten Informationen sind über das Template "Laboratory Report Data Processing Entry" abzubilden, welches die Grundlage für die Strukturierung (Abschnitte, Formatierung, etc.) von "section/text" bildet. Die Darstellung der Analyseergebnisse in "section/text" hat tabellarisch zu erfolgen, wobei die Tabelle wie folgt aufgebaut sein sollte. Die Optionalität in dieser Tabelle bezieht sich auf die Darstellung des jeweiligen Elements in der Tabelle. Die Befüllung ergibt sich aus den Vorgaben für CDA Level 3 (z.B. ist die "Einheit" in dieser Tabelle mit [R] angegeben, d.h. das Tabellenelement ist verpflichtend anzugeben, befüllt muss es aber nur werden, wenn tatsächlich eine Einheit vorhanden ist). | Spaltenname | Optionalität | Beschreibung |
|---|
| Analyse | [R] | Bezeichung der Analyse (MUSS dem "Begriff"/"displayName" aus dem Value Set "ELGA_Laborparameter" entsprechen). | | Ergebnis | [R] | Numerisches, nominales, ordinales oder narratives Ergebnis der
Analyse. | | Einheit | [R] | Einheit; falls zutreffend, ist der UCUM printName anzugeben. | | Referenzbereich | [O] | Der für die gegebenen Rahmenbedinungen (Patient, Analyse, Untersuchungsmaterial, etc.) passende Referenzbereich ist anzugeben. | | Interpretation | [O] | Codierte Angabe der Interpretation des Ergebnisses (siehe unten). | | Externes Labor | [O] | Angabe von "E", wenn die Analyse von einem externen Dienstleister durchgeführt wurde. |
Anmerkungen zur Tabelle: - Spezialuntersuchungen, die nicht in das angegebene Schema passen (z.B Allergiediagnostik etc.), können bei Bedarf
auch anders dargestellt werden. Ensprechende Beispieldokumente stehen zur Verfügung.
- Für die Spalte "Ergebnis" und "Referenzbereich" wird EMPFOHLEN, bei Dezimalzahlen einen Punkt als Dezimaltrennzeichen zu verwenden – gleich wie im maschinenlesbaren Teil.
Interpretation der Ergebnisse In Laborbefunden ist es üblich, eine codierte Bewertung zu jedem Ergebnis anzugeben. Basierend auf dem Auszug aus dem Value Set "ELGA_ObservationInterpretation" stellt die folgende Tabelle dar, wie eine Darstellung in CDA Level 2, in Abhängigkeit von den CDA Level 3 codierten Interpretationen, aussehen kann. | Darstellung CDA Level 2 | Codierung CDA Level 3 | Beschreibung |
|---|
| Befundinterpretation für numerische Ergebnisse |
|---|
| ++ | HH | Oberhalb des Referenzbereiches und über einer oberen Warngrenze | | + | H | Oberhalb des Referenzbereiches | | | N | Normal (innerhalb des Referenzbereiches) | | - | L | Unterhalb des
Referenzbereiches | | -- | LL | Unterhalb des Referenzbereiches und unter einer unteren Warngrenze | | Befundinterpretation für nicht numerische Ergebnisse |
|---|
| | N | Normal (innerhalb des
Referenzbereiches) | | * | A | Abnormal | | ** | AA | Abnormal Warngrenze |
Darstellung auffälliger/pathologischer Ergebnisse Auffällige/pathologische Ergebnisse SOLLEN für die Darstellung mit dem Stylecode " xELGA_red" versehen werden. Dieser wird für die betroffene Tabellenzeile vergeben (siehe Beispiel). |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.2.110 |
|---|
| Klassifikation | CDA Section level template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.25 | Containment |  Laboratory Report Data Processing Entry (1.0.1) Laboratory Report Data Processing Entry (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.2.8 | Containment |  Übersetzung (1.0.2+20230717) Übersetzung (1.0.2+20230717) | DYNAMIC |
|
|
|---|
| Beziehung | Spezialisierung: Template 1.2.40.0.34.6.0.11.2.102 Laboratory Specialty Section (2021‑01‑21 11:16:21) ref at-lab-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.3.2.1 Speciality-Section (2013‑11‑07) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.2.110"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="15220000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Laboratory test (procedure)"/> <title>Weitere Analysen</title> <!-- CDA Level 2 -->
<text>
<table>
<thead>
<tr>
<th>Analyse</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereich</th> <th>Interpretation</th> </tr> </thead> <tbody>
<!-- auffälliges/pathologisches Ergebnis gekennzeichnet durch styleCode -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>+</td> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <act classCode="ACT" moodCode="EVN">
<code code="15220000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Laboratory test (procedure)"/> <statusCode code="completed"/> <!-- Codierung von "Norovirus-RNA" als "Laboratory Observation" -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="26464-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Leukozyten"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime value="20190201092200+0100"/> <value unit="10*9/L" value="26.0" xsi:type="PQ"/> <interpretationCode code="H" codeSystem="2.16.840.1.113883.5.83" displayName="High"/> <referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-1"/> </text> <value xsi:type="IVL_PQ">
<low value="4" unit="10*9/L"/> <high value="10" unit="10*9/L" inclusive="false"/> </value> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83"/> </observationRange> </referenceRange> </observation> </entryRelationship> </act> </entry></section> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 0 … 1 | F | DOCSECT |  | @moodCode
|
| cs | 0 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Specialty Section (Weitere Analysen) | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.2.110 |  | hl7:templateId
|
| II | | NP | IHE PalM TF3 Rev.10, 6.3.3.1 Laboratory Specialty Section
Strukturell basiert diese Section auf den Vorgaben der IHE. Die Codierung von "section/code" erfolgt allerdings nicht nach den von IHE vorgegebenen LOINC-Codes. Deshalb darf diese templateID nicht angegeben werden. | | | wo [@root='1.3.6.1.4.1.19376.1.3.3.2.1'] | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.2.1 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Section auf Basis eines lokalen Nummernkreises. | | | wo [not(@nullFlavor)] | |  | hl7:code
|
| CE | 1 … 1 | M | Definition des Befundbereiches. | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 15220000 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Laboratory test (procedure) |  | hl7:title
|
| ST | 1 … 1 | M | | | | | CONF | | Elementinhalt muss "Weitere Analysen" sein |
|  | hl7:text
|
| SD.TEXT | 1 … 1 | M | Narrativer Text. Entspricht CDA Level 2. | |  | hl7:entry
|
| | 1 … 1 | M | Enthält die CDA Level 3 codierten Informationen.
Beinhaltet 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | DRIV | | | DRIV (is derived from) deutet an, dass "section/text" aus den CDA Level 3 Entries gerendert wurde und keine medizinisch relevanten Inhalte enthält, die nicht aus den Entries stammen.
|  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:component
|
| | 0 … * | R | Optionale Subsections zur Angabe von Übersetzungen des "text"-Elements in andere Sprachen.
Beinhaltet 1.2.40.0.34.6.0.11.2.8 Übersetzung (DYNAMIC) | |  |  | @typeCode
|
| cs | 0 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.4.15 Befundbewertung
14.4.4.16 Brieftext
14.4.4.17 Beilagen
ELGA Labor- und Mikrobiologiebefunde können alle Kriterien erfüllen, die für Befundberichte von der Akkreditierung für medizinische Laboratorien (ISO 15189:2012) gefordert sind.
Ein Anhängen des PDF-Befundes ist NICHT erforderlich!
Für die vollständige Erfüllung aller Akkreditierungsanforderungen an Befundberichte ist das erstellende Labor verantwortlich; die Akkreditierung wird von der Abt. Akkreditierung Austria im Bundesministerium für Digitalisierung und Wirtschaftsstandort durchgeführt.
Mit Rücksicht auf eine einfache Verwendbarkeit der Befunde durch die Benutzer (z.B. niedergelassene Ärzte), die häufig mit einer großen Anzahl von Laborbefunden eines Patienten konfrontiert sind, ist eine Duplizierung der Daten durch Anhängen einer PDF-Ansicht daher NICHT gestattet.
14.4.4.18 Abschließende Bemerkung
14.4.4.19 Übersetzung
14.4.5 Entry Level Templates
14.4.5.1 Konsultationsgrund Problem Concern Entry
14.4.5.2 Konsultationsgrund Problem Entry
| Id | 1.2.40.0.34.6.0.11.3.31 ref at-cda-bbr- | Gültigkeit ab | 2023‑02‑02 15:40:05 |
|---|
| Status |  Entwurf Entwurf | Versions-Label | 1.1.3 |
|---|
| Name | elgagab_entry_KonsultationsgrundProblem | Bezeichnung | Konsultationsgrund Problem Entry |
|---|
| Beschreibung | Mit dieser Observation wird ein bekanntes relevantes Gesundheitsproblem des Patienten codiert dargestellt.
Die Zeitspanne, in der ein Gesundheitsproblem besteht oder bestanden hat, wird hier angegeben. |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.31 |
|---|
| Klassifikation | CDA Entry Level Template
Template-Typ nicht spezifiziert |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 12 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.1 | Inklusion |  Narrative Text Reference (1.0.1+20210512) Narrative Text Reference (1.0.1+20210512) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.2 | Inklusion |  Original Text Reference (1.0.0+20210219) Original Text Reference (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.42 | Containment |  Laterality Qualifier (1.0.0+20210219) Laterality Qualifier (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.17 | Containment |  Performer Body (1.0.0+20210219) Performer Body (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.36 | Containment |  Author Body (1.0.1+20230717) Author Body (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.3 | Containment |  Informant Body (1.0.1+20211213) Informant Body (1.0.1+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.13 | Containment |  Participant Body (1.0.1+20210628) Participant Body (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.11 | Containment |  Comment Entry (1.0.0+20210219) Comment Entry (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.38 | Containment |  Severity Observation (1.0.0+20210219) Severity Observation (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.35 | Containment |  Criticality Observation (1.0.1+20210628) Criticality Observation (1.0.1+20210628) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.36 | Containment |  Certainty Observation (1.0.0+20210219) Certainty Observation (1.0.0+20210219) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.49 | Containment |  Problem Status Observation (1.0.0+20210219) Problem Status Observation (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Version: Template 1.2.40.0.34.6.0.11.3.31 Konsultationsgrund Problem Entry (2022‑06‑02 15:20:46) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.3.31 Konsultationsgrund Problem Entry (2021‑08‑10 08:43:36) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.3.31 Konsultationsgrund Problem Entry (2021‑02‑19 10:46:53) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.3.31 Konsultationsgrund Problem Entry (2020‑11‑05 15:01:55) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.3.31 Konsultationsgrund Problem Entry (2018‑11‑20 09:56:38) ref at-cda-bbr-
Adaptation: Template 1.2.40.0.34.6.0.11.3.6 Problem Entry (DYNAMIC) ref at-cda-bbr- |
|---|
| Beispiel | | Strukturbeispiel | <observation classCode="OBS" moodCode="EVN" negationInd="false">
<templateId root="1.2.40.0.34.6.0.11.3.31"/> <templateId root="2.16.840.1.113883.10.20.1.28"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.5"/> <id/> <code code="55607006" codeSystem="2.16.840.1.113883.6.96" displayName="Problem (finding)"/> <!-- include template 1.2.40.0.34.6.0.11.9.1 'Narrative Text Reference' (dynamic) 1..1 M -->
<statusCode code="completed"/> <effectiveTime>
<low value="2019"/> </effectiveTime> <!-- choice: 1..1
element hl7:value[not(@nullFlavor)]
element hl7:value[@nullFlavor='OTH']
element hl7:value[@nullFlavor='NA']
-->
<targetSiteCode>
<!-- template 1.2.40.0.34.6.0.11.9.42 'Laterality Qualifier' (2020-02-20T09:00:36) -->
</targetSiteCode> <performer>
<!-- template 1.2.40.0.34.6.0.11.9.17 'Performer Body' (2019-01-17T12:44:16) -->
</performer> <author>
<!-- template 1.2.40.0.34.6.0.11.9.36 'Author Body' (2019-11-20T12:13:04) -->
</author> <informant>
<!-- template 1.2.40.0.34.6.0.11.9.3 'Informant Body' (2019-02-07T13:29:32) -->
</informant> <participant>
<!-- template 1.2.40.0.34.6.0.11.9.13 'Participant Body' (2019-04-03T12:08:16) -->
</participant> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.11 'Comment Entry' (2019-02-07T13:10:44) -->
</entryRelationship> <entryRelationship typeCode="SUBJ" inversionInd="true" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.38 'Severity Observation' (2019-11-21T09:31:57) -->
</entryRelationship> <entryRelationship typeCode="SUBJ" inversionInd="true" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.35 'Criticality Observation' (2019-11-21T09:04:18) -->
</entryRelationship> <entryRelationship typeCode="SUBJ" inversionInd="true" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.36 'Certainty Observation' (2019-11-21T09:11:18) -->
</entryRelationship> <entryRelationship typeCode="REFR" inversionInd="false" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.49 'Problem Status Observation' (2019-12-03T09:46:18) -->
</entryRelationship></observation> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | OBS |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | @negationInd
|
| bl | 1 … 1 | R |
SOLL standardmäßig auf false gesetzt werden.
Kann auf true gesetzt werden, um anzuzeigen, dass das dokumentierte Problem nicht beobachtet wurde.
|  | hl7:templateId
|
| II | 1 … 1 | M | | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.31 |  | hl7:templateId
|
| II | 1 … 1 | M | HL7 CCD Problem observation | |  |  | @root
|
| uid | 1 … 1 | F | 2.16.840.1.113883.10.20.1.28 |  | hl7:templateId
|
| II | 1 … 1 | M | IHE Problem Entry | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.5.3.1.4.5 |  | hl7:id
|
| | 1 … * | M | Systeminterne ID des entsprechenden Problems - zur Referenzierung oder Aggregierung.
Auch wenn nur ein Problem-Entry angegeben ist, soll sich die ID von der ID des Problem/Bedenken-Entry unterscheiden.
| |  | hl7:code
|
| CE | 1 … 1 | M | Das "code"-Element enthält die Art des beschriebenen Gesundheitsproblems, die Verwendung von 'Problem' ist empfohlen.
| | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.205 ELGA_TypeOfProblem_VS (DYNAMIC) |
| | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.1 Narrative Text Reference (DYNAMIC) |  | hl7:text
|
| ED | 1 … 1 | M | | |  |  | hl7:reference
|
| TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im menschenlesbaren Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts mit Zusatzinformationen umschließen.
Alternativ kann @value auch mit dem url-scheme "http" oder "https" beginnen.
| | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') or starts-with(@value,'http') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element, or begin with the 'http' or 'https' url-scheme. | |  | hl7:statusCode
|
| CS | 1 … 1 | M | Fixer Wert: completed | |  |  | @code
|
| CONF | 1 … 1 | F | completed |  | hl7:effectiveTime
|
| IVL_TS | 1 … 1 | M |
Zeitintervall, in dem das Gesundheitsproblems existent war/ist.
Grundsätzlich sind die Vorgaben für „Zeit-Elemente“ zu befolgen.
| |  |  | hl7:low
|
| TS.AT.VAR | 1 … 1 | R | Zeitpunkt des Beginns des Gesundheitsproblems (für den Patienten).
Ist dieser Zeitpunkt nicht bekannt, ist effectiveTime.low mit nullFlavor "UNK" anzugeben
| |  |  | hl7:high
|
| TS.AT.VAR | 0 … 1 | C | Zeitpunkt, an dem das Gesundheitsproblem gelöst wurde oder seitdem das Gesundheitsproblem nicht mehr besteht.
Ist dieser Zeitpunkt nicht bekannt, ist effectiveTime.high mit nullFlavor "UNK" anzugeben
| | | Auswahl | 1 … 1 | | Angabe des Gesundheitsproblems:
-
Codierte Angabe des Gesundheitsproblems:
@value enthält den Code des Gesundheitsproblems aus dem Value Set (ICD-10, ICPC2, ...)
-
Codierte Angabe ohne passenden Code:
xsi:type='CD', nullFlavor: OTH
in diesem Fall ist das Element Translation <translation> verpflichtend
originalText.reference enthält den Verweis auf die narrative Beschreibung des Problems
-
Uncodierte Angabe:
xsi:type='CD', nullFlavor: NA
in diesem Fall ist die Textreferenz <originalText> verpflichtend
originalText.reference enthält den Verweis auf die narrative Beschreibung des Problems
- hl7:value[not(@nullFlavor)]
- hl7:value[@nullFlavor='OTH']
- hl7:value[@nullFlavor='NA']
|  |  | hl7:value
|
| | 0 … 1 | | Codierte Angabe des Gesundheitsproblems
Codesysteme bitte in der aktuellen Version verwenden. Z.B.:
- 1.2.40.0.34.5.184 - ICD-10 BMASGK
- 1.2.40.0.34.5.175 - ICPC2 (International Classification of Primary Care)
- 2.16.840.1.113883.6.254 - ICF (WHO International Classification of Function)
- 2.16.840.1.113883.6.96 - SNOMED CT
- etc.
| | | wo [not(@nullFlavor)] | | | | 1 … 1 | F | CD | | Eingefügt | 0 … 1 | R | von 1.2.40.0.34.6.0.11.9.2 Original Text Reference (DYNAMIC)
Eingegebener Freitext, der die Grundlage der im Entry angegebenen Information ist.
Das Element verweist auf die Stelle im Textbereich (section.text), in dem das Problem beschrieben ist (ohne zusätzliche Informationen, wie Datum, Beschreibung, etc).
Grundsätzlich sind die Vorgaben für „Codierungs-Elemente“ zu befolgen.
| | ED | 0 … 1 | R | Textinhalt, der codiert wurde.
| | | TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im narrativen Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem content-Element mit ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts umschließen und KEINE zusätzlichen Markup oder Strukturelemente. | | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element. | | | CR | 0 … * | |
Qualifier zur genaueren Beschreibung des Problems.
z.B. zur Angabe der Art der Diagnose.
| | | CV | 1 … 1 | M | | | | cs | 1 … 1 | F | 106229004 | | oid | 1 … 1 | F | 2.16.840.1.113883.6.96 | | CD | 1 … 1 | M | | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.23 ELGA_ArtderDiagnose (DYNAMIC) |
| | CD | 0 … * | |
Dieses Feld wird verwendet, wenn Codes aus einem abweichenden Value Set angegeben werden.
z. B. für Übersetzungen in alternative Codesysteme oder wenn kein geeigneter Code im vorgegebene VS vorhanden ist.
| |  |  | hl7:value
|
| | 0 … 1 | | Codierte Angabe ohne passenden Code
| | | wo [@nullFlavor='OTH'] | | | | 1 … 1 | F | CD | | cs | 1 … 1 | F | OTH | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.2 Original Text Reference (DYNAMIC)
Eingegebener Freitext, der die Grundlage der im Entry angegebenen Information ist.
Das Element verweist auf die Stelle im Textbereich (section.text), in dem das Problem beschrieben ist (ohne zusätzliche Informationen, wie Datum, Beschreibung, etc).
Grundsätzlich sind die Vorgaben für „Codierungs-Elemente“ zu befolgen.
| | ED | 1 … 1 | M | Textinhalt, der codiert wurde.
| | | TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im narrativen Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem content-Element mit ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts umschließen und KEINE zusätzlichen Markup oder Strukturelemente. | | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element. | | | CD | 1 … * | M |
Dieses Feld wird verwendet, wenn Codes aus einem abweichenden Value Set angegeben werden.
z. B. für Übersetzungen in alternative Codesysteme oder wenn kein geeigneter Code im vorgegebene VS vorhanden ist.
| |  |  | hl7:value
|
| | 0 … 1 | | Uncodierte Angabe
| | | wo [@nullFlavor='NA'] | | | | 1 … 1 | F | CD | | cs | 1 … 1 | F | NA | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.2 Original Text Reference (DYNAMIC)
Eingegebener Freitext, der die Grundlage der im Entry angegebenen Information ist.
Das Element verweist auf die Stelle im Textbereich (section.text), in dem das Problem beschrieben ist (ohne zusätzliche Informationen, wie Datum, Beschreibung, etc).
Grundsätzlich sind die Vorgaben für „Codierungs-Elemente“ zu befolgen.
| | ED | 1 … 1 | M | Textinhalt, der codiert wurde.
| | | TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im narrativen Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem content-Element mit ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts umschließen und KEINE zusätzlichen Markup oder Strukturelemente. | | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element. | |  | hl7:targetSiteCode
|
| CD | 0 … * | | Anatomische Lage des Problems
Beinhaltet 1.2.40.0.34.6.0.11.9.42 Laterality Qualifier (DYNAMIC) | |  | hl7:performer
|
| | 0 … * | R | Beinhaltet 1.2.40.0.34.6.0.11.9.17 Performer Body (DYNAMIC) | |  | hl7:author
|
| | 0 … * | R | Dieses Author-Element KANN verwendet werden, um anzugeben, wer das Problem dokumentiert hat. Wenn nicht angegeben, gilt das jeweils "darüberlegende" Author-Element (Section, Document)
Beinhaltet 1.2.40.0.34.6.0.11.9.36 Author Body (DYNAMIC) | |  | hl7:informant
|
| | 0 … * | R | Beinhaltet 1.2.40.0.34.6.0.11.9.3 Informant Body (DYNAMIC) | |  | hl7:participant
|
| | 0 … * | R | Beinhaltet 1.2.40.0.34.6.0.11.9.13 Participant Body (DYNAMIC) | |  | hl7:entryRelationship
|
| | 0 … * | R | Beinhaltet 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:entryRelationship
|
| | 0 … 1 | R | Dieses EntryRelationship dient zur Darstellung des Schweregrads des Gesundheitsproblems.
Beinhaltet 1.2.40.0.34.6.0.11.3.38 Severity Observation (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | SUBJ |  |  | @inversionInd
|
| bl | 1 … 1 | F | true |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:entryRelationship
|
| | 0 … 1 | R | Dieses EntryRelationship dient zur Darstellung der Kritizität des Gesundheitsproblems.
Beinhaltet 1.2.40.0.34.6.0.11.3.35 Criticality Observation (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | SUBJ |  |  | @inversionInd
|
| bl | 1 … 1 | F | true |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:entryRelationship
|
| | 0 … 1 | R | Dieses EntryRelationship dient zur Darstellung der Gewissheit, mit der das Gesundheitsproblem besteht
Beinhaltet 1.2.40.0.34.6.0.11.3.36 Certainty Observation (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | SUBJ |  |  | @inversionInd
|
| bl | 1 … 1 | F | true |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  | hl7:entryRelationship
|
| | 0 … 1 | R | Klinischer Status des Gesundheitsproblems
Beinhaltet 1.2.40.0.34.6.0.11.3.49 Problem Status Observation (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | REFR |  |  | @inversionInd
|
| bl | 1 … 1 | F | false |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.5.3 Criticality Observation
14.4.5.4 Anamnese Entry - Labor und Mikrobiologie
| Id | 1.2.40.0.34.6.0.11.3.12 | Gültigkeit ab | 2021‑01‑20 12:36:01 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_entry_AnamneseEntryLaborUndMikrobiologie | Bezeichnung | Anamnese Entry - Labor und Mikrobiologie |
|---|
| Beschreibung | Der "Anamnese Entry - Labor und Mikrobiologie" ermöglicht die Dokumentation der Anamnese. |
|---|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.12 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 3 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.36 | Containment |  Author Body (1.0.1+20230717) Author Body (1.0.1+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.3 | Containment |  Informant Body (1.0.1+20211213) Informant Body (1.0.1+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.164 | Containment |  Anamnese Observation - Labor und Mikrobiologie (1.0.0+20211213) Anamnese Observation - Labor und Mikrobiologie (1.0.0+20211213) | DYNAMIC |
|
|
|---|
| Beispiel | | Beispiel | <entry typeCode="DRIV">
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.12"/> <code code="10164-2" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="History of Present illness Narrative"/> <statusCode code="completed"/> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.164 'Anamnese Observation - Labor und Mikrobiologie' -->
</entryRelationship> <!-- für jeden Aspekt der Krankengeschichte ein "entryRelationship"-Element mit darinliegender Observation -->
</act></entry> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | ACT |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Anamnese Entry - Labor und Mikrobiologie | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.12 |  | hl7:code
|
| CD | 1 … 1 | M | | |  |  | @codeSystemName
|
| st | 0 … 1 | F | LOINC |  |  | @code
|
| CONF | 1 … 1 | F | 10164-2 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.1 (LOINC) |  |  | @displayName
|
| 1 … 1 | F | History of Present illness Narrative |  | hl7:statusCode
|
| CS | 1 … 1 | M | | |  |  | @code
|
| CONF | 1 … 1 | F | completed |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | Datum und Zeit der Anamnesedurchführung | |  | hl7:author
|
| | 0 … * | R | Autor der enthaltenen Informationen (GDA)
Beinhaltet 1.2.40.0.34.6.0.11.9.36 Author Body (DYNAMIC) | |  | hl7:informant
|
| | 0 … * | R | Quelle für die enthaltene Information.
Name der Person und ihre Beziehung zum Patienten (Patient oder Angehöriger, Auskunftsperson - nicht-GDA)
Beinhaltet 1.2.40.0.34.6.0.11.9.3 Informant Body (DYNAMIC) | |  | hl7:entryRelationship
|
| | 1 … * | M | Anamneseinformation
Beinhaltet 1.2.40.0.34.6.0.11.3.164 Anamnese Observation - Labor und Mikrobiologie (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |
|
14.4.5.5 Anamnese Observation - Labor und Mikrobiologie
| Id | 1.2.40.0.34.6.0.11.3.164 | Gültigkeit ab | 2021‑05‑05 10:16:09 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_entry_AnamneseObservationLaborUndMikrobiologie | Bezeichnung | Anamnese Observation - Labor und Mikrobiologie |
|---|
| Beschreibung | Codierung eines Aspekts der Krankengeschichte und ob dieser für einen Patienten zutrifft oder nicht. |
|---|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.164 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.1 | Inklusion |  Narrative Text Reference (1.0.1+20210512) Narrative Text Reference (1.0.1+20210512) | DYNAMIC |
|
|
|---|
| Beispiel | | Beispiel | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1">
<td>Vorgeschichte einer chronischen Harnwegsinfektion</td> <td>trifft zu</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.164"/> <code code="441547007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="History of chronic urinary tract infection (situation)"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <value xsi:type="BL" value="true"/></observation> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | OBS |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Anamnese Observation - Labor und Mikrobiologie
| |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.164 |  | hl7:id
|
| II | 0 … 1 | | Zwecks Rückverfolgbarkeit kann eine ID angegeben werden.
| |  | hl7:code
|
| CD | 1 … 1 | M | Code des Aspekts der Krankengeschichte | |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.66 ELGA_AnamneseLaborMikrobiologie_VS (DYNAMIC) |
| | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.9.1 Narrative Text Reference (DYNAMIC)
Das "text"-Element wird verwendet, um einen Verweis zum narrativen Text ("section/text") herzustellen.
|  | hl7:text
|
| ED | 0 … 1 | | | |  |  | hl7:reference
|
| TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im menschenlesbaren Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts mit Zusatzinformationen umschließen.
Alternativ kann @value auch mit dem url-scheme "http" oder "https" beginnen.
| | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') or starts-with(@value,'http') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element, or begin with the 'http' or 'https' url-scheme. | |  | hl7:statusCode
|
| CS | 1 … 1 | M | | |  |  | @code
|
| CONF | 1 … 1 | F | completed |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | Medizinisch relevantes Datum und Zeit. | |  | hl7:value
|
| BL | 1 … 1 | M | Codiert, ob der Aspekt der Krankengeschichte für den Patienten zutrifft oder nicht. | | | wo [@xsi:type='BL'] | |
|
14.4.5.6 Angeforderte Untersuchung Entry
| Id | 1.2.40.0.34.6.0.11.3.169 | Gültigkeit ab | 2021‑05‑05 14:57:08 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_entry_AngeforderteUntersuchungEntry | Bezeichnung | Angeforderte Untersuchung Entry |
|---|
| Beschreibung | Entry zur codierten Darstellung einer angeforderten Untersuchung. |
|---|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.169 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.1 | Inklusion |  Narrative Text Reference (1.0.1+20210512) Narrative Text Reference (1.0.1+20210512) | DYNAMIC |
|
|
|---|
| Beispiel | | Beispiel | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="req-1">
<td>MRSA-Screening</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<procedure classCode="PROC" moodCode="RQO">
<templateId root="1.2.40.0.34.6.0.11.3.169"/> <code code="301786007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Multi-resistant staphylococcus aureus screening (procedure)"/> <text>
<reference value="#req-1"/> </text></procedure> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | PROC |  | @moodCode
|
| cs | 1 … 1 | F | RQO |  | hl7:templateId
|
| II | 1 … 1 | M | Angeforderte Untersuchung Entry | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.169 |  | hl7:id
|
| II | 0 … 1 | | Zwecks Rückverfolgbarkeit kann eine ID angegeben werden. | | | Auswahl | 1 … 1 | | Code der angeforderten Untersuchung. Elemente in der Auswahl:- hl7:code[not(@nullFlavor)]
- hl7:code[@nullFlavor='OTH']
|  |  | hl7:code
|
| CD | 0 … 1 | | Codierung auf Basis des Value Sets "ELGA_RequestedProcedures_VS".
Codes, die im aktuellen Value Set nicht vorhanden sind, werden von der ELGA GmbH ergänzt. Bitte dazu einen Vorschlag aus einem Code System (SNOMED CT, LOINC, etc.) frei wählen und an cda@elga.gv.at zusenden. | | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.70 ELGA_RequestedProcedures_VS (DYNAMIC) |
|  |  | hl7:code
|
| CD | 0 … 1 | | Codierung von angeforderten Untersuchungen, die nicht im aktuellen Value Set "ELGA_RequestedProcedures_VS" enthalten sind.
| | | wo [@nullFlavor='OTH'] | | | | Beispiel | Codierung einer angeforderten Untersuchung ohne passenden Code <code nullFlavor="OTH">
<translation code="alternativerCode" codeSystem="alternativeCodeSystem" codeSystemName="NameDesCodeSystems" displayName="klarTextDarstellung"/></code> | | | Schematron assert | role | error | | | | test | hl7:translation[not(@nullFlavor)] | | | | Meldung | Wenn code[@nullFlavor='OTH'] dann MUSS "code/translation" anwesend sein. | | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.9.1 Narrative Text Reference (DYNAMIC)
Das "text"-Element wird verwendet, um einen Verweis zum narrativen Text ("section/text") herzustellen.
|  | hl7:text
|
| ED | 0 … 1 | | | |  |  | hl7:reference
|
| TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im menschenlesbaren Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts mit Zusatzinformationen umschließen.
Alternativ kann @value auch mit dem url-scheme "http" oder "https" beginnen.
| | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') or starts-with(@value,'http') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element, or begin with the 'http' or 'https' url-scheme. | |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | Datum und Zeit der Anforderung. | |
|
14.4.5.7 Probeninformation (Specimen Entry)
14.4.5.8 Specimen Collection
| Id | 1.2.40.0.34.6.0.11.3.161 ref at-cda-bbr- | Gültigkeit ab | 2021‑01‑18 13:46:31 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_entry_SpecimenCollection | Bezeichnung | Specimen Collection |
|---|
| Beschreibung | Die "Specimen Collection" stellt die CDA Level 3 Codierung von GENAU einem Untersuchungsmaterial dar.
Für die Darstellung der Information in CDA Level 2 bitte das Template "Probeninformation (Specimen Section)" konsultieren. |
|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 2 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.24 | Containment |  Performer - Laboratory (1.0.0+20211213) Performer - Laboratory (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.162 | Containment |  Specimen Received (1.0.0+20211213) Specimen Received (1.0.0+20211213) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.11.30021 Abnahmeinformationen (Specimen Collection) (2014‑03‑04) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Material-ID</th> <th>Probenentnahme</th> <th>Untersuchtes Material</th> <th>Probenentnahme durch</th> <th>Probeneingang</th> <th>Bemerkung Labor</th> </tr> </thead> <tbody>
<tr ID="SPEC-1-1">
<td>PL-081201-02</td> <td>01.12.2012 06:34</td> <td>Plasma, Linke Ellenbeuge</td> <td>Dr. Humpel, Amadeus Spital</td> <td>01.12.2012 08:15</td> <td ID="SpecimenComment01">leicht hämolytisch</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<procedure classCode="PROC" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.161"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.2"/> <code code="33882-2" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Collection date of Specimen"/> <effectiveTime value="20121201063400+0100"/> <targetSiteCode code="LACF" codeSystem="2.16.840.1.113883.5.1052" codeSystemName="HL7:ActSite" displayName="left antecubital fossa"/> <!-- Für die Abnahme verantwortliche Person/Organisation -->
<performer typeCode="PRF">
<templateId root="1.2.40.0.34.6.0.11.9.24"/> <templateId root="1.3.6.1.4.1.19376.1.3.3.1.7"/> <effectiveTime value="20121201063400+0100"/> <assignedEntity>
<id root="1.2.40.0.34.3.1.99"/> <addr>
<streetName>Währinger G.</streetName> <houseNumber>18-20</houseNumber> <postalCode>1090</postalCode> <city>Wien</city> <state>Wien</state> <country>AUT</country> </addr> <telecom value="tel:+43.1.40400"/> <telecom value="fax:+43.1.40400.1212"/> <telecom value="http://www.amadeusspital.at "/> <assignedPerson>
<name>
<prefix qualifier="AC">Dr.</prefix> <family>Humpel</family> <given>Robert</given> </name> </assignedPerson> <representedOrganization>
<id root="1.2.40.0.34.99.111.0.1"/> <name>Amadeus Spital</name> <telecom value="tel:+43.1.40400"/> <addr nullFlavor="UNK"/> </representedOrganization> </assignedEntity> </performer> <!-- Untersuchungsmaterial -->
<participant typeCode="PRD">
<participantRole classCode="SPEC">
<id extension="PL-081201-02" root="2.16.840.1.113883.3.933.1.1"/> <playingEntity>
<code code="119361006" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Plasma specimen"/> </playingEntity> </participantRole> </participant> <entryRelationship>
<!-- template 1.2.40.0.34.6.0.11.3.162 'Specimen Received' -->
</entryRelationship></procedure> |
|
|---|
|
14.4.5.9 Specimen Received
| Id | 1.2.40.0.34.6.0.11.3.162 ref at-cda-bbr- | Gültigkeit ab | 2021‑01‑18 14:25:23 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atlab_entry_SpecimenReceived | Bezeichnung | Specimen Received |
|---|
| Beschreibung | Das Template "Specimen Received" codiert den Eingang von GENAU einem Untersuchungsmaterial in CDA Level 3.
Für die Darstellung der Information in CDA Level 2 bitte das Template "Probeninformation (Specimen Section)" konsultieren. |
|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.3.11 | Containment |  Comment Entry (1.0.0+20210219) Comment Entry (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.11.30022 Annahmeinformationen (Specimen Received) (2014‑03‑04) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Material-ID</th> <th>Probenentnahme</th> <th>Untersuchtes Material</th> <th>Probenentnahme durch</th> <th>Probeneingang</th> <th>Bemerkung Labor</th> </tr> </thead> <tbody>
<tr ID="SPEC-1-1">
<td>PL-081201-02</td> <td>01.12.2012 06:34</td> <td>Plasma, Linke Ellenbeuge</td> <td>Dr. Humpel, Amadeus Spital</td> <td>01.12.2012 08:15</td> <td ID="SpecimenComment01">leicht hämolytisch</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.162"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.3"/> <code code="SPRECEIVE" codeSystem="1.3.5.1.4.1.19376.1.5.3.2" codeSystemName="IHEActCode"/> <effectiveTime value="20121201081500+0100"/> <entryRelationship>
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.11"/> <templateId root="2.16.840.1.113883.10.20.1.40"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.2"/> <code code="48767-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Annotation comment"/> <text>
<reference value="#SpecimenComment01"/> </text> <statusCode code="completed"/> </act> </entryRelationship></act> |
|
|---|
|
14.4.5.10 Laboratory Report Data Processing Entry
| Id | 1.2.40.0.34.6.0.11.3.25 ref at-cda-bbr- | Gültigkeit ab | 2023‑06‑01 13:53:03Andere Versionen mit dieser Id:  atcdabbr_entry_LaboratoryReportDataProcessing vom 2019‑05‑07 12:59:27 atcdabbr_entry_LaboratoryReportDataProcessing vom 2019‑05‑07 12:59:27
|
|---|
| Status |  Entwurf Entwurf | Versions-Label | 1.0.1 |
|---|
| Name | atcdabbr_entry_LaboratoryReportDataProcessing | Bezeichnung | Laboratory Report Data Processing Entry |
|---|
| Beschreibung | Der "Laboratory Report Data Processing Entry" repräsentiert die CDA Level 3 Codierung der Ergebnisse eines Befundbereiches. Im zugehörigen "section/text" befinden sich diese Ergebnisse in CDA Level 2. Das "entry"-Element MUSS mit dem Attribut @typeCode="DRIV" versehen werden, um anzuzeigen, dass der CDA Level 2 vollständig aus dem CDA Level 3 erzeugt werden kann. Das "entry"-Element enthält genau ein "act"-Subelement, das gemäß IHE PaLM TF3 "Specimen Act" genannt wird (nicht zu verwechseln mit den Templates "Probeninformation (Specimen Section)", "Probeninformation (Specimen Entry)", "Specimen Collection" oder "Specimen Received").
Innerhalb des "Specimen Act" werden alle weiteren Elemente (Untersuchungsmaterial, Befundgruppen, Analysen, etc.) codiert. Der "Specimen Act" MUSS zumindest ein weiteres Element beinhalten. |
|
| Kontext | Geschwisterknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.25 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 8 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.24 | Containment |  Performer - Laboratory (1.0.0+20211213) Performer - Laboratory (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.161 | Containment |  Specimen Collection (1.0.0+20211213) Specimen Collection (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.165 | Containment |  Notification Organizer (1.0.0+20211213) Notification Organizer (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.167 | Containment |  Laboratory Isolate Organizer (1.0.1) Laboratory Isolate Organizer (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.26 | Containment |  Laboratory Battery Organizer (1.0.1) Laboratory Battery Organizer (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.27 | Containment |  Laboratory Observation (2.0.1) Laboratory Observation (2.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.19 | Containment |  Eingebettetes Objekt Entry (1.0.2+20230717) Eingebettetes Objekt Entry (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.11 | Containment |  Comment Entry (1.0.0+20210219) Comment Entry (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Version: Template 1.2.40.0.34.6.0.11.3.25 Laboratory Report Data Processing Entry (2019‑05‑07 12:59:27) ref at-cda-bbr-
Spezialisierung: Template 1.3.6.1.4.1.19376.1.3.1 BC Laboratory Report Data Processing Entry (DYNAMIC) ref bccdapilot- |
|---|
| Beispiel | | Beispiel | <entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <templateId root="1.3.6.1.4.1.19376.1.3.1"/> <!-- "Specimen Act" gemäß IHE PaLM TF3 -->
<act classCode="ACT" moodCode="EVN">
<code code="600" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Endokrinologie"/> <statusCode code="completed"/> <!-- choice: 1..* -->
<entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.161 'Specimen Collection' -->
</entryRelationship> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.165 'Notification Organizer' -->
</entryRelationship> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.167 'Laboratory Isolate Organizer' -->
</entryRelationship> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.26 'Laboratory Battery Organizer' -->
</entryRelationship> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.27 'Laboratory Observation' -->
</entryRelationship> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.19 'Eingebettetes Objekt Entry' -->
</entryRelationship> <entryRelationship typeCode="COMP" contextConductionInd="true">
<!-- template 1.2.40.0.34.6.0.11.3.11 'Comment Entry' -->
</entryRelationship> </act></entry> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| II | 1 … 1 | M | Laboratory Report Data Processing Entry | |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.25 | | II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.4.2 Laboratory Report Data Processing Entry | |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.1 | | | 1 … 1 | M | Specimen Act | |  | @classCode
|
| cs | 1 … 1 | F | ACT |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:code
|
| CD | 1 … 1 | M | Der Code für den Befundbereich MUSS dem der übergeordneten "Laboratory Specialty Section" entsprechen. | |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | @code=../../../hl7:code/@code and @codeSystem=../../../hl7:code/@codeSystem | | | | Meldung | Die hier angegebenen @code und @codeSystem MÜSSEN mit den Werten der übergeordneten Section ident sein. | |  | hl7:statusCode
|
| CS | 1 … 1 | M | Der "statusCode" kann folgende Werte annehmen:
-
completed: zu verwenden, wenn alle erwarteten Analyseergebnisse für diesen Befundbereich abgeschlossen sind.
-
aborted: zu verwenden, wenn zumindest ein Analyseergebnis für diesen Befundbereich nicht abgeschlossen werden konnte (abgebrochen wurde).
Der Wert "active" DARF NICHT verwendet werden. Befunde, die noch nicht abgeschlossen sind, sind mit /ClinicalDocument/sdtc:statusCode[@code="active"] zu codieren. Siehe dazu auch Template "Laboratory Observation", wo ein Beispiel zu einer noch nicht abgeschlossenen Analyse ("Wert folgt") angegeben ist. | | | | CONF | | @code muss "completed" sein | | oder | | @code muss "aborted" sein |
|  | hl7:performer
|
| | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).
Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.161 Specimen Collection (DYNAMIC)
- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.165 Notification Organizer (DYNAMIC)
- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.167 Laboratory Isolate Organizer (DYNAMIC)
- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.26 Laboratory Battery Organizer (DYNAMIC)
- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (DYNAMIC)
- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.19 Eingebettetes Objekt Entry (DYNAMIC)
- hl7:entryRelationship welches enthält Template 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC)
|  |  | hl7:entryRelationship
|
| | 0 … * | C | Probeninformation
Informationen zur CDA Level 2 Darstellung können der Section "Probeninformation (Specimen Section)" entnommen werden.
Beinhaltet 1.2.40.0.34.6.0.11.3.161 Specimen Collection (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true | | | Constraint | Die Verwendung von "Specimen Collection" innerhalb des "Laboratory Report Data Processing Entry" ist NUR dann zulässig, wenn NUR EIN Befundbereich ("Laboratory Specialty Section") im gesamten Befund vorkommt und die "Specimen Collection" nicht schon als Teil der Section "Probeninformation (Specimen Section)" codiert wurde. |  |  | hl7:entryRelationship
|
| | 0 … 1 | | Sammlung von wichtigen Erregern ("Notifiable Condition") bzw. Dokumentation von separat erfolgten Meldungen ans EMS ("Case Identification").
Beinhaltet 1.2.40.0.34.6.0.11.3.165 Notification Organizer (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:entryRelationship
|
| | 0 … * | | Codierung von kulturellen Erregernachweisen.
Beinhaltet 1.2.40.0.34.6.0.11.3.167 Laboratory Isolate Organizer (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:entryRelationship
|
| | 0 … * | | Codierung einer Befundgruppe innerhalb eines Befundbereiches.
Beinhaltet 1.2.40.0.34.6.0.11.3.26 Laboratory Battery Organizer (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:entryRelationship
|
| | 0 … * | | Codierung von Analyseergebnissen, die direkt einem Befundbereich ("Laboratory Specialty Section") zugeordnet werden. Das kann z.B. im Rahmen eines Mikrobiologiebefundes erforderlich sein, in dem prinzpiell eine weitere Gliederung in Befundgruppen ("Laboratory Battery Organizer") wegfällt.
Beinhaltet 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:entryRelationship
|
| | 0 … * | | Beinhaltet 1.2.40.0.34.6.0.11.3.19 Eingebettetes Objekt Entry (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:entryRelationship
|
| | 0 … * | | Codierung des Kommentars für einen gesamten Befundbereich.
Beinhaltet 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true | | | Beispiel | Kommentar zum Befundbereich <!-- Befundbereich -->
<section>
<templateId root="1.2.40.0.34.6.0.11.2.102"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="300" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Hämatologie"/> <title>Hämatologie</title> <!-- CDA Level 2 -->
<text>
<!-- ... -->
<!-- Inhalte von meldepflichtigen Erkrankungen, Befundgruppen, etc. -->
<!-- ... -->
<!-- Kommentar zum Befundbereich als letztes Element in "section/text" -->
<table>
<thead>
<tr>
<th>Kommentar zur Hämatologie</th> </tr> </thead> <tbody>
<tr>
<td>
<paragraph>
<content ID="haematologyComment1">Das ist ein Kommentar zum Befundbereich "Hämatologie".</content> </paragraph> </td> </tr> </tbody> </table> </text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <templateId root="1.3.6.1.4.1.19376.1.3.1"/> <act classCode="ACT" moodCode="EVN">
<code code="300" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Hämatologie"/> <statusCode code="completed"/> <!-- ... -->
<!-- Codierung von meldepflichtigen Erkrankungen, Befundgruppen, Analysen, etc. -->
<!-- ... -->
<!-- Codierung des Kommentars zum Befundbereich -->
<entryRelationship typeCode="COMP">
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <templateId root="2.16.840.1.113883.10.20.1.40"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.2"/> <code code="48767-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Annotation Comment"/> <text>
<reference value="#haematologyComment1"/> </text> <statusCode code="completed"/> </act> </entryRelationship> </act> </entry></section> |
|
14.4.5.11 Notification Organizer
14.4.5.12 Notifiable Condition
14.4.5.13 Case Identification
| Id | 1.2.40.0.34.6.0.11.3.170 ref at-cda-bbr- | Gültigkeit ab | 2020‑09‑29 11:27:13 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atcdabbr_entry_caseIdentification | Bezeichnung | Case Identification |
|---|
| Beschreibung | Die Case Identification kann verwendet werden, um die EMS Fall-ID anzugeben, die im Rahmen einer EMS-Meldung vergeben wurde. Sollte eine Meldung an das EMS fehlgeschlagen sein, kann dieser Umstand mit observation/id[@nullFlavor='NI'] codiert werden. |
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.170 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.1 | Inklusion |  Narrative Text Reference (1.0.1+20210512) Narrative Text Reference (1.0.1+20210512) | DYNAMIC |
|
|
|---|
| Beziehung | Adaptation: Template 1.2.40.0.34.6.0.11.3.91 EMS Case Identification Labormeldung (2020‑04‑22 15:31:59) ref epims- |
|---|
| Beispiel | | Beispiel | <observation classCode="CASE" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.170"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.1.2"/> <id root="1.2.40.0.34.3.1.1" extension="Beispiel-EMS-Fall-ID"/> <id root="1.2.3.999" extension="--example only--"/> <code code="416341003" codeSystem="2.16.840.1.113883.6.96" displayName="Case Management Started"/> <statusCode code="completed"/> <effectiveTime value="20200725145409+0100"/> <value code="A00" codeSystem="1.2.40.0.34.5.171" displayName="Cholera"/></observation> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | CASE |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Case Identification | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.170 |  | hl7:templateId
|
| II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.4.9 Case Identification | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.1.1.2 | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:id
- hl7:id[@nullFlavor='NI']
|  |  | hl7:id
|
| II | 0 … 1 | | Angabe der EMS Fall-ID. | | | uid | 1 … 1 | F | 1.2.40.0.34.3.1.1 | | st | 1 … 1 | R | Angabe der EMS Fall-ID. |  |  | hl7:id
|
| II | 0 … 1 | | Sollte die EMS-Meldung fehlgeschlagen sein, wird dies mit @nullFlavor='NI' dokumentiert. | | | wo [@nullFlavor='NI'] | |  | hl7:id
|
| II | 0 … * | | Fall-Identifikatoren außerhalb der EMS Domäne (z.B.: lokale IDs).
Es gelten die Vorgaben des entsprechenden Kapitels des Allgemeinen Leitfadens. | |  | hl7:code
|
| CD | 1 … 1 | M | | |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 1 … 1 | F | 416341003 |  |  | @codeSystem
|
| 1 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 1 … 1 | F | Case Management Started | | Eingefügt | 0 … 1 | | von 1.2.40.0.34.6.0.11.9.1 Narrative Text Reference (DYNAMIC)
Das "text"-Element wird verwendet, um einen Verweis zum narrativen Text ("section/text") herzustellen. |  | hl7:text
|
| ED | 0 … 1 | | | |  |  | hl7:reference
|
| TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im menschenlesbaren Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts mit Zusatzinformationen umschließen.
Alternativ kann @value auch mit dem url-scheme "http" oder "https" beginnen.
| | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') or starts-with(@value,'http') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element, or begin with the 'http' or 'https' url-scheme. | |  | hl7:statusCode
|
| CS | 1 … 1 | M | | |  |  | @code
|
| CONF | 1 … 1 | F | completed |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | R | Diagnosedatum | |  | hl7:value
|
| CD | 1 … 1 | M | Codierung der meldepflichtigen Krankheit. | |  |  | @xsi:type
|
| cs | 1 … 1 | F | CD |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.19 EMS Meldepflichtige Krankheiten VS (DYNAMIC) |
|
|
14.4.5.14 Laboratory Isolate Organizer
| Id | 1.2.40.0.34.6.0.11.3.167 ref at-cda-bbr- | Gültigkeit ab | 2023‑06‑01 13:39:19Andere Versionen mit dieser Id:  atcdabbr_entry_LaboratoryIsolateOrganizer vom 2021‑02‑02 11:18:12 atcdabbr_entry_LaboratoryIsolateOrganizer vom 2021‑02‑02 11:18:12
|
|---|
| Status |  Entwurf Entwurf | Versions-Label | 1.0.1 |
|---|
| Name | atcdabbr_entry_LaboratoryIsolateOrganizer | Bezeichnung | Laboratory Isolate Organizer |
|---|
| Beschreibung | Alle kulturellen Erregernachweise werden im zugehörigen "section/text" (siehe "Laboratory Specialty Section (Kultureller Erregernachweis)") als Tabelle dargestellt. Jede Zeile dieser Tabelle enthält die Bezeichnung des Erregers, die Methodik der Untersuchungsdurchführung sowie das Ergebnis bzw. die Keimzahl. Kommentare zu einzelnen Nachweisen werden gegebenenfalls als Fußnoten zur Tabelle dargestellt. Für jeden einzelnen Erregernachweises wird für die Codierung ein "Laboratory Isolate Organizer" verwendet.
Für die Codierung
- des Erregers MUSS das Value Set "ELGA_Mikroorganismen_VS" verwendet werden;
- der Methodik enthält das Value Set "ELGA_Laborparameter" entsprechende Werte;
- des Ergebnisses bzw. der Keimzahl kann z.B.:
- ein Wert aus dem Value Set "ELGA_NachweisErreger_VS" verwendet werden;
- quantitativ (z.B. Angabe der koloniebildenden Einheiten) erfolgen;
Die Methodik sowie das Ergebnis bzw. die Keimzahl werden über eine "Laboratory Observation" abgebildet.
Eventuell vorliegende Antibiogramme werden in einer separaten Tabelle in "section/text" (siehe "Laboratory Specialty Section (Kultureller Erregernachweis)") dargestellt.
Innerhalb des "Laboratory Isolate Organizers" wird das Antibiogram eines Erregers über einen "Laboratory Battery Organizer" und darin enthaltene "Laboratory Observations" (für jedes Antibiotikum eine Observation) codiert. "observation/code" enthält den Code des getesteten Antibiotikums aus dem Value Set "ELGA_Antibiogramm_VS". Das Ergebnis kann sowohl quantitativ (MHK-Wert) über "observation/value" als auch qualitativ (RSI-Interpretation) über "observation/interpretationCode" codiert werden. |
|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 5 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.24 | Containment |  Performer - Laboratory (1.0.0+20211213) Performer - Laboratory (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.26 | Containment |  Laboratory Battery Organizer (1.0.1) Laboratory Battery Organizer (1.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.27 | Containment |  Laboratory Observation (2.0.1) Laboratory Observation (2.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.19 | Containment |  Eingebettetes Objekt Entry (1.0.2+20230717) Eingebettetes Objekt Entry (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.11 | Containment |  Comment Entry (1.0.0+20210219) Comment Entry (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Version: Template 1.2.40.0.34.6.0.11.3.167 Laboratory Isolate Organizer (2021‑02‑02 11:18:12) ref at-cda-bbr-
Spezialisierung: Template 1.2.40.0.34.11.30025 Kultureller Keimnachweis (Laboratory Isolate Organzier) (2017‑02‑23) ref elgabbr-
Version: Template 1.2.40.0.34.11.30025 Kultureller Keimnachweis (Laboratory Isolate Organzier) (2017‑02‑23) ref elgabbr- |
|---|
| Beispiel | | Beispiel | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Erreger</th> <th>Methode</th> <th>Ergebnis / Keimzahl</th> </tr> </thead> <tbody>
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">Staphylococcus epidermidis</td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">nachgewiesen</td> </tr> <tr ID="OBS-MIBI-2">
<!-- Dokumentation eines weiteren Erregers -->
</tr> </tbody></table><paragraph styleCode="xELGA_h3">Antibiogramm</paragraph><table>
<thead>
<tr>
<th>Wirkstoff</th> <th>Staphylococcus epidermidis</th> <th>
<!-- Dokumentation eines weiteren Erregers -->
</th> </tr> </thead> <tfoot>
<tr>
<td>S = sensibel bei Standarddosierung, I = sensibel bei erhöhter Exposition, R = resistent, [] minimale Hemmkonzentration in mg/L</td> </tr> </tfoot> <tbody>
<tr>
<td ID="OBS-AB-1">Penicillin</td> <td ID="OBS-AB-1-1">R</td> <td ID="OBS-AB-1-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> <tr>
<td ID="OBS-AB-2">Oxacillin</td> <td ID="OBS-AB-2-1">S</td> <td ID="OBS-AB-2-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> <tr>
<td ID="OBS-AB-3">Erythromycin</td> <td ID="OBS-AB-3-1">S</td> <td ID="OBS-AB-3-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> <tr>
<td ID="OBS-AB-4">Clindamycin</td> <td ID="OBS-AB-4-1">S</td> <td ID="OBS-AB-4-2">
<!-- Resistenz eines weiteren Erregers -->
</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="60875001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Staphylococcus epidermidis">
<originalText>
<reference value="#OBS-MIBI-1-1"/> </originalText> </code> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="11475-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Microorganism identified in Specimen by Culture">
<originalText>
<reference value="#OBS-MIBI-1-2"/> </originalText> </code> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" displayName="Detected (qualifier value)">
<originalText>
<reference value="#OBS-MIBI-1-3"/> </originalText> </value> </observation> </component> <!-- Antibiogramm -->
<component typeCode="COMP">
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="365705006" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Finding of antimicrobial susceptibility (finding)"/> <statusCode code="completed"/> <component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18964-7" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Penicillin [Susceptibility]"/> <text>
<reference value="#OBS-AB-1-1"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" nullFlavor="UNK"/> <interpretationCode code="R" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Resistant"/> </observation> </component> <!-- ... -->
<!-- Codierung weiterer Antibiogrammergebnisse für "Staphylococcus epidermidis" -->
<!-- ... -->
</organizer> </component></organizer> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | CLUSTER |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Isolate Organizer | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.167 |  | hl7:templateId
|
| II | 1 … 1 | R | IHE PalM TF3 Rev.10, 6.3.4.11 Laboratory Isolate Organizer | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.1.5 |  | hl7:statusCode
|
| CS | 1 … 1 | M | Der "statusCode" kann folgende Werte annehmen:
-
completed: zu verwenden, wenn alle erwarteten Analyseergebnisse für dieses Isolat abgeschlossen sind.
-
aborted: zu verwenden, wenn zumindest ein Analyseergebnis für dieses Isolat nicht abgeschlossen werden konnte (abgebrochen wurde).
Der Wert "active" DARF NICHT verwendet werden. Befunde, die noch nicht abgeschlossen sind, sind mit /ClinicalDocument/sdtc:statusCode[@code="active"] zu codieren. Siehe dazu auch Template "Laboratory Observation", wo ein Beispiel zu einer noch nicht abgeschlossenen Analyse ("Wert folgt") angegeben ist. | | | | CONF | | @code muss "completed" sein | | oder | | @code muss "aborted" sein |
| | Auswahl | 1 … 1 | | Medizinisch relevantes Datum und Zeit. In der Regel Abnahmedatum/-zeit des Untersuchungsmaterials. Elemente in der Auswahl:- hl7:effectiveTime[not(@nullFlavor)]
- hl7:effectiveTime[@nullFlavor='UNK']
|  |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | | | | wo [not(@nullFlavor)] | |  |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | |  | hl7:specimen
|
| | 1 … 1 | M | | |  |  | @typeCode
|
| cs | 1 … 1 | F | SPC |  |  | hl7:specimenRole
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | SPEC | | II | 0 … 1 | | Eindeutige ID des Labors für dieses Isolat. | |  |  |  | hl7:specimenPlayingEntity
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | MIC | | Auswahl | 1 … 1 | | Codierung des ermittelten Keims.
Für die Codierung des ermittelten Keims SOLL grundsätzlich ein Wert aus dem Value Set "ELGA_Mikroorganismen_VS" verwendet werden. Über "code/translation" ist es möglich, weitere Codes anzugeben, die den Keim noch präziser beschreiben.
Sollte im Value Set "ELGA_Mikroorganismen_VS" kein Code für den Keim verfügbar sein, kann dieser dennoch maschinenlesbar hinterlegt werden mit der Kennzeichnung, dass der Wert nicht aus dem Value Set stammt. Das gilt auch für den Fall, dass ein passender SNOMED CT Code exisitiert, aber nicht im aktuellen Value Set "ELGA_Mikroorganismen_VS" enthalten ist. Es wird gebeten, dass benötigte Codes umgehend an ELGA gemeldet werden, um diese im Zuge von Reviewzyklen in die Codelisten und Value Sets einpflegen zu können. - hl7:code[not(@nullFlavor)]
- hl7:code[@nullFlavor='OTH']
| | CD | 0 … 1 | | Codierung des ermittelten Keims.
| | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.188 ELGA_Mikroorganismen_VS (DYNAMIC) |
| | CD | 0 … 1 | | Codierung des ermittelten Keims, der nicht im aktuellen Value Set "ELGA_Mikroorganismen_VS" enthalten ist.
| | | wo [@nullFlavor='OTH'] | | | | Schematron assert | role | error | | | | test | hl7:translation[not(@nullFlavor)] | | | | Meldung | Wenn code[@nullFlavor='OTH'] dann MUSS "code/translation" anwesend sein. | |  | hl7:performer
|
| | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).
Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.26 Laboratory Battery Organizer (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.19 Eingebettetes Objekt Entry (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC)
|  |  | hl7:component
|
| | 0 … * | | Der "Laboratory Battery Organizer" wird verwendet, um z.B. das Antibiogramm des nachgewiesenen Erregers abzubilden.
Beinhaltet 1.2.40.0.34.6.0.11.3.26 Laboratory Battery Organizer (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | | Beispiel | Interpretation (R, S, I) und minimale Hemmkonzentration (MHK) <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<paragraph styleCode="xELGA_h3">Antibiogramm</paragraph><table>
<thead>
<tr>
<th>Wirkstoff</th> <th>Escherichia coli</th> <th>Francisella tularensis</th> </tr> </thead> <tfoot>
<tr>
<td>S = sensibel bei Standarddosierung, I = sensibel bei erhöhter Exposition, R = resistent, [] minimale Hemmkonzentration in mg/L</td> </tr> </tfoot> <tbody>
<!-- ... -->
<!-- Dokumentation weiterer Antibiogrammergebnisse -->
<!-- ... -->
<tr>
<td ID="OBS-AB-1">Gentamicin</td> <td ID="OBS-AB-1-1">R</td> <td ID="OBS-AB-1-2">[0.75]</td> </tr> <tr>
<td ID="OBS-AB-1">Tigecyclin</td> <td ID="OBS-AB-1-1">S [0.25]</td> <td ID="OBS-AB-1-2"/> </tr> <!-- ... -->
<!-- Dokumentation weiterer Antibiogrammergebnisse -->
<!-- ... -->
</tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Escherichia coli" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="112283007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Escherichia coli"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<!-- Codierung von Methode und Ergebnis / Keimzahl -->
</component> <!-- Antibiogramm -->
<component typeCode="COMP">
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="365705006" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Finding of antimicrobial susceptibility (finding)"/> <statusCode code="completed"/> <component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18928-2" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Gentamicin [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" nullFlavor="UNK"/> <interpretationCode code="R" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Resistant"/> </observation> </component> <component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="42357-4" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Tigecycline [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" value="0.25" unit="mg/L"/> <interpretationCode code="S" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Susceptible"/> </observation> </component> </organizer> </component></organizer><!-- "Laboratory Isolate Organizer" für Keim "Francisella tularensis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="51526001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Francisella tularensis"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<!-- Codierung von Methode und Ergebnis / Keimzahl -->
</component> <!-- Antibiogramm -->
<component typeCode="COMP">
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="365705006" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Finding of antimicrobial susceptibility (finding)"/> <statusCode code="completed"/> <component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18928-2" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Gentamicin [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" value="0.75" unit="mg/L"/> </observation> </component> </organizer> </component></organizer> |  |  | hl7:component
|
| | 0 … * | | Die "Laboratory Observation" wird verwendet, um z.B. die Untersuchungsmethode sowie das ermittelte Ergebnis / Keimzahl abzubilden.
Beinhaltet 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | | Beispiel | Erreger nicht nachweisbar <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Erreger</th> <th>Methode</th> <th>Ergebnis / Keimzahl</th> </tr> </thead> <tbody>
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">Staphylococcus epidermidis</td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">nicht nachgewiesen</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="60875001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Staphylococcus epidermidis"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="11475-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Microorganism identified in Specimen by Culture"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260415000" codeSystem="2.16.840.1.113883.6.96" displayName="Not detected (qualifier value)">
<originalText>
<reference value="#OBS-MIBI-1-3"/> </originalText> </value> </observation> </component></organizer> | | | Beispiel | Keime (oder Mikroorganismen) nicht nachweisbar <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Erreger</th> <th>Methode</th> <th>Ergebnis / Keimzahl</th> </tr> </thead> <tbody>
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">Keime (oder Mikroorganismen)</td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">nicht nachgewiesen</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="264395009" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Microorganism (Organism)"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="11475-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Microorganism identified in Specimen by Culture"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260415000" codeSystem="2.16.840.1.113883.6.96" displayName="Not detected (qualifier value)">
<originalText>
<reference value="#OBS-MIBI-1-3"/> </originalText> </value> </observation> </component></organizer> | | | Beispiel | Koloniebildende Einheiten <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Erreger</th> <th>Methode</th> <th>Ergebnis / Keimzahl</th> </tr> </thead> <tbody>
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">Staphylococcus aureus</td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">>500KBE</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="3092008" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Staphylococcus aureus"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="11475-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Microorganism identified in Specimen by Culture"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="IVL_PQ">
<low value="500" unit="[CFU]"/> <high nullFlavor="PINF"/> </value> </observation> </component></organizer> |  |  | hl7:component
|
| | 0 … * | | Beinhaltet 1.2.40.0.34.6.0.11.3.19 Eingebettetes Objekt Entry (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:component
|
| | 0 … * | | Codierung des Kommentars zum kulturellen Erregernachweis bzw. zum Antibiogramm des nachgewiesenen Erregers.
Beinhaltet 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | | Beispiel | Kommentar zu kulurellem Erregernachweis und Antibiogramm <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Erreger</th> <th>Methode</th> <th>Ergebnis / Keimzahl</th> </tr> </thead> <tfoot>
<tr>
<td>
<footnote ID="isolateComment1">
<sup>1)</sup> Kommentar zum Erreger wie z.B., dass er meldepflichtig ist. </footnote> </td> </tr> </tfoot> <tbody>
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">
Staphylococcus epidermidis <sup>1)</sup> </td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">nachgewiesen</td> </tr> </tbody></table><paragraph styleCode="xELGA_h3">Antibiogramm</paragraph><table>
<thead>
<tr>
<th>Wirkstoff</th> <th>Staphylococcus epidermidis</th> </tr> </thead> <tfoot>
<tr>
<td>S = sensibel bei Standarddosierung, I = sensibel bei erhöhter Exposition, R = resistent, [] minimale Hemmkonzentration in mg/L</td> </tr> <tr>
<td>
<footnote ID="antibiogramComment1">
<sup>1)</sup> Kommentar zum Antibiogramm eines Erregers. </footnote> </td> </tr> </tfoot> <tbody>
<tr>
<td ID="OBS-AB-1">Penicillin</td> <td ID="OBS-AB-1-1">R</td> </tr> <tr>
<td ID="OBS-AB-2">Oxacillin</td> <td ID="OBS-AB-2-1">
S <sup>1)</sup> </td> </tr> <tr>
<td ID="OBS-AB-3">Erythromycin</td> <td ID="OBS-AB-3-1">S</td> </tr> <tr>
<td ID="OBS-AB-4">Clindamycin</td> <td ID="OBS-AB-4-1">S</td> </tr> </tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="60875001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Staphylococcus epidermidis"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<!-- Codierung von Methode und Keimzahl -->
</component> <!-- Antibiogramm -->
<component typeCode="COMP">
<!-- Codierung des Antibiogramms -->
</component> <!-- Codierung des Kommentars zum kulturellen Erregernachweis -->
<component typeCode="COMP">
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.11"/> <templateId root="2.16.840.1.113883.10.20.1.40"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.2"/> <code code="48767-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Annotation Comment"/> <text>
<reference value="#isolateComment1"/> </text> <statusCode code="completed"/> </act> </component> <!-- Codierung des Kommentars zum Antibiogramm -->
<component typeCode="COMP">
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.11"/> <templateId root="2.16.840.1.113883.10.20.1.40"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.2"/> <code code="48767-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Annotation Comment"/> <text>
<reference value="#antibiogramComment1"/> </text> <statusCode code="completed"/> </act> </component></organizer> |
|
14.4.5.15 Laboratory Battery Organizer
| Id | 1.2.40.0.34.6.0.11.3.26 ref at-cda-bbr- | Gültigkeit ab | 2023‑06‑01 13:50:16Andere Versionen mit dieser Id:  atcdabbr_entry_LaboratoryBatteryOrganizer vom 2019‑05‑29 10:51:34 atcdabbr_entry_LaboratoryBatteryOrganizer vom 2019‑05‑29 10:51:34
|
|---|
| Status |  Entwurf Entwurf | Versions-Label | 1.0.1 |
|---|
| Name | atcdabbr_entry_LaboratoryBatteryOrganizer | Bezeichnung | Laboratory Battery Organizer |
|---|
| Beschreibung | Der "Laboratory Battery Organizer" kann
- eine Befundgruppe innerhalb eines Befundbereiches ("Laboratory Specialty Section") darstellen. Für den "organizer/code" MUSS in diesem Fall ein Code aus dem Level 2 des hierarchischen Value Sets "ELGA_Laborstruktur" verwendet werden.
- ein Antibiogramm innerhalb des "Laboratory Isolate Organizer" darstellen. In diesem Fall MUSS für den "organizer/code" der Code "365705006 - Finding of antimicrobial susceptibility (finding)" angegeben werden.
|
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.26 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 4 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.24 | Containment |  Performer - Laboratory (1.0.0+20211213) Performer - Laboratory (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.27 | Containment |  Laboratory Observation (2.0.1) Laboratory Observation (2.0.1) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.19 | Containment |  Eingebettetes Objekt Entry (1.0.2+20230717) Eingebettetes Objekt Entry (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.11 | Containment |  Comment Entry (1.0.0+20210219) Comment Entry (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Version: Template 1.2.40.0.34.6.0.11.3.26 Laboratory Battery Organizer (2019‑05‑29 10:51:34) ref at-cda-bbr- |
|---|
| Beispiel | | Befundgruppe | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<!-- Befundgruppe "Blutbild" -->
<paragraph styleCode="xELGA_h3">Blutbild</paragraph><table>
<thead>
<tr>
<th>Analyse</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereiche</th> <th>Interpretation</th> </tr> </thead> <tbody>
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>+</td> </tr> <!-- Dokumentation weiterer Analyseergebnisse -->
</tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- Codierung der Befundgruppe "Blutbild" -->
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="03010" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Blutbild"/> <statusCode code="completed"/> <effectiveTime>
<low value="20190201081400+0100"/> <high value="20190201092200+0100"/> </effectiveTime> <component typeCode="COMP" contextConductionInd="true">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <id extension="OBS-1-1" root="1.2.40.0.34.99.4613.122082.1.3.5"/> <code code="26464-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Leukozyten"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime value="20191201073406+0100"/> <value unit="10*9/L" value="26" xsi:type="PQ"/> <interpretationCode code="H" codeSystemName="HL7:ObservationInterpretation" codeSystem="2.16.840.1.113883.5.83" displayName="High"/> <referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-1"/> </text> <value xsi:type="IVL_PQ">
<low value="4.0" unit="10*9/L"/> <high value="10.0" unit="10*9/L"/> </value> <interpretationCode code="N" codeSystemName="HL7:ObservationInterpretation" codeSystem="2.16.840.1.113883.5.83" displayName="normal"/> </observationRange> </referenceRange> </observation> <!-- Codierung weiterer Analyseergebnisse -->
</component></organizer> |
|
|---|
| Beispiel | | Antibiogramm | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<!-- Antibiogramm zum Erreger "Staphylococcus epidermidis" -->
<paragraph styleCode="xELGA_h3">Antibiogramm</paragraph><table>
<thead>
<tr>
<th>Wirkstoff</th> <th>Staphylococcus epidermidis</th> </tr> </thead> <tfoot>
<tr>
<td> S = sensibel bei Standarddosierung, I = sensibel bei erhöhter Exposition, R = resistent, [] minimale Hemmkonzentration in mg/L </td> </tr> </tfoot> <tbody>
<tr>
<td ID="OBS-AB-1">Penicillin</td> <td ID="OBS-AB-1-1">R</td> </tr> <!-- Dokumentation weiterer Antibiogrammergebnisse -->
</tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- Codierung des Antibiogramms -->
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="365705006" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Finding of antimicrobial susceptibility (finding)"/> <statusCode code="completed"/> <effectiveTime>
<low value="20190201081400+0100"/> <high value="20190201092200+0100"/> </effectiveTime> <component typeCode="COMP" contextConductionInd="true">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18964-7" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Penicillin [Susceptibility]">
<originalText>
<reference value="#OBS-AB-1"/> </originalText> </code> <text>
<reference value="#OBS-AB-1-1"/> </text> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" nullFlavor="UNK"/> <interpretationCode code="R" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Resistant"/> </observation> </component> <!-- ... -->
<!-- Codierung weiterer Antibiogrammergebnisse für "Staphylococcus epidermidis" -->
<!-- ... -->
</organizer> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | BATTERY |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Battery Organizer | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.26 |  | hl7:templateId
|
| II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.4.12 Laboratory Battery Organizer | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.1.4 | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:code[concat(@code, @codeSystem) = doc('include/voc-1.2.40.0.34.10.47-DYNAMIC.xml')//valueSet[1]/conceptList/concept/concat(@code, @codeSystem) or @nullFlavor]
- hl7:code[(@code = '365705006' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
|  |  | hl7:code
|
| CD | 0 … 1 | | Eindeutiger Code für die Befundgruppe als Teil eines Befundbereiches ("Laboratory Specialty Section"). | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | Constraint | Es MUSS ein Code aus Level 2 des hierarchischen Value Sets "ELGA_Laborstruktur" verwendet werden. | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.47 ELGA_Laborstruktur (DYNAMIC) |
|  |  | hl7:code
|
| CD | 0 … 1 | | Eindeutiger Code für ein Antibiogramm als Teil des Templates "Laboratory Isolate Organizer". | | | st | 0 … 1 | F | SNOMED CT | | CONF | 0 … 1 | F | 365705006 | | 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) | | 0 … 1 | F | Finding of antimicrobial susceptibility (finding) | | | Schematron assert | role | error | | | | test | hl7:code[not(@nullFlavor)] | | | | Meldung | @nullFlavor ist für organizer/code NICHT erlaubt. | |  | hl7:statusCode
|
| CS | 1 … 1 | M | Der "statusCode" kann folgende Werte annehmen:
-
completed: zu verwenden, wenn alle erwarteten Analyseergebnisse für diesen Organizer abgeschlossen sind.
-
aborted: zu verwenden, wenn zumindest ein Analyseergebnis für diesen Organizer nicht abgeschlossen werden konnte (abgebrochen wurde).
| | | | CONF | | @code muss "completed" sein | | oder | | @code muss "aborted" sein |
|  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | Fertigstellungszeitpunkt der enthaltenen Tests.
| |  |  | hl7:low
|
| TS.AT.TZ | 1 … 1 | M | | |  |  | hl7:high
|
| TS.AT.TZ | 1 … 1 | M | | |  | hl7:performer
|
| | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).
Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. | | Auswahl | 0 … * | | Elemente in der Auswahl:- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.19 Eingebettetes Objekt Entry (DYNAMIC)
- hl7:component welches enthält Template 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC)
|  |  | hl7:component
|
| | 0 … * | |
- Im Fall einer Befundgruppe werden in "Laboratory Observations" die einzelnen Analyseergebnisse codiert.
- Im Fall eines Antibiogramms werden in "Laboratory Observations" die Testergebnisse einzelner Antibiotika codiert.
Beinhaltet 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:component
|
| | 0 … * | | Beinhaltet 1.2.40.0.34.6.0.11.3.19 Eingebettetes Objekt Entry (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true |  |  | hl7:component
|
| | 0 … * | | Codierung des Kommentars für diesen "Laboratory Battery Organizer".
Beinhaltet 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC) | | | cs | 1 … 1 | F | COMP | | cs | 0 … 1 | F | true | | | Beispiel | Kommentar zur Befundgruppe <!-- Befundbereich -->
<section>
<templateId root="1.2.40.0.34.6.0.11.2.102"/> <id extension="P-body" root="2.16.840.1.113883.3.933.1.1"/> <code code="300" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Hämatologie"/> <title>Hämatologie</title> <!-- CDA Level 2 -->
<text>
<!-- Befundgruppe "Blutbild" -->
<paragraph styleCode="xELGA_h3">Blutbild</paragraph> <table>
<thead>
<tr>
<th>Analyse</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereiche</th> <th>Interpretation</th> </tr> </thead> <tbody>
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>+</td> </tr> <!-- ... -->
</tbody> </table> <!-- Kommentar zur Befundgruppe "Blutbild" -->
<paragraph>
<content ID="blutbildComment">Das ist ein Kommentar zur Befundgruppe "Blutbild".</content> </paragraph> <!-- ... -->
<!-- weitere Befundgruppen -->
<!-- ... -->
</text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<templateId root="1.2.40.0.34.6.0.11.3.25"/> <templateId root="1.3.6.1.4.1.19376.1.3.1"/> <act classCode="ACT" moodCode="EVN">
<code code="300" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Hämatologie"/> <statusCode code="completed"/> <entryRelationship typeCode="COMP">
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.26"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.4"/> <code code="03010" codeSystem="1.2.40.0.34.5.11" codeSystemName="ELGA_LaborparameterErgaenzung" displayName="Blutbild"/> <statusCode code="completed"/> <!-- ... -->
<!-- Codierung der Analyseergebnisse -->
<!-- ... -->
<!-- Codierung des Kommentars zur Befundgruppe -->
<component typeCode="COMP">
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.11"/> <templateId root="2.16.840.1.113883.10.20.1.40"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.2"/> <code code="48767-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Annotation Comment"/> <text>
<reference value="#blutbildComment"/> </text> <statusCode code="completed"/> </act> </component> </organizer> </entryRelationship> </act> </entry></section> |
|
14.4.5.16 Laboratory Observation
| Id | 1.2.40.0.34.6.0.11.3.27 ref at-cda-bbr- | Gültigkeit ab | 2023‑06‑01 13:58:20Andere Versionen mit dieser Id:  atcdabbr_entry_LaboratoryObservation vom 2021‑04‑19 16:15:55 atcdabbr_entry_LaboratoryObservation vom 2021‑04‑19 16:15:55 atcdabbr_entry_LaboratoryObservation vom 2019‑05‑07 13:44:02 atcdabbr_entry_LaboratoryObservation vom 2019‑05‑07 13:44:02
|
|---|
| Status |  Entwurf Entwurf | Versions-Label | 2.0.1 |
|---|
| Name | atcdabbr_entry_LaboratoryObservation | Bezeichnung | Laboratory Observation |
|---|
| Beschreibung | Ergebnis einer Analyse (Laboruntersuchung, Test) wird als "observation" codiert. Jede "observation" stellt das Ergebnis genau einer Analyse dar. Die "observation" kann
- als Einzelanalyse direkt unter dem Specimen-Act (siehe "Laboratory Report Data Processing Entry");
- als Ergebnis eines kulturellen Erregernachweises unter dem "Laboratory Isolate Organizer";
- als Teil einer Befundgruppe bzw. eines Antibiogramms (siehe "Laboratory Battery Organizer");
vorkommen.
|
|
| Kontext | Elternknoten des Template-Element mit Id 1.2.40.0.34.6.0.11.3.27 |
|---|
| Klassifikation | CDA Entry Level Template |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 5 Templates | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.1 | Inklusion |  Narrative Text Reference (1.0.1+20210512) Narrative Text Reference (1.0.1+20210512) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.23 | Inklusion |  Laboratory Observation Value (1.0.0+20211213) Laboratory Observation Value (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.24 | Containment |  Performer - Laboratory (1.0.0+20211213) Performer - Laboratory (1.0.0+20211213) | DYNAMIC | | 1.2.40.0.34.6.0.11.9.10 | Containment |  Address Compilation Minimal (1.0.2+20230717) Address Compilation Minimal (1.0.2+20230717) | DYNAMIC | | 1.2.40.0.34.6.0.11.3.11 | Containment |  Comment Entry (1.0.0+20210219) Comment Entry (1.0.0+20210219) | DYNAMIC |
|
|
|---|
| Beziehung | Version: Template 1.2.40.0.34.6.0.11.3.27 Laboratory Observation (2021‑04‑19 16:15:55) ref at-cda-bbr-
Version: Template 1.2.40.0.34.6.0.11.3.27 Laboratory Observation Entry (2019‑05‑07 13:44:02) ref at-cda-bbr- |
|---|
| Beispiel | | Analyse (Laboruntersuchung) | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>+</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <id root="1.2.3.999" extension="--example only--"/> <code code="26464-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Leukozyten"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime value="20190201092200+0100"/> <value unit="10*9/L" value="26.0" xsi:type="PQ"/> <interpretationCode code="H" codeSystem="2.16.840.1.113883.5.83" displayName="High"/> <referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-1"/> </text> <value xsi:type="IVL_PQ">
<low value="4" unit="10*9/L"/> <high value="10" unit="10*9/L" inclusive="false"/> </value> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83"/> </observationRange> </referenceRange></observation> |
|
|---|
| Beispiel | | Kultureller Erregernachweis | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-MIBI-1">
<td ID="OBS-MIBI-1-1">Staphylococcus epidermidis</td> <td ID="OBS-MIBI-1-2">Kultur</td> <td ID="OBS-MIBI-1-3">nachgewiesen</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<!-- "Laboratory Isolate Organizer" für Keim "Staphylococcus epidermidis" -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.167"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.5"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <specimen typeCode="SPC">
<specimenRole classCode="SPEC">
<specimenPlayingEntity classCode="MIC">
<code code="60875001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Staphylococcus epidermidis"/> </specimenPlayingEntity> </specimenRole> </specimen> <!-- Laboratory Observation für Methode und Ergebnis / Keimzahl -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="11475-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Microorganism identified in Specimen by Culture">
<originalText>
<reference value="#OBS-MIBI-1-2"/> </originalText> </code> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" displayName="Detected (qualifier value)">
<originalText>
<reference value="#OBS-MIBI-1-3"/> </originalText> </value> </observation> </component></organizer> |
|
|---|
| Beispiel | | Zu wenig Material | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1">
<td>HIV AK/Ag</td> <td ID="OBS-1-1-result">zu wenig Material</td> <td/> <td/> <td/></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <id root="1.2.3.999" extension="--example only--"/> <code code="56888-1" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="HIV 1+2 Ab+HIV1 p24 Ag [Presence] in Serum or Plasma by Immunoassay"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime value="UNK"/> <value xsi:type="CD" code="281268007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Insufficient sample (finding)">
<translation>
<originalText>
<reference value="#OBS-1-1-result"/> </originalText> </translation> </value></observation><!-- ... -->
|
|
|---|
| Beispiel | | Analyse in Arbeit ('Wert folgt') | <ClinicalDocument>
<!-- CDA Header -->
<!-- ... -->
<!-- Angabe des Status "active", um anzuzeigen, dass Ergebnisse einzelner Analysen noch ausständig sind -->
<sdtc:statusCode code="active"/> <!-- ... -->
<!-- CDA Body -->
<component typeCode="COMP">
<structuredBody classCode="DOCBODY">
<component typeCode="COMP">
<section classCode="DOCSECT">
<!-- ... -->
<text>
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1">
<td>Leukozyten</td> <td ID="OBS-1-1-result">Wert folgt</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td/> </tr> <!-- ... -->
</text> <!-- CDA Level 3 -->
<entry typeCode="DRIV">
<!-- ... -->
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <id root="1.2.3.999" extension="--example only--"/> <code code="26464-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Leukozyten"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime value="UNK"/> <value xsi:type="CD" code="255599008" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Incomplete (qualifier value)">
<translation>
<originalText>
<reference value="#OBS-1-1-result"/> </originalText> </translation> </value> </observation> <!-- ... -->
</entry> </section> </component> </structuredBody> </component></ClinicalDocument> |
|
|---|
| Beispiel | | Antibiogrammergebnisse | <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr>
<td ID="OBS-AB-1">Tigecyclin</td> <td ID="OBS-AB-1-1">S [0.25]</td></tr><tr>
<td ID="OBS-AB-2">Penicillin</td> <td ID="OBS-AB-2-1">R</td></tr><tr>
<td ID="OBS-AB-3">Doxycyclin</td> <td ID="OBS-AB-3-1">[0.25]</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="42357-4" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Tigecycline [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" value="0.25" unit="mg/L"/> <interpretationCode code="S" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Susceptible"/> </observation></component><component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18964-7" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Penicillin [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" nullFlavor="UNK"/> <interpretationCode code="R" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Resistant"/> </observation></component><component typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="18917-5" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Doxycycline [Susceptibility]"/> <statusCode code="completed"/> <effectiveTime nullFlavor="UNK"/> <value xsi:type="PQ" value="0.25" unit="mg/L"/> </observation></component> |
|
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| | | | | |  | @classCode
|
| cs | 1 … 1 | F | OBS |  | @moodCode
|
| cs | 1 … 1 | F | EVN |  | hl7:templateId
|
| II | 1 … 1 | M | Laboratory Observation | |  |  | @root
|
| uid | 1 … 1 | F | 1.2.40.0.34.6.0.11.3.27 |  | hl7:templateId
|
| II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.4.13 Laboratory Observation | |  |  | @root
|
| uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.1.6 |  | hl7:id
|
| II | 0 … 1 | | Eindeutige ID der Analyse auf Basis eines lokalen Nummernkreises.
| | | Auswahl | 1 … 1 | | Für die Codierung der Analyse bzw. des Antibiotikums, das im Rahmen eines Antibiogramms getestet wurde, SOLLEN grundsätzlich die Werte aus den Value Sets "ELGA_Laborparameter" bzw. "ELGA_Antibiogramm_VS" verwendet werden.
Über "code/translation" ist es möglich, weitere Codes anzugeben, die die Analyse noch präziser beschreiben. Hier kann z.B. ein methodenspezifischer LOINC angegeben werden. LOINC-Codes sind in diesem Zusammenhang bevorzugt anzugeben - es können aber auch andere angegeben werden.
Sollte im Value Set "ELGA_Laborparameter" oder "ELGA_Antibiogramm_VS" kein Code für die Analyse bzw. das getestete Antibiotikum verfügbar sein, kann diese dennoch maschinenlesbar hinterlegt werden mit der Kennzeichnung, dass der Wert nicht aus dem Value Set stammt. Das gilt auch für den Fall, dass ein passender LOINC-Code exisitiert, aber nicht im aktuellen Value Set "ELGA_Laborparameter" oder "ELGA_Antibiogramm_VS" enthalten ist. Es wird gebeten, dass benötigte Codes umgehend an ELGA gemeldet werden, um diese im Zuge von Reviewzyklen in die Codelisten und Value Sets einpflegen zu können. Für die Befunddarstellung solcher Analysen wird empfohlen, dass diese jeweils am Ende der thematisch passendsten Gruppe einzureihen sind, solange es noch keine andere Vorgabe oder Empfehlung gibt. - hl7:code[not(@nullFlavor)]
- hl7:code[@nullFlavor='OTH']
|  |  | hl7:code
|
| CD | 0 … 1 | | Codierung der Analyse bzw. des Antibiotikums, das im Rahmen eines Antibiogramms getestet wurde.
| | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.44 ELGA_Laborparameter (DYNAMIC) | | oder | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.53 ELGA_Antibiogramm_VS (DYNAMIC) |
| | | Beispiel | Codierung der Analyse <code code="26464-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Leukozyten"/> | | | Beispiel | Angabe von Translations zur Präzisierung der Analyse <code code="6584-7" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Virus identified in Specimen by Culture">
<translation code="9782-4" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Adenovirus sp identified in Specimen by Organism specific culture"/> <translation code="122268003" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Adenovirus species culture (procedure)"/></code> | | | Beispiel | Codierung des Antibiotikums <code code="18961-3" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Oxacillin [Susceptibility]"/> |  |  | hl7:code
|
| CD | 0 … 1 | | Codierung von Codes, die nicht im aktuellen Value Set "ELGA_Laborparameter" oder "ELGA_Antibiogramm_VS" enthalten sind.
| | | wo [@nullFlavor='OTH'] | | | | Beispiel | Codierung einer Analyse ohne passenden Code <code nullFlavor="OTH">
<translation code="alternativerCode" codeSystem="alternativeCodeSystem" codeSystemName="NameDesCodeSystems" displayName="klarTextDarstellung"/></code> | | | Schematron assert | role | error | | | | test | hl7:translation[not(@nullFlavor)] | | | | Meldung | Wenn code[@nullFlavor='OTH'] dann MUSS "code/translation" anwesend sein. | | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.1 Narrative Text Reference (DYNAMIC)
Das "text"-Element wird verwendet, um einen Verweis zum narrativen Text ("section/text") der Analyse herzustellen.
|  | hl7:text
|
| ED | 1 … 1 | M | | |  |  | hl7:reference
|
| TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im menschenlesbaren Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts mit Zusatzinformationen umschließen.
Alternativ kann @value auch mit dem url-scheme "http" oder "https" beginnen.
| | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') or starts-with(@value,'http') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element, or begin with the 'http' or 'https' url-scheme. | |  | hl7:statusCode
|
| CS | 1 … 1 | M | Der "statusCode" kann folgende Werte annehmen:
-
completed: zu verwenden, wenn die Analyse abgeschlossen ist und das Ergebnis vorliegt.
-
aborted: zu verwenden, wenn die Analyse nicht durchgeführt werden konnte (abgebrochen wurde).
| | | | CONF | | @code muss "completed" sein | | oder | | @code muss "aborted" sein |
| | Auswahl | 1 … 1 | | Medizinisch relevantes Datum und Zeit. In der Regel Abnahmedatum/-zeit des Untersuchungsmaterials. Elemente in der Auswahl:- hl7:effectiveTime[not(@nullFlavor)]
- hl7:effectiveTime[@nullFlavor='UNK']
|  |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | | | | wo [not(@nullFlavor)] | |  |  | hl7:effectiveTime
|
| IVL_TS | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | Eingefügt | 0 … 1 | C | von 1.2.40.0.34.6.0.11.9.23 Laboratory Observation Value (DYNAMIC) | | | Constraint | Kann bei stornierten (observation/statusCode[@code='aborted']) Analysen entfallen. | | Auswahl | 0 … 1 | | Codiertes Ergebnis im entsprechenden Datentyp.
Numerische Ergebnisse werden in der Regel als "physical quantity" PQ dargestellt, was die Angabe einer UCUM codierten Einheit erforderlich macht. Es MUSS die "case sensitive" Variante (c/s) der maschinenlesbaren Form des UCUM verwendet werden. Die bevorzugte Einheit für jede Analyse wird im Value Set "ELGA_Laborparameter" vorgeschlagen, jeweils in der "print" Variante (für die Darstellung) und in der maschinenlesbaren Form.
Es wird EMPFOHLEN, anstelle von Einheitenpräfixen ("Giga", "Mega", "Milli", "Mikro" etc.) eine Potenzschreibweise zu wählen, vor allem, wenn die Groß/Kleinschreibung eine Rolle spielt und Verwechslungen möglich sind (z.B. "G/L"=Giga pro Liter vs. "g/L"=Gramm/Liter). Also "10^6" statt "M" (Mega), "10^9" statt "G" (Giga) usw.
Unterelemente können je nach Datentyp notwendig sein, z.B. high/low für IVL oder numerator/denominator für RTO.
Elemente in der Auswahl:- hl7:value[@xsi:type='PQ']
- hl7:value[@xsi:type='IVL_PQ']
- hl7:value[@xsi:type='INT']
- hl7:value[@xsi:type='IVL_INT']
- hl7:value[@xsi:type='BL']
- hl7:value[@xsi:type='ST']
- hl7:value[@xsi:type='CV']
- hl7:value[concat(@code, @codeSystem) = doc('include/voc-1.2.40.0.34.10.186-DYNAMIC.xml')//valueSet[1]/conceptList/concept/concat(@code, @codeSystem) or @nullFlavor]
- hl7:value[(@code = '281268007' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
- hl7:value[(@code = '255599008' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
- hl7:value[@xsi:type='CD']
- hl7:value[@xsi:type='RTO']
- hl7:value[@xsi:type='RTO_PQ_PQ']
|  |  | hl7:value
|
| PQ | 0 … 1 | | Codierung einer physikalischen Größe | | | wo [@xsi:type='PQ'] | | | | Beispiel | Codierung eines numerischen Ergebnisses <value xsi:type="PQ" value="49.7" unit="%"/> | | | Beispiel | Codierung von numerischen, dimensionslosen Ergebnissen <!-- Für dimensionslose Einheiten wird in UCUM häufig eine Einheit angegeben, wie z.B. "[pH]" für den pH-Wert. -->
<value xsi:type="PQ" value="7" unit="[pH]"/> | | | Beispiel | Codierung von numerischen, dimensionslosen Ergebnissen <!-- Wenn keine UCUM-Einheit vorgeschlagen ist, können dimensionslose Einheiten auch mit @unit="1" dargestellt werden, hier für INR. -->
<value xsi:type="PQ" value="1.1" unit="1"/> | | PQR | 0 … 1 | | Alternative Repräsentation derselben physikalischen Größe, mit unterschiedlicher Einheit in @code und @codeSystem und einem möglicherweise unterschiedlichen Wert. | |  |  | hl7:value
|
| IVL_PQ | 0 … 1 | | Codierung eines Intervalls von physikalischen Größen | | | wo [@xsi:type='IVL_PQ'] | | | | Beispiel | Codierung eines Intervalls <!-- Angabe von 20 - 30 mg/dl -->
<!-- @inclusive gibt mit true/false an, ob die Intervallgrenze im Intervall enthalten ist oder nicht (offenes oder geschlossenes Intervall). -->
<value xsi:type="IVL_PQ">
<low value="20" unit="mg/dl" inclusive="true"/> <high value="30" unit="mg/dl" inclusive="true"/></value> |  |  | hl7:value
|
| INT | 0 … 1 | | Codierung eines numerischen Ergebnisses. | | | wo [@xsi:type='INT'] | |  |  | hl7:value
|
| IVL_INT | 0 … 1 | | Codierung eines numerischen Intervalls. | | | wo [@xsi:type='IVL_INT'] | |  |  | hl7:value
|
| BL | 0 … 1 | | Codierung eines bool'schen Ergebnisses. | | | wo [@xsi:type='BL'] | |  |  | hl7:value
|
| ST | 0 … 1 | | Codierung eines textuellen Ergebnisses. Die Angabe des Ergebnisses erfolgt hier als Wert des Elements.
Im narrativen Block MUSS derselbe Text wie im Entry dargestellt werden. | | | wo [@xsi:type='ST'] | | | | Beispiel | Codierung eines textuellen Ergebnisses <value xsi:type="ST">strohgelb</value> |  |  | hl7:value
|
| CV | 0 … 1 | | Codierung des Ergebnisses in Form eines Codes. | | | wo [@xsi:type='CV'] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | |  |  | hl7:value
|
| CD | 0 … 1 | | Wenn ein Ergebnis vorliegt, das durch das Value Set "ELGA_NachweisErgebnis_VS" abgedeckt werden kann, MUSS ein Wert aus diesem verwendet werden.
Das Value Set definiert auch, wie die Darstellung in CDA Level 2 als Semigrafik erfolgen kann (siehe Beispiel). | | | cs | 1 … 1 | F | CD | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.186 ELGA_NachweisErgebnis_VS (DYNAMIC) |
| | | Beispiel | Codierung, wenn ein Erreger nachgewiesen werden konnte. <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<td>nachgewiesen</td><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Detected (qualifier value)"/> | | | Beispiel | Codierung und Darstellung mittels Semigrafik <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<td>+++</td><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<value xsi:type="CD" code="260354000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Moderate number (qualifier value)"/> |  |  | hl7:value
|
| CD | 0 … 1 | | Codierung, dass nicht ausreichend Material für die Durchführung der Analyse vorgelegen ist. | | | cs | 1 … 1 | F | CD | | st | 0 … 1 | F | SNOMED CT | | CONF | 0 … 1 | F | 281268007 | | 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) | | 0 … 1 | F | Insufficient sample (finding) | | | Beispiel | Zu wenig Material <value xsi:type="CD" code="281268007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Insufficient sample (finding)"/> |  |  | hl7:value
|
| CD | 0 … 1 | | Codierung, dass das Ergebnis der Analyse noch ausstehend bzw. noch in Arbeit ist. | | | cs | 1 … 1 | F | CD | | st | 0 … 1 | F | SNOMED CT | | CONF | 0 … 1 | F | 255599008 | | 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) | | 0 … 1 | F | Incomplete (qualifier value) | | | Beispiel | Ausstehendes Ergebnis ('Wert folgt') <value xsi:type="CD" code="255599008" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Incomplete (qualifier value)"/> |  |  | hl7:value
|
| CD | 0 … 1 | | Codierung des Ergebnisses in Form eines Codes. | | | wo [@xsi:type='CD'] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | |  |  | hl7:value
|
| RTO | 0 … 1 | | Codierung einer Verhältnisangabe (z.B. Titer). | | | wo [@xsi:type='RTO'] | | | | Beispiel | Verhältnisangabe für Titer (z.B. '1:125') <value xsi:type="RTO">
<numerator value="1" xsi:type="INT"/> <denominator value="128" xsi:type="INT"/></value> |  |  | hl7:value
|
| RTO_PQ_PQ | 0 … 1 | | Codierung einer Verhältnisangabe von physikalischen Größen. | | | wo [@xsi:type='RTO_PQ_PQ'] | | | | Schematron assert | role | error | | | | test | hl7:value or hl7:statusCode[@code='aborted'] | | | | Meldung | Das "value"-Element darf nur im Fall von stornierten Analysen (observation/statusCode[@code='aborted']) entfallen. | | | | Schematron assert | role | error | | | | test | not(hl7:value[@nullFlavor]) or (hl7:value[@xsi:type='PQ'][@nullFlavor='UNK'] and (hl7:interpretationCode[not(@nullFlavor)] or hl7:interpretationCode[@nullFlavor='OTH']) and ../../hl7:code[@code='365705006'][@codeSystem='2.16.840.1.113883.6.96']) | | | | Meldung | Für Antibiogrammergebnisse kann value[@nullFlavor='UNK'] verwendet werden, wenn interpretationCode oder
interpretationCode[@nullFlavor='OTH'] vorhanden ist. Ansonsten ist die Verwendung von value[@nullFlavor] NICHT ERLAUBT. | | | | Schematron assert | role | error | | | | test | not(hl7:value[@code='255599008'][@codeSystem='2.16.840.1.113883.6.96']) or /hl7:ClinicalDocument/sdtc:statusCode[@code='active'] | | | | Meldung | Wenn eine Analyse als "in Arbeit" (SCT "255599008 - Incomplete (qualifier value)") markiert ist, MUSS der gesamte Befund als aktiv (/ClinicalDocument/sdtc:statusCode[@code='active']) gekennzeichnet werden. | | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:interpretationCode[not(@nullFlavor)]
- hl7:interpretationCode[@nullFlavor='OTH']
|  |  | hl7:interpretationCode
|
| CE | 0 … 1 | | Codierte Bewertung des Ergebnisses. Wird für Referenzbereichbewertungen, für die Codierung der RAST-Klassen sowie für die Codierung von Antibiogrammergebnissen verwendet.
Basierend auf dem Auszug aus dem Value Set "ELGA_ObservationInterpretation" stellt die folgende Tabelle dar, wie eine Darstellung in CDA Level 2, in Abhängigkeit von den CDA Level 3 codierten Interpretationen, aussehen kann.
<TBODY>
| Darstellung CDA Level 2 |
Codierung CDA Level 3 |
Beschreibung |
| Befundinterpretation für numerische Ergebnisse |
| ++ |
HH |
Oberhalb des Referenzbereiches und über einer oberen Warngrenze |
| + |
H |
Oberhalb des Referenzbereiches |
|
N |
Normal (innerhalb des Referenzbereiches) |
| - |
L |
Unterhalb des Referenzbereiches |
| -- |
LL |
Unterhalb des Referenzbereiches und unter einer unteren Warngrenze |
| Befundinterpretation für nicht numerische Ergebnisse |
|
N |
Normal (innerhalb des Referenzbereiches) |
| * |
A |
Abnormal |
| ** |
AA |
Abnormal Warngrenze |
</TBODY>
| | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.13 ELGA_ObservationInterpretation (DYNAMIC) |
| | | Beispiel | Bewertung eines numerischen Ergebnisses <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>+</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<interpretationCode code="H" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="High"/> | | | Beispiel | Bewertung eines nicht-numerischen Ergebnisses <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>Leukozyten</td> <td>26</td> <td>10^9/L</td> <td ID="OBSREF-1-1">4-10</td> <td>*</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<interpretationCode code="A" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Abnormal"/> | | | Beispiel | Interpretation eines Antibiogramms <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr>
<td>Tigecyclin</td> <td>R</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<interpretationCode code="R" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Resistant"/> |  |  | hl7:interpretationCode
|
| CE | 0 … 1 | | Codierte Bewertung des Ergebnisses mit einem Code, der aktuell nicht im Value Set "ELGA_ObservationInterpretation" enthalten ist.
| | | wo [@nullFlavor='OTH'] | | | | Schematron assert | role | error | | | | test | hl7:translation[not(@nullFlavor)] | | | | Meldung | Wenn interpretationCode[@nullFlavor='OTH'] dann MUSS "interpretationCode/translation" anwesend sein. | | | | Schematron assert | role | error | | | | test | not(hl7:referenceRange) or hl7:interpretationCode | | | | Meldung | Wenn zu einer Analyse ein Referenzbereich mit "observation/referenceRange" angeführt wird, MUSS auch eine Befundinterpretation in "observation/interpretationCode" erfolgen. | |  | hl7:performer
|
| | 0 … * | C | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter) - z.B. externes Labor.
Beinhaltet 1.2.40.0.34.6.0.11.9.24 Performer - Laboratory (DYNAMIC) | | | | Constraint | Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf[1]/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Analyse ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene).
Für den Fall, dass Analysen von einem externen Labor durchgeführt wurden, MUSS assignedEntity/code mit @code="E", @codeSystem="2.16.840.1.113883.2.16.1.4.9", @codeSystemName="HL7.at.Laborkennzeichnung" und @displayName="EXTERN" angegeben werden. |  | hl7:participant
|
| | 0 … 1 | | Validierende Person.
| |  |  | @typeCode
|
| cs | 1 … 1 | F | AUTHEN |  |  | @contextControlCode
|
| cs | 0 … 1 | F | OP | | | Constraint | Wird zu eine Analyse eine validierende Person gelistet, ist diese auch im Header als "authenticator" anzuführen. | | | Beispiel | Codierung der validierenden Person <participant typeCode="AUTHEN">
<templateId root="1.3.6.1.4.1.19376.1.3.3.1.5"/> <time value="20210123211000+0100"/> <participantRole>
<id extension="9999" root="1.2.3.999"/> <addr nullFlavor="UNK"/> <telecom value="tel:312.555.5555"/> <playingEntity>
<name>Susanne Hecht</name> </playingEntity> </participantRole></participant> |  |  | hl7:templateId
|
| II | 1 … 1 | M | IHE PalM TF3 Rev.10, 6.3.2.16 Laboratory Results Validator | | | uid | 1 … 1 | F | 1.3.6.1.4.1.19376.1.3.3.1.5 |  |  | hl7:time
|
| IVL_TS | 1 … 1 | M | | |  |  | hl7:participantRole
|
| | 1 … 1 | M | | | | cs | 0 … 1 | F | ROL | | II | 1 … 1 | M | | | | Auswahl | 1 … 1 | | Elemente in der Auswahl:- hl7:addr[not(@nullFlavor)] welches enthält Template 1.2.40.0.34.6.0.11.9.10 Address Compilation Minimal (DYNAMIC)
- hl7:addr[@nullFlavor='UNK']
| | AD | 0 … 1 | | Beinhaltet 1.2.40.0.34.6.0.11.9.10 Address Compilation Minimal (DYNAMIC) | | | wo [not(@nullFlavor)] | | | AD | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | Auswahl | 1 … * | | Elemente in der Auswahl:- hl7:telecom[not(@nullFlavor)]
- hl7:telecom[@nullFlavor='UNK']
| | TEL.AT | 0 … * | | | | | wo [not(@nullFlavor)] | | | st | 1 … 1 | R |
Formatkonvention siehe "telecom – Format Konventionen für Telekom-Daten"
Zulässige Werteliste für telecom-Präfixe gemäß "ELGA_URLScheme"
| | set_cs | 0 … 1 | |
Bedeutung des angegebenen Kontakts (Heim, Arbeitsplatz, …), z.B. WP
Zulässige Werte gemäß Value-Set "ELGA_TelecomAddressUse"
| | | Constraint | Werden mehrere gleichartige "telecom"-Elemente strukturiert, MUSS jeweils das Attribut @use angeführt sein.
| | TEL.AT | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | | 1 … 1 | M | | | | cs | 0 … 1 | F | ENT | | cs | 0 … 1 | F | INSTANCE | | PN | 1 … 1 | M | | |  | hl7:entryRelationship
|
| | 0 … * | | Beinhaltet 1.2.40.0.34.6.0.11.3.11 Comment Entry (DYNAMIC) | |  |  | @typeCode
|
| cs | 1 … 1 | F | COMP |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true | | | Beispiel | Kommentar zur Analyse <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<table>
<thead>
<tr>
<th>Analyse</th> <th>Ergebnis</th> <th>Einheit</th> <th>Referenzbereiche</th> <th>Interpretation</th> </tr> </thead> <tfoot>
<tr>
<td>
<footnote ID="fn1">
<sup>1)</sup> INR nur gültig bei oraler Antikoagulation </footnote> </td> </tr> </tfoot> <tbody>
<!-- ... -->
<tr ID="OBS-1-1" styleCode="xELGA_red">
<td>INR</td> <td>
1.0 <sup>1)</sup> </td> <td/> <td ID="OBSREF-1-1">2.0-3.5</td> <td>-</td> </tr> <!-- ... -->
</tbody></table><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.27"/> <templateId root="1.3.6.1.4.1.19376.1.3.1.6"/> <code code="6301-6" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="INR"/> <text>
<reference value="#OBS-1-1"/> </text> <statusCode code="completed"/> <effectiveTime value="20161201073406+0100"/> <value xsi:type="PQ" value="1.0" unit="1"/> <interpretationCode code="L" codeSystemName="HL7:ObservationInterpretation" codeSystem="2.16.840.1.113883.5.83" displayName="Low"/> <!-- Codierter Kommentar zur Analyse -->
<entryRelationship typeCode="COMP">
<act classCode="ACT" moodCode="EVN">
<templateId root="1.2.40.0.34.6.0.11.3.11"/> <templateId root="2.16.840.1.113883.10.20.1.40"/> <templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.2"/> <code code="48767-8" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC" displayName="Annotation Comment"/> <text>
<reference value="#fn1"/> </text> <statusCode code="completed"/> </act> </entryRelationship> <referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-1"/> </text> <value xsi:type="IVL_PQ">
<low value="2.0" unit="1" inclusive="true"/> <high value="3.5" unit="1" inclusive="true"/> </value> <interpretationCode code="N" codeSystemName="HL7:ObservationInterpretation" codeSystem="2.16.840.1.113883.5.83" displayName="normal"/> </observationRange> </referenceRange></observation> |  | hl7:entryRelationship
|
| | 0 … * | | Angabe von früheren Ergebnissen für denselben Patient, dieselbe Analyse (Test + Methode), in derselben Einheit. | |  |  | @typeCode
|
| cs | 1 … 1 | F | REFR |  |  | @contextConductionInd
|
| cs | 0 … 1 | F | true |  |  | hl7:observation
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | OBS | | cs | 1 … 1 | F | EVN | | | Constraint |
Der hier angegebene Code MUSS derselbe sein wie in observation/code.
| | Auswahl | 1 … 1 | | Für die Codierung der Analyse bzw. des Antibiotikums.
- hl7:code[not(@nullFlavor)]
- hl7:code[@nullFlavor='OTH']
| | CD | 0 … 1 | | Codierung der Analyse bzw. des Antibiotikums, das im Rahmen eines Antibiogramms getestet wurde.
| | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.44 ELGA_Laborparameter (DYNAMIC) | | oder | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.6.0.10.53 ELGA_Antibiogramm_VS (DYNAMIC) |
| | CD | 0 … 1 | | Codierung von Codes, die nicht im aktuellen Value Set "ELGA_Laborparameter" oder "ELGA_Antibiogramm_VS" enthalten sind.
| | | wo [@nullFlavor='OTH'] | | | | Schematron assert | role | error | | | | test | hl7:translation[not(@nullFlavor)] | | | | Meldung | Wenn code[@nullFlavor='OTH'] dann MUSS "code/translation" anwesend sein. | | | CS | 1 … 1 | M | | | | CONF | 1 … 1 | F | completed | | Auswahl | 1 … 1 | | Medizinisch relevantes Datum und Zeit. In der Regel Abnahmedatum/-zeit des Untersuchungsmaterials. Elemente in der Auswahl:- hl7:effectiveTime[not(@nullFlavor)]
- hl7:effectiveTime[@nullFlavor='UNK']
| | IVL_TS | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | IVL_TS | 0 … 1 | | | | | wo [@nullFlavor='UNK'] | | | Eingefügt | 1 … 1 | R | von 1.2.40.0.34.6.0.11.9.23 Laboratory Observation Value (DYNAMIC) | | Auswahl | 1 … 1 | | Codiertes Ergebnis im entsprechenden Datentyp.
Numerische Ergebnisse werden in der Regel als "physical quantity" PQ dargestellt, was die Angabe einer UCUM codierten Einheit erforderlich macht. Es MUSS die "case sensitive" Variante (c/s) der maschinenlesbaren Form des UCUM verwendet werden. Die bevorzugte Einheit für jede Analyse wird im Value Set "ELGA_Laborparameter" vorgeschlagen, jeweils in der "print" Variante (für die Darstellung) und in der maschinenlesbaren Form.
Es wird EMPFOHLEN, anstelle von Einheitenpräfixen ("Giga", "Mega", "Milli", "Mikro" etc.) eine Potenzschreibweise zu wählen, vor allem, wenn die Groß/Kleinschreibung eine Rolle spielt und Verwechslungen möglich sind (z.B. "G/L"=Giga pro Liter vs. "g/L"=Gramm/Liter). Also "10^6" statt "M" (Mega), "10^9" statt "G" (Giga) usw.
Unterelemente können je nach Datentyp notwendig sein, z.B. high/low für IVL oder numerator/denominator für RTO.
Elemente in der Auswahl:- hl7:value[@xsi:type='PQ']
- hl7:value[@xsi:type='IVL_PQ']
- hl7:value[@xsi:type='INT']
- hl7:value[@xsi:type='IVL_INT']
- hl7:value[@xsi:type='BL']
- hl7:value[@xsi:type='ST']
- hl7:value[@xsi:type='CV']
- hl7:value[concat(@code, @codeSystem) = doc('include/voc-1.2.40.0.34.10.186-DYNAMIC.xml')//valueSet[1]/conceptList/concept/concat(@code, @codeSystem) or @nullFlavor]
- hl7:value[(@code = '281268007' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
- hl7:value[(@code = '255599008' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
- hl7:value[@xsi:type='CD']
- hl7:value[@xsi:type='RTO']
- hl7:value[@xsi:type='RTO_PQ_PQ']
| | PQ | 0 … 1 | | Codierung einer physikalischen Größe | | | wo [@xsi:type='PQ'] | | | | Beispiel | Codierung eines numerischen Ergebnisses <value xsi:type="PQ" value="49.7" unit="%"/> | | | Beispiel | Codierung von numerischen, dimensionslosen Ergebnissen <!-- Für dimensionslose Einheiten wird in UCUM häufig eine Einheit angegeben, wie z.B. "[pH]" für den pH-Wert. -->
<value xsi:type="PQ" value="7" unit="[pH]"/> | | | Beispiel | Codierung von numerischen, dimensionslosen Ergebnissen <!-- Wenn keine UCUM-Einheit vorgeschlagen ist, können dimensionslose Einheiten auch mit @unit="1" dargestellt werden, hier für INR. -->
<value xsi:type="PQ" value="1.1" unit="1"/> | | PQR | 0 … 1 | | Alternative Repräsentation derselben physikalischen Größe, mit unterschiedlicher Einheit in @code und @codeSystem und einem möglicherweise unterschiedlichen Wert. | | | IVL_PQ | 0 … 1 | | Codierung eines Intervalls von physikalischen Größen | | | wo [@xsi:type='IVL_PQ'] | | | | Beispiel | Codierung eines Intervalls <!-- Angabe von 20 - 30 mg/dl -->
<!-- @inclusive gibt mit true/false an, ob die Intervallgrenze im Intervall enthalten ist oder nicht (offenes oder geschlossenes Intervall). -->
<value xsi:type="IVL_PQ">
<low value="20" unit="mg/dl" inclusive="true"/> <high value="30" unit="mg/dl" inclusive="true"/></value> | | INT | 0 … 1 | | Codierung eines numerischen Ergebnisses. | | | wo [@xsi:type='INT'] | | | IVL_INT | 0 … 1 | | Codierung eines numerischen Intervalls. | | | wo [@xsi:type='IVL_INT'] | | | BL | 0 … 1 | | Codierung eines bool'schen Ergebnisses. | | | wo [@xsi:type='BL'] | | | ST | 0 … 1 | | Codierung eines textuellen Ergebnisses. Die Angabe des Ergebnisses erfolgt hier als Wert des Elements.
Im narrativen Block MUSS derselbe Text wie im Entry dargestellt werden. | | | wo [@xsi:type='ST'] | | | | Beispiel | Codierung eines textuellen Ergebnisses <value xsi:type="ST">strohgelb</value> | | CV | 0 … 1 | | Codierung des Ergebnisses in Form eines Codes. | | | wo [@xsi:type='CV'] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | CD | 0 … 1 | | Wenn ein Ergebnis vorliegt, das durch das Value Set "ELGA_NachweisErgebnis_VS" abgedeckt werden kann, MUSS ein Wert aus diesem verwendet werden.
Das Value Set definiert auch, wie die Darstellung in CDA Level 2 als Semigrafik erfolgen kann (siehe Beispiel). | | | cs | 1 … 1 | F | CD | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.186 ELGA_NachweisErgebnis_VS (DYNAMIC) |
| | | Beispiel | Codierung, wenn ein Erreger nachgewiesen werden konnte. <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<td>nachgewiesen</td><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Detected (qualifier value)"/> | | | Beispiel | Codierung und Darstellung mittels Semigrafik <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<td>+++</td><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<value xsi:type="CD" code="260354000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Moderate number (qualifier value)"/> | | CD | 0 … 1 | | Codierung, dass nicht ausreichend Material für die Durchführung der Analyse vorgelegen ist. | | | cs | 1 … 1 | F | CD | | st | 0 … 1 | F | SNOMED CT | | CONF | 0 … 1 | F | 281268007 | | 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) | | 0 … 1 | F | Insufficient sample (finding) | | | Beispiel | Zu wenig Material <value xsi:type="CD" code="281268007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Insufficient sample (finding)"/> | | CD | 0 … 1 | | Codierung, dass das Ergebnis der Analyse noch ausstehend bzw. noch in Arbeit ist. | | | cs | 1 … 1 | F | CD | | st | 0 … 1 | F | SNOMED CT | | CONF | 0 … 1 | F | 255599008 | | 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) | | 0 … 1 | F | Incomplete (qualifier value) | | | Beispiel | Ausstehendes Ergebnis ('Wert folgt') <value xsi:type="CD" code="255599008" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Incomplete (qualifier value)"/> | | CD | 0 … 1 | | Codierung des Ergebnisses in Form eines Codes. | | | wo [@xsi:type='CD'] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | RTO | 0 … 1 | | Codierung einer Verhältnisangabe (z.B. Titer). | | | wo [@xsi:type='RTO'] | | | | Beispiel | Verhältnisangabe für Titer (z.B. '1:125') <value xsi:type="RTO">
<numerator value="1" xsi:type="INT"/> <denominator value="128" xsi:type="INT"/></value> | | RTO_PQ_PQ | 0 … 1 | | Codierung einer Verhältnisangabe von physikalischen Größen. | | | wo [@xsi:type='RTO_PQ_PQ'] | | | | Schematron assert | role | error | | | | test | not(hl7:value[@nullFlavor]) or (hl7:value[@xsi:type='PQ'][@nullFlavor='UNK'] and (hl7:interpretationCode[not(@nullFlavor)] or hl7:interpretationCode[@nullFlavor='OTH']) and ../../../../hl7:code[@code='365705006'][@codeSystem='2.16.840.1.113883.6.96']) | | | | Meldung | Für Antibiogrammergebnisse kann value[@nullFlavor='UNK'] verwendet werden, wenn
interpretationCode oder interpretationCode[@nullFlavor='OTH'] vorhanden ist. Ansonsten ist die Verwendung von value[@nullFlavor] NICHT ERLAUBT. | | | | Schematron assert | role | error | | | | test | not(hl7:value[@code='255599008'][@codeSystem='2.16.840.1.113883.6.96']) | | | | Meldung | Ergebnisse früherer Analysen DÜRFEN NICHT als "in Arbeit" (SCT "255599008 - Incomplete (qualifier value)") markiert sein. | | | Auswahl | 0 … 1 | | Elemente in der Auswahl:- hl7:interpretationCode[not(@nullFlavor)]
- hl7:interpretationCode[@nullFlavor='OTH']
| | CE | 0 … 1 | | Codierte Bewertung des Ergebnisses. Wird für Referenzbereichbewertungen, für die Codierung der RAST-Klassen sowie für die Codierung von Antibiogrammergebnissen verwendet.
| | | wo [not(@nullFlavor)] | | | cs | 1 … 1 | R | | | oid | 1 … 1 | R | | | st | 0 … 1 | | | | st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.13 ELGA_ObservationInterpretation (DYNAMIC) |
| | CE | 0 … 1 | | Codierte Bewertung des Ergebnisses mit einem Code, der aktuell nicht im Value Set "ELGA_ObservationInterpretation" enthalten ist.
| | | wo [@nullFlavor='OTH'] | | | | Schematron assert | role | error | | | | test | hl7:translation[not(@nullFlavor)] | | | | Meldung | Wenn interpretationCode[@nullFlavor='OTH'] dann MUSS "interpretationCode/translation" anwesend sein. | |  | hl7:referenceRange
|
| | 0 … 1 | | Codierung des Referenzbereiches.
| |  |  | @typeCode
|
| cs | 1 … 1 | F | REFV | | | Beispiel | 43.0% - 49.0% (im zugehörigen section/text steht der entsprechende Text) <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-1">
<td>--Analyse--</td> <td>--Ergebnis--</td> <td>--Einheit--</td> <td ID="OBSREF-1-1">43.0% - 49.0%</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-1"/> </text> <value type="IVL_PQ">
<low value="43.0" unit="%"/> <high value="49.0" unit="%"/> </value> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Normal"/> </observationRange></referenceRange> | | | Beispiel | Phase 1: 43.0 - 49.0 Phase 2: 45.0 – 55.0 Phase 3: 49.0 – 63.0 (im zugehörigen section/text steht der entsprechende Text) <!-- Da oftmals die Kriterien für die Bewertung von Laborergebnissen nicht vollständig vorliegen, muss für einen Laborbefund die Angabe mehrerer möglicher Referenzbereiche möglich sein, welche sich durch unterschiedliche Vorbedingungen (preconditions) unterscheiden. Leider ist die Angabe solcher unter dem referenceRange laut CDA Rel.2 Definition nicht möglich. Deshalb MUSS an dieser Stelle eine Angabe in Textform erfolgen. Nachfolgendes Beispiel zeigt die Verwendung der "referenceRange" mit mehreren Referenzbereichen mit Preconditions. -->
<!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-2">
<td>--Analyse--</td> <td>--Ergebnis--</td> <td>--Einheit--</td> <td ID="OBSREF-1-2">
Phase 1: 43.0 - 49.0 <br/> Phase 2: 45.0 – 55.0 <br/> Phase 3: 49.0 – 63.0 </td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-2"/> </text> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Normal"/> </observationRange></referenceRange> | | | Beispiel | >40.0% (im zugehörigen section/text steht der entsprechende Text) <!-- Im Falle eines einseitig unbeschränkten Intervalls wie z.B. bei ">40" wird entweder nur der "low" bzw. "high" Wert angegeben, wobei auf der Seite, auf der die Intervallgrenze fehlt, ein @nullFlavor angegeben werden MUSS. Ein "kleiner als" Referenzbereich wie z.B. "<17" SOLL als Intervall von 0 bis 17 beschrieben werden. -->
<!-- ... -->
<!-- WICHTIG: Im text-Bereich MUSS ">" durch "& gt;" und "<" durch "& lt;" ersetzt werden (jeweils ohne Leerzeichen zwischen "&" und "g" bzw. "l"). -->
<!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-3">
<td>--Analyse--</td> <td>--Ergebnis--</td> <td>--Einheit--</td> <td ID="OBSREF-1-3">>40.0%</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-3"/> </text> <value type="IVL_PQ">
<low value="40.0" unit="%" inclusive="false"/> <high nullFlavor="PINF"/> </value> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Normal"/> </observationRange></referenceRange> | | | Beispiel | 150 - 360 G/L (im zugehörigen section/text steht der entsprechende Text) <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<tr ID="OBS-1-4">
<td>--Analyse--</td> <td>--Ergebnis--</td> <td>--Einheit--</td> <td ID="OBSREF-1-4">150 - 360 G/L</td></tr><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<referenceRange typeCode="REFV">
<observationRange classCode="OBS" moodCode="EVN.CRT">
<text>
<reference value="#OBSREF-1-4"/> </text> <value type="IVL_PQ">
<low value="150" unit="G/L"/> <high value="360" unit="G/L"/> </value> <interpretationCode code="N" codeSystem="2.16.840.1.113883.5.83" codeSystemName="HL7:ObservationInterpretation" displayName="Normal"/> </observationRange></referenceRange> |  |  | hl7:observationRange
|
| | 1 … 1 | M | | | | cs | 1 … 1 | F | OBS | | cs | 1 … 1 | F | EVN.CRT | | Eingefügt | 1 … 1 | M | von 1.2.40.0.34.6.0.11.9.1 Narrative Text Reference (DYNAMIC)
Das Element "text" wird verwendet, um einen Verweis zum narrativen Text des Referenzbereiches herzustellen. | | ED | 1 … 1 | M | | | | TEL | 1 … 1 | M | Die Referenz auf den entsprechenden Text im menschenlesbaren Teil muss durch Bezugnahme auf den Inhalt[@ID] angegeben werden: reference[@value='#xxx'].
Die Referenz ist mit einem ID-Attribut anzugeben, dieses Element DARF NUR den Textinhalt des codierten Inhalts mit Zusatzinformationen umschließen.
Alternativ kann @value auch mit dem url-scheme "http" oder "https" beginnen.
| | | | 1 … 1 | R | | | | Schematron assert | role | error | | | | test | starts-with(@value,'#') or starts-with(@value,'http') | | | | Meldung | The @value attribute content MUST conform to the format '#xxx', where xxx is the ID of the corresponding 'content'-element, or begin with the 'http' or 'https' url-scheme. | | | IVL_PQ | 0 … 1 | | | | | Auswahl | 1 … 1 | | Unterer Grenzwert Elemente in der Auswahl:- hl7:low[not(@nullFlavor)]
- hl7:low[@nullFlavor='NA']
- hl7:low[@nullFlavor='NINF']
| | IVXB_PQ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | IVXB_PQ | 0 … 1 | | | | | wo [@nullFlavor='NA'] | | | IVXB_PQ | 0 … 1 | | | | | wo [@nullFlavor='NINF'] | | | Auswahl | 1 … 1 | | Oberer Grenzwert Elemente in der Auswahl:- hl7:high[not(@nullFlavor)]
- hl7:high[@nullFlavor='NA']
- hl7:high[@nullFlavor='PINF']
| | IVXB_PQ | 0 … 1 | | | | | wo [not(@nullFlavor)] | | | IVXB_PQ | 0 … 1 | | | | | wo [@nullFlavor='NA'] | | | IVXB_PQ | 0 … 1 | | | | | wo [@nullFlavor='PINF'] | | | CE | 1 … 1 | M | | | | st | 0 … 1 | F | HL7:ObservationInterpretation | | CONF | 1 … 1 | F | N | | 1 … 1 | F | 2.16.840.1.113883.5.83 (Observation Interpretation) | | 1 … 1 | F | Normal |
|
14.4.5.17 Logo Entry
14.4.5.18 Certainty Observation
14.4.5.19 Severity Observation
14.4.5.20 Problem Status Observation
14.4.5.21 Laterality Qualifier
14.4.5.23 External Document Entry
14.4.5.24 Eingebettetes Objekt Entry
14.4.6 Weitere CDA Fragmente
14.4.6.1 Laboratory Observation Value
| Id | 1.2.40.0.34.6.0.11.9.23 ref at-cda-bbr- | Gültigkeit ab | 2021‑01‑19 14:53:23 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atcdabbr_other_LaboratoryObservationValue | Bezeichnung | Laboratory Observation Value |
|---|
| Beschreibung | Codiertes Ergebnis im entsprechenden Datentyp. |
|---|
| Klassifikation | Template-Typ nicht spezifiziert |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Item | DT | Kard | Konf | Beschreibung | Label |
|---|
| Auswahl | | | Codiertes Ergebnis im entsprechenden Datentyp.
Numerische Ergebnisse werden in der Regel als "physical quantity" PQ dargestellt, was die Angabe einer UCUM codierten Einheit erforderlich macht. Es MUSS die "case sensitive" Variante (c/s) der maschinenlesbaren Form des UCUM verwendet werden. Die bevorzugte Einheit für jede Analyse wird im Value Set "ELGA_Laborparameter" vorgeschlagen, jeweils in der "print" Variante (für die Darstellung) und in der maschinenlesbaren Form.
Es wird EMPFOHLEN, anstelle von Einheitenpräfixen ("Giga", "Mega", "Milli", "Mikro" etc.) eine Potenzschreibweise zu wählen, vor allem, wenn die Groß/Kleinschreibung eine Rolle spielt und Verwechslungen möglich sind (z.B. "G/L"=Giga pro Liter vs. "g/L"=Gramm/Liter). Also "10^6" statt "M" (Mega), "10^9" statt "G" (Giga) usw.
Unterelemente können je nach Datentyp notwendig sein, z.B. high/low für IVL oder numerator/denominator für RTO.
Elemente in der Auswahl:- hl7:value[@xsi:type='PQ']
- hl7:value[@xsi:type='IVL_PQ']
- hl7:value[@xsi:type='INT']
- hl7:value[@xsi:type='IVL_INT']
- hl7:value[@xsi:type='BL']
- hl7:value[@xsi:type='ST']
- hl7:value[@xsi:type='CV']
- hl7:value[concat(@code, @codeSystem) = doc('include/voc-1.2.40.0.34.10.186-DYNAMIC.xml')//valueSet[1]/conceptList/concept/concat(@code, @codeSystem) or @nullFlavor]
- hl7:value[(@code = '281268007' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
- hl7:value[(@code = '255599008' and @codeSystem = '2.16.840.1.113883.6.96') or @nullFlavor]
- hl7:value[@xsi:type='CD']
- hl7:value[@xsi:type='RTO']
- hl7:value[@xsi:type='RTO_PQ_PQ']
|  | hl7:value
|
| PQ | 0 … 1 | | Codierung einer physikalischen Größe | | | wo [@xsi:type='PQ'] | | | | Beispiel | Codierung eines numerischen Ergebnisses <value xsi:type="PQ" value="49.7" unit="%"/> | | | Beispiel | Codierung von numerischen, dimensionslosen Ergebnissen <!-- Für dimensionslose Einheiten wird in UCUM häufig eine Einheit angegeben, wie z.B. "[pH]" für den pH-Wert. -->
<value xsi:type="PQ" value="7" unit="[pH]"/> | | | Beispiel | Codierung von numerischen, dimensionslosen Ergebnissen <!-- Wenn keine UCUM-Einheit vorgeschlagen ist, können dimensionslose Einheiten auch mit @unit="1" dargestellt werden, hier für INR. -->
<value xsi:type="PQ" value="1.1" unit="1"/> |  |  | hl7:translation
|
| PQR | 0 … 1 | | Alternative Repräsentation derselben physikalischen Größe, mit unterschiedlicher Einheit in @code und @codeSystem und einem möglicherweise unterschiedlichen Wert. | |  | hl7:value
|
| IVL_PQ | 0 … 1 | | Codierung eines Intervalls von physikalischen Größen | | | wo [@xsi:type='IVL_PQ'] | | | | Beispiel | Codierung eines Intervalls <!-- Angabe von 20 - 30 mg/dl -->
<!-- @inclusive gibt mit true/false an, ob die Intervallgrenze im Intervall enthalten ist oder nicht (offenes oder geschlossenes Intervall). -->
<value xsi:type="IVL_PQ">
<low value="20" unit="mg/dl" inclusive="true"/> <high value="30" unit="mg/dl" inclusive="true"/></value> |  | hl7:value
|
| INT | 0 … 1 | | Codierung eines numerischen Ergebnisses. | | | wo [@xsi:type='INT'] | |  | hl7:value
|
| IVL_INT | 0 … 1 | | Codierung eines numerischen Intervalls. | | | wo [@xsi:type='IVL_INT'] | |  | hl7:value
|
| BL | 0 … 1 | | Codierung eines bool'schen Ergebnisses. | | | wo [@xsi:type='BL'] | |  | hl7:value
|
| ST | 0 … 1 | | Codierung eines textuellen Ergebnisses. Die Angabe des Ergebnisses erfolgt hier als Wert des Elements.
Im narrativen Block MUSS derselbe Text wie im Entry dargestellt werden. | | | wo [@xsi:type='ST'] | | | | Beispiel | Codierung eines textuellen Ergebnisses <value xsi:type="ST">strohgelb</value> |  | hl7:value
|
| CV | 0 … 1 | | Codierung des Ergebnisses in Form eines Codes. | | | wo [@xsi:type='CV'] | |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | |  | hl7:value
|
| CD | 0 … 1 | | Wenn ein Ergebnis vorliegt, das durch das Value Set "ELGA_NachweisErgebnis_VS" abgedeckt werden kann, MUSS ein Wert aus diesem verwendet werden.
Das Value Set definiert auch, wie die Darstellung in CDA Level 2 als Semigrafik erfolgen kann (siehe Beispiel). | |  |  | @xsi:type
|
| cs | 1 … 1 | F | CD |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | | | | CONF | | Der Wert von @code muss gewählt werden aus dem Value Set 1.2.40.0.34.10.186 ELGA_NachweisErgebnis_VS (DYNAMIC) |
| | | Beispiel | Codierung, wenn ein Erreger nachgewiesen werden konnte. <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<td>nachgewiesen</td><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<value xsi:type="CD" code="260373001" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Detected (qualifier value)"/> | | | Beispiel | Codierung und Darstellung mittels Semigrafik <!-- ... -->
<!-- CDA Level 2 -->
<!-- ... -->
<td>+++</td><!-- ... -->
<!-- CDA Level 3 -->
<!-- ... -->
<value xsi:type="CD" code="260354000" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Moderate number (qualifier value)"/> |  | hl7:value
|
| CD | 0 … 1 | | Codierung, dass nicht ausreichend Material für die Durchführung der Analyse vorgelegen ist. | |  |  | @xsi:type
|
| cs | 1 … 1 | F | CD |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 0 … 1 | F | 281268007 |  |  | @codeSystem
|
| 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 0 … 1 | F | Insufficient sample (finding) | | | Beispiel | Zu wenig Material <value xsi:type="CD" code="281268007" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Insufficient sample (finding)"/> |  | hl7:value
|
| CD | 0 … 1 | | Codierung, dass das Ergebnis der Analyse noch ausstehend bzw. noch in Arbeit ist. | |  |  | @xsi:type
|
| cs | 1 … 1 | F | CD |  |  | @codeSystemName
|
| st | 0 … 1 | F | SNOMED CT |  |  | @code
|
| CONF | 0 … 1 | F | 255599008 |  |  | @codeSystem
|
| 0 … 1 | F | 2.16.840.1.113883.6.96 (SNOMED Clinical Terms) |  |  | @displayName
|
| 0 … 1 | F | Incomplete (qualifier value) | | | Beispiel | Ausstehendes Ergebnis ('Wert folgt') <value xsi:type="CD" code="255599008" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED CT" displayName="Incomplete (qualifier value)"/> |  | hl7:value
|
| CD | 0 … 1 | | Codierung des Ergebnisses in Form eines Codes. | | | wo [@xsi:type='CD'] | |  |  | @code
|
| cs | 1 … 1 | R | |  |  | @codeSystem
|
| oid | 1 … 1 | R | |  |  | @codeSystemName
|
| st | 0 … 1 | | |  |  | @displayName
|
| st | 1 … 1 | R | |  | hl7:value
|
| RTO | 0 … 1 | | Codierung einer Verhältnisangabe (z.B. Titer). | | | wo [@xsi:type='RTO'] | | | | Beispiel | Verhältnisangabe für Titer (z.B. '1:125') <value xsi:type="RTO">
<numerator value="1" xsi:type="INT"/> <denominator value="128" xsi:type="INT"/></value> |  | hl7:value
|
| RTO_PQ_PQ | 0 … 1 | | Codierung einer Verhältnisangabe von physikalischen Größen. | | | wo [@xsi:type='RTO_PQ_PQ'] | |
|
14.4.6.2 Performer - Laboratory
| Id | 1.2.40.0.34.6.0.11.9.24 ref at-cda-bbr- | Gültigkeit ab | 2021‑04‑08 11:49:48 |
|---|
| Status |  Aktiv Aktiv | Versions-Label | 1.0.0+20211213 |
|---|
| Name | atcdabbr_other_PerformerLaboratory | Bezeichnung | Performer - Laboratory |
|---|
| Beschreibung | Erbringer der Gesundheitsdienstleistung (Labor mit seinem Leiter).
Wurde der Befund nur von einem Labor erstellt, MUSS dieses in "/ClinicalDocument/documentationOf/serviceEvent/performer" dokumentiert werden.
Sind mehrere Labors an der Erstellung beteiligt, MUSS das Labor im "structuredBody" entweder auf "entry"-Ebene oder im Rahmen eines "organizer"-Elementes oder direkt bei der Einzeluntersuchung ("observation"-Element) angegeben werden. Angaben in tieferen Ebenen (z.B. "observation"-Ebene) überschreiben solche auf höheren Ebenen (z.B. "organizer"-Ebene). |
|
| Klassifikation | Template-Typ nicht spezifiziert |
|---|
| Offen/Geschlossen | Geschlossen (nur definierte Elemente sind erlaubt) |
|---|
| Benutzt | | Benutzt 1 Template | | Benutzt | als | Name | Version |
|---|
| 1.2.40.0.34.6.0.11.9.41 | Containment |  Assigned Entity with id, name, addr and telecom (1.0.1+20211213) Assigned Entity with id, name, addr and telecom (1.0.1+20211213) | DYNAMIC |
|
|
|---|
| Beispiel | | Beispiel | <performer typeCode="PRF">
<templateId root="1.2.40.0.34.6.0.11.9.24"/> <templateId root="1.3.6.1.4.1.19376.1.3.3.1.7"/> <time value="20121201061325+0100"/> <assignedEntity>
<!-- template 1.2.40.0.34.6.0.11.9.41 'Assigned Entity with id, name, addr and telecom' -->
</assignedEntity></performer> |
|
|---|
|
14.4.6.3 Address Compilation
14.4.6.4 Address Compilation Minimal
14.4.6.5 Assigned Entity
14.4.6.6 Assigned Entity Body
14.4.6.7 Assigned Entity Body with name, addr and telecom
14.4.6.8 Assigned Entity with id, name, addr and telecom
14.4.6.9 Author Body
14.4.6.10 Device Compilation
14.4.6.11 Informant Body
14.4.6.12 Narrative Text Reference
14.4.6.13 Organization Compilation with id, name
14.4.6.14 Organization Compilation with name
14.4.6.15 Organization Compilation with name, addr minimal
14.4.6.16 Organization Compilation with name, addr minimal and telecom
14.4.6.17 Organization Name Compilation
14.4.6.18 Original Text Reference
14.4.6.19 Participant Body
14.4.6.20 Performer Body
14.4.6.21 Person Name Compilation G1 M
14.4.6.22 Person Name Compilation G2
14.4.6.23 Person Name Compilation G2 M
14.4.6.24 Time Interval Information minimal
14.5 Terminologien
Die erforderlichen Terminologien sind im Folgenden aufgelistet.
Falls Werte in den definierten Value Sets fehlen, dann bitten wir um die Meldung mit einem Vorschlag an
cda@elga.gv.at.
15 Anhang
15.1 Abbildungsverzeichnis
- ↑ Gliederung nach Befundbereichen (Laboratory Specialty Sections)
- ↑ Strukturierungsmöglichkeiten im Laborbefund im Body
- ↑ Ausschnitt aus einem Laborbefund
- ↑ Ausschnitt aus einem Mikrobiologiebefund
- ↑ Angabe des Akkreditierungs-Logos im Brieftext
15.2 Tabellenverzeichnis
- ↑ Übersichtstabelle über die Dokument-Metadaten (XDS-Metadaten)
- ↑ Im Labor- und Mikrobiologiebefund abzubildende medizinische Daten
- ↑ Liste der Befundbereiche, auszugsweise gemäß Value Set "ELGA_Laborstruktur", die sich auch im Value Set "ELGA_Laborparameter" wiederfinden.
- ↑ Übersichtstabelle der CDA Strukturen des Headers
- ↑ Übersichtstabelle der Header-Elemente für Zeitpunkte/Zeitspannen
- ↑ Übersichtstabelle der CDA Strukturen des Bodys des Laborbefundes
- ↑ Übersichtstabelle der CDA Strukturen des Bodys des Mikrobiologiebefundes
- ↑ Übersichtstabelle der CDA Strukturen des Laboratory Report Data Processing Entries
15.3 Literatur und Weblinks
- ↑ Logical Observation Identifiers Names & Codes (LOINC) loinc.org
- ↑ 2,0 2,1 Regenstrief Institute, Inc. www.regenstrief.org
- ↑ Unified Code for Units of Measure (UCUM) www.unitsofmeasure.org
- ↑ WHO ICD-10 www.who.int/classifications/icd/en/
- ↑ 5,0 5,1 www.who.int
- ↑ Internationale statistische Klassifikation der Krankheiten und verwandter Gesundheitsprobleme 10. Revision – aktuelle Version bitte unter Gesundheitssystem - Krankenanstalten heraussuchen.
- ↑ Anatomical Therapeutic Chemical Classification System (ATC) https://www.who.int/tools/atc-ddd-toolkit/atc-classification
- ↑ ARGE Pharma im Fachverband der chemischen Industrie Österreichs (FCIO) argepharma.fcio.at
- ↑ EDQM Council of Europe www.edqm.eu
- ↑ Health informatics - Medical / health device communication standards ISO/IEEE 11073 Nomenclature Part 10101: Nomenclature
- ↑ Health informatics - Medical / health device communication standards ISO/IEEE 11073 Nomenclature Amendment 1 Part 10101: Nomenclature Amendment 1: Additional Definitions
- ↑ Health Level Seven International www.hl7.org
- ↑ ISO/HL7 27932:2009 Data Exchange Standards — HL7 Clinical Document Architecture, Release 2 [1]
- ↑ World Wide Web Consortium. Extensible Markup Language, 1.0, 5th Edition. [2]
- ↑ HL7 Version 3 Product Suite [3]
- ↑ ART-DECOR® www.art-decor.org
- ↑ HL7 Clinical Document Architecture (CDA) [4]
- ↑ 18,0 18,1 HL7 Version 3: Reference Information Model (RIM) [5]
- ↑ HL7 Version 3 Standard: Data Types – Abstract Specification, Release 2[6]
- ↑ HL7 Templates Standard: Specification and Use of Reusable Information Constraint Templates, Release 1 [7]
- ↑ HL7 Austria www.hl7.at
- ↑ 22,0 22,1 22,2 22,3 IHE Pathology and Laboratory Medicine (PALM) Technical Framework Volume 3 (PaLM TF-3) Revision 10.0, 2019 [8]
- ↑ Leitfaden zur Verwendung von LOINC® im ELGA CDA® R2 Laborbefund [9]
- ↑ ISO 15189:2012 "Medizinische Laboratorien - Anforderungen an die Qualität und Kompetenz" [10]
15.4 Release-Log, Ausblick und weitere Informationen
Auf der Diskussionsseite zu diesem Leitfaden können das Release-Log, das einen Überblick über die in diesem Leitfaden implementierten Neuerungen gibt, und der Ausblick auf Änderungen, die in zukünftigen Leitfadenversionen geplant sind, eingesehen werden. Gegebenenfalls werden Inhalte aktueller Diskussionen, bekannte Probleme oder weitere Hinweise aufgeführt.